转录调控高级分析结果详解(WGCNA\GSEA\PPI) |
您所在的位置:网站首页 › wgcna分析解读 › 转录调控高级分析结果详解(WGCNA\GSEA\PPI) |
转录调控高级分析结果详解(WGCNA\GSEA\PPI)
|
WGCNA简介
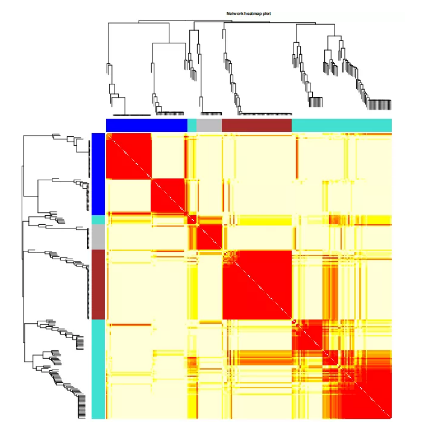
WGCNA(Weighted Gene Co-Expression Network Analysis, 加权基因共表达网络分析),是用来描述不同样品之间基因关联模式的系统生物学方法。可鉴定表达模式相似的基因集合(module),解析基因集合与样品表型之间的联系,绘制基因集合中基因之间的调控网络并鉴定关键调控基因。要了解 WGCNA, 首先需要理解什么是基因共表达网络。我们定义每个节点为一个基因,在不同样本中存在表达共性的基因处于同一个基因网络,而基因之间的共表达关系一般由它们之间的表达相关系数衡量。WGCNA 算法首先假定基因网络服从无尺度分布,并定义基因共表达相关矩阵、基因网络形成的邻接函数,然后计算不同节点的相异系数,并据此构建分层聚类树(hierarchical clustering tree),该聚类树的不同分支代表不同的基因模块(module),模块基因共表达程度高,而分属不同模块的基因共表达程度低。最后探索模块与特定表型或疾病的关联关系,最终达到鉴定基因网络的目的。 结果说明1. 基因共表达网络热图分析 WGCNA的核心步骤其实就是通过对TOM矩阵进行分层聚类,TOM (Topological overlap matrix)即为把邻接矩阵转换为拓扑重叠矩阵,以降低噪音和假相关,获得的新距离矩阵,可以用来计算基因之间关联程度。基于TOM矩阵,图A中的行和列均代表基因,灰色模块为无法分配到初级模块中的基因,树枝的尖端为核心基因,颜色越深代表TOM值越高。热图中方块的颜色越深(红)表示共表达相关性越高,越浅(黄)表示相关性越弱。
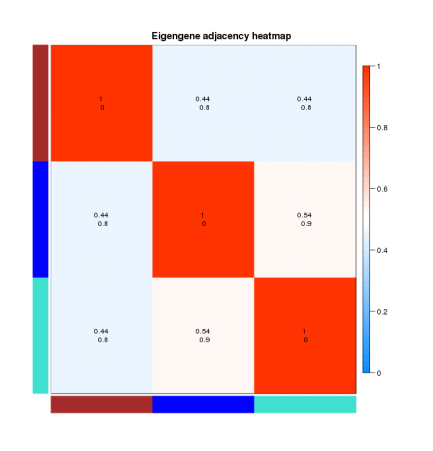
2. 模块间相关性热图 下图展示模块与模块间的相关性。横轴纵轴分别代表不同的模块,相关性越弱则呈现蓝色,相关性越强则呈现红色。
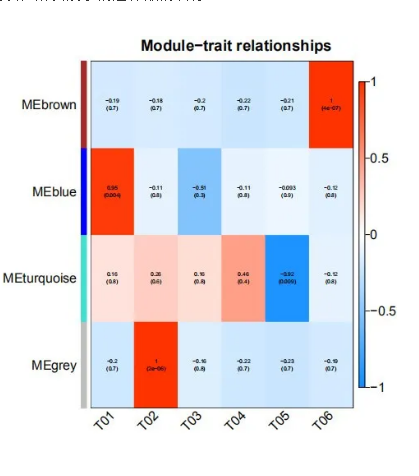
3. 模块与性状间的热图 模块与性状相关性热图:下图展示模块与给定性状的相关系热图。某一性状与某一模块的相关性越接近 1 的绝对值,很可能此性状与模块的基因功能相关。横轴代表着性状,纵轴代表着模块(下图中我们没有选择性状,所以展示的是样品的名称)。
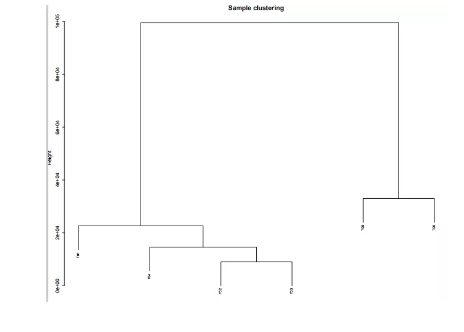
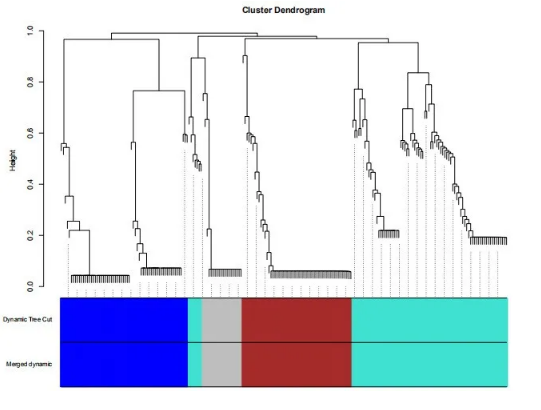
基于基因表达量的样本系统聚类树: 在聚类中欧氏距离求两点的距离,聚类中用类平均法(average)计算集合之间的距离。同一枝上的样本具有更高的相似性。
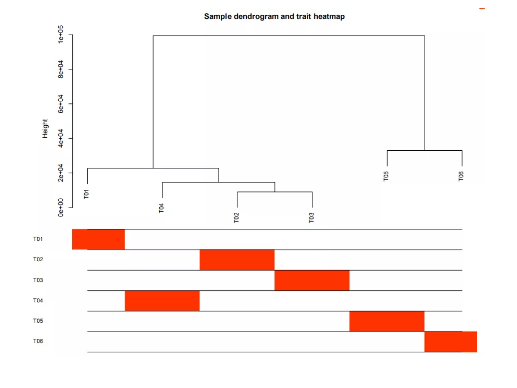
5.样本系统聚类树及相关性状热图 在基因表达量的样本系统聚类树的基础上我们添加了热图,样本与性状之间的相关性越高热图中模块颜色也会越深。例如我们将热图左边的T01看作是性状标签,它与分支样本TP01的热图模块呈现红色,说明性状T01与样本T01的相关性非常高。
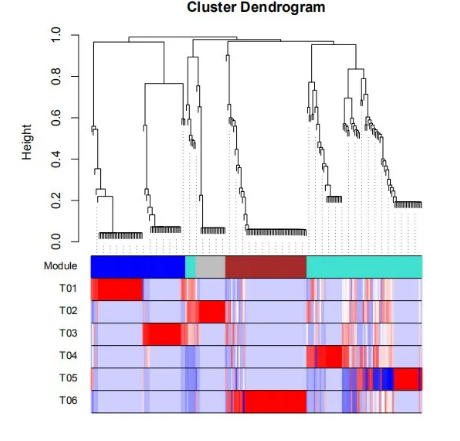
6.基因的系统树图及性状相关性热图 基因的系统树图及性状相关性热图主要分为三个部分:第一个部分为基因聚类绘制的聚类树,分枝代表基因;第二部分为基因聚类树根据不同的聚类对应到的不同模块;第三部分为性状的热图,如图中T01~T06为性状,对应热图中的颜色越深,说明该模块中的基因与该性状的相关性越高。
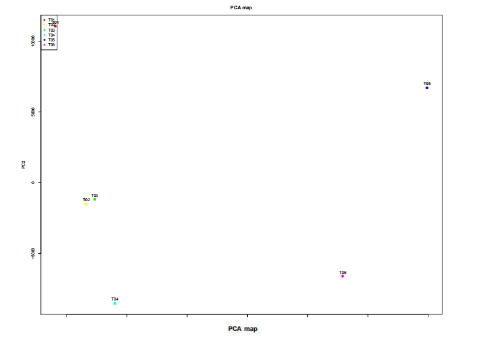
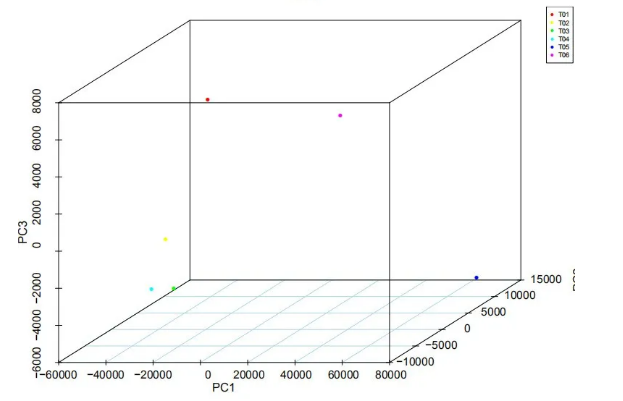
7.样品 PCA 分析二/三维图 PCA分析横坐标代表着第一主成分,纵坐标代表第二主成分,可以根据图看出在第一主成分方向上样本的距离越近,说明它们的主成分越相近,相关性越高。
8.基因网络模块 基因网络模块主要分为三个部分:第一部分为基因的聚类树;第二部分为根据基因聚类树划分的不同模块;第三部分为根据第二部分模块进行了矫正,最后结果为校正后的模块。
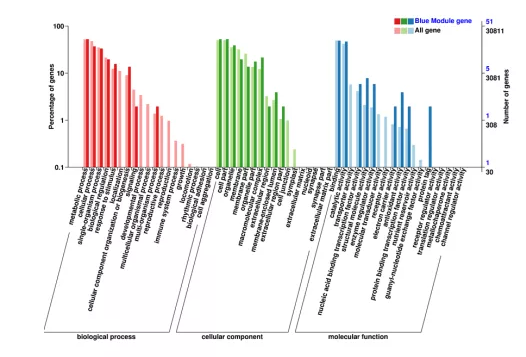
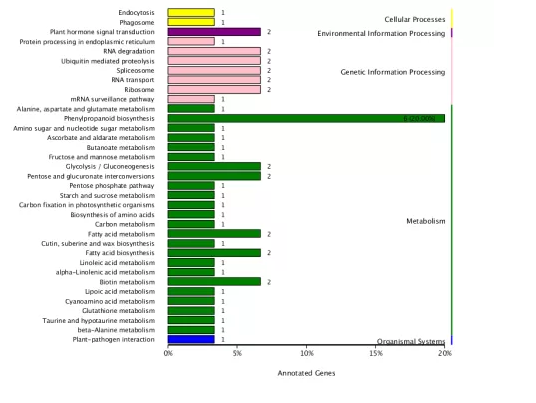
9.模块COG/KEGG/GO二级分类图 将每一个模块中的基因绘制了跟它功能相关的COG/KEGG/GO分类富集图。
在COG富集分析中横轴代表功能分类,纵轴代表基因频数。
在GO富集分析中横轴代表GO term,左侧纵轴代表基因占比,右侧为基因的数目。
在KEGG富集中纵轴代表路径,横轴代表注释到的基因数目
基因集富集分析(GSEA: Gene Set Enrichment Analysis),可以在没有先验经验存在的情况下根据所有基因表达情况对所有基因进行富集分析。一般的差异分析通常只集中关注于一些显著的上调或下调基因,而这会遗漏部分差异表达不显著却有重要生物学意义的基因。而GSEA不会设置差异阈值,能够检测出微弱但是一致的趋势。其输入数据包含两部分:一为已知功能的基因集 (可以是GO注释、MsigDB的注释或其它符合格式的基因集定义);二是表达矩阵 (也可以是排序好的列表)。 软件会对基因根据其与表型的关联度从大到小排序,然后判断基因集内每条注释下的基因是否富集于表型相关度排序后基因表的上部或下部,从而判断此基因集内基因的协同变化对表型变化的影响。 结果说明百迈客中的GSEA分析采用KEGG通路以及GO的BP、CC、MF分支的基因集作为感兴趣的基因集合,以每个差异分组的log2FC作为背景基因集的打分来分析感兴趣基因集合的富集情况,最后控制pvalue |
【本文地址】
 4.基因表达量的样本系统聚类树
4.基因表达量的样本系统聚类树