最全进化树构建知识点大盘点 |
您所在的位置:网站首页 › 进化树节点数值 › 最全进化树构建知识点大盘点 |
最全进化树构建知识点大盘点
|
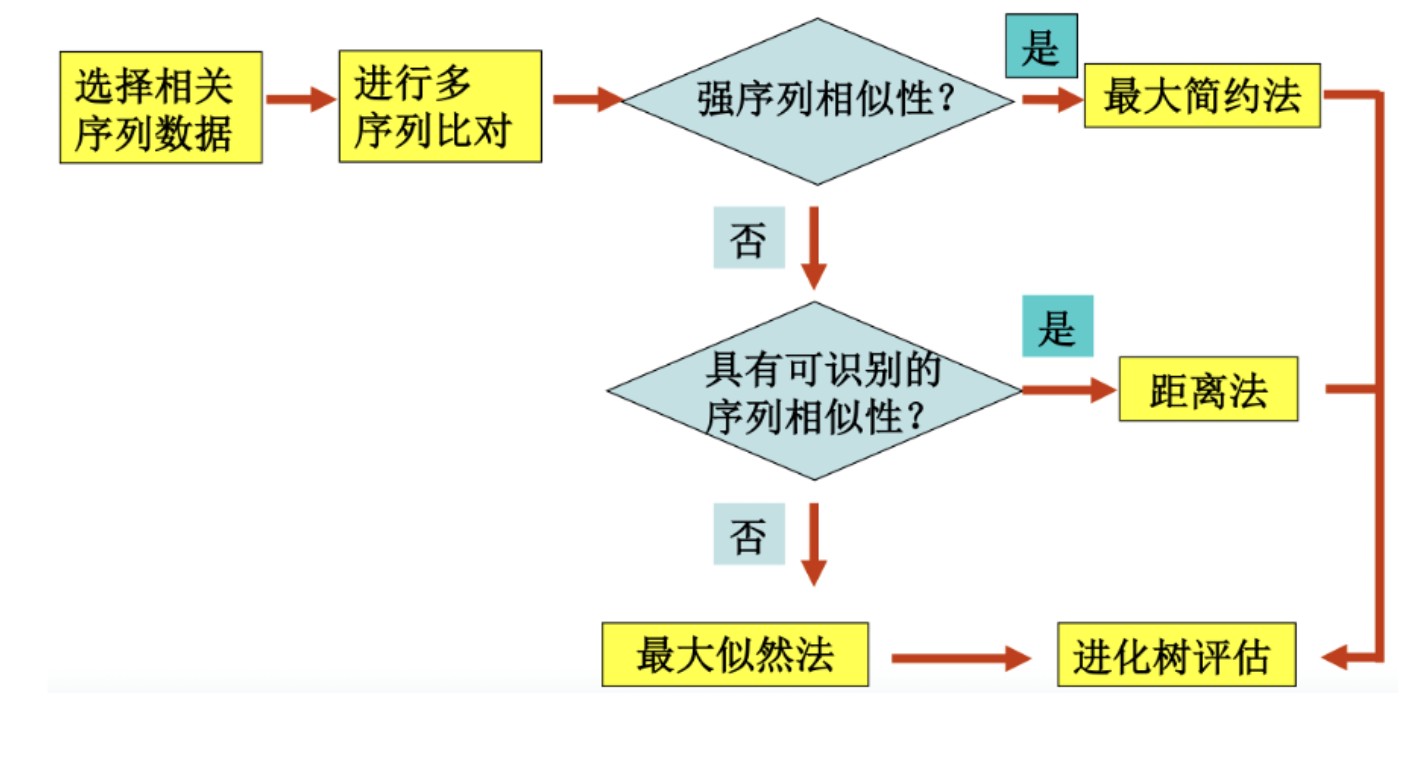
从计算速度来看,最快的是基于距离的方法,几十条序列几秒钟即可完成,其次是最大简约法,最大似然法就要慢得多,最慢的是贝叶斯法。但是从计算准确度来看,算得最慢的贝叶斯法确是最准确,而算得最快的基于距离法结果确是最粗糙。从实用的角度,建议使用最大似然法。因为这种方法无论从速度还是准确度都比较适中。 氨基酸/核酸替换模型的选择 p距离(p-distance ):两条蛋白质序列之间的氨基酸差异数为d, 序列的氨基酸数目均为n,则P距离:
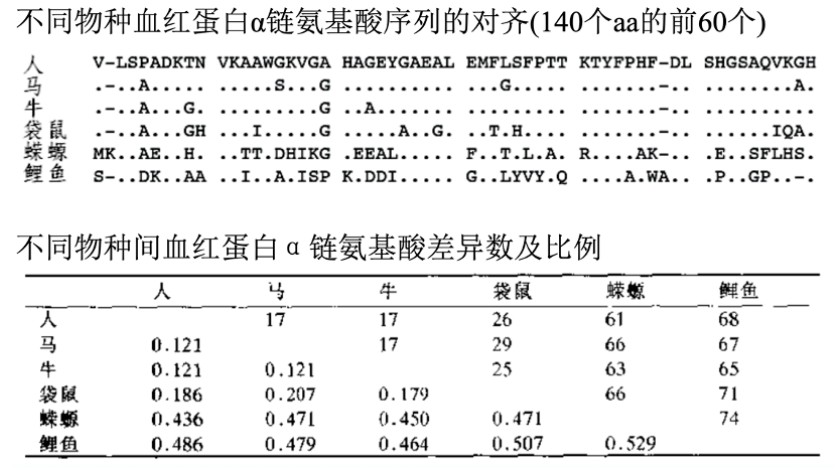
以下为不同物种血红蛋白相对人类的P距离计算得到距离矩阵,就可以用于进化树构建了;
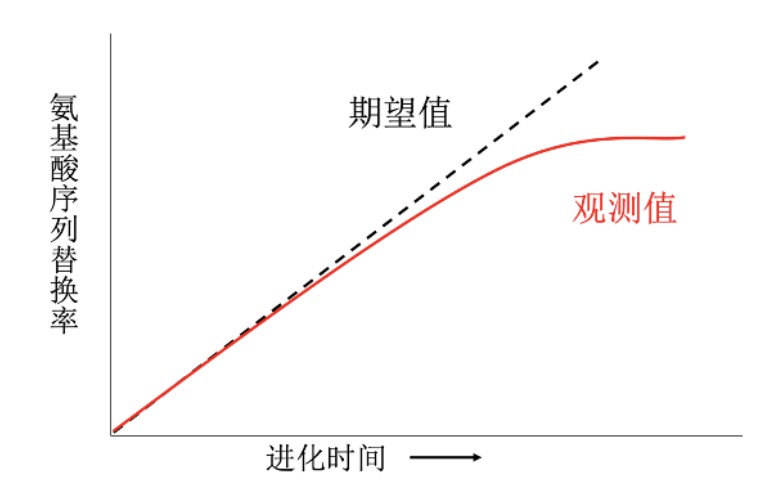
但是,p距离不是很严格地与进化时间成比例,进化时间较短时,两者大致成线性关系;当进化时间较长时,两者成非线性关系。
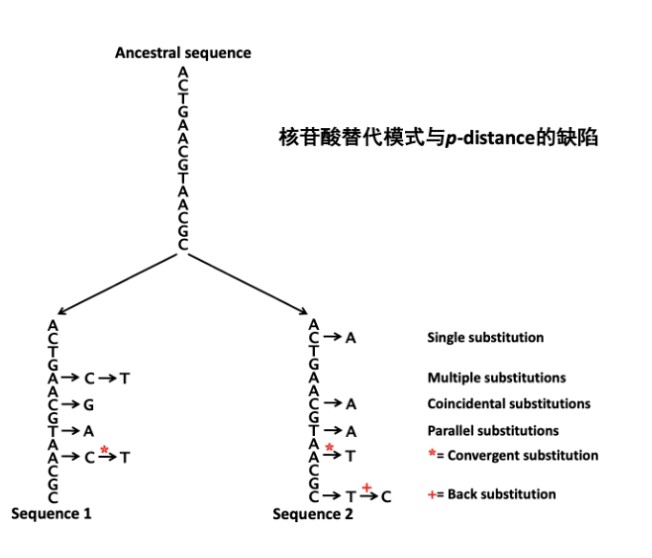
这是因为我们只能观察到现在的序列,祖先序列以及序列如何从祖先序列演化成现在序列的过程中发生了多少次替换我们并不知道。例如下图,祖先碱基C,通过回反替换C->T->C 又变成了C,我们观察到这个位点没有替换,但实际上已经发生了两次替换,这就是P距离的缺陷。
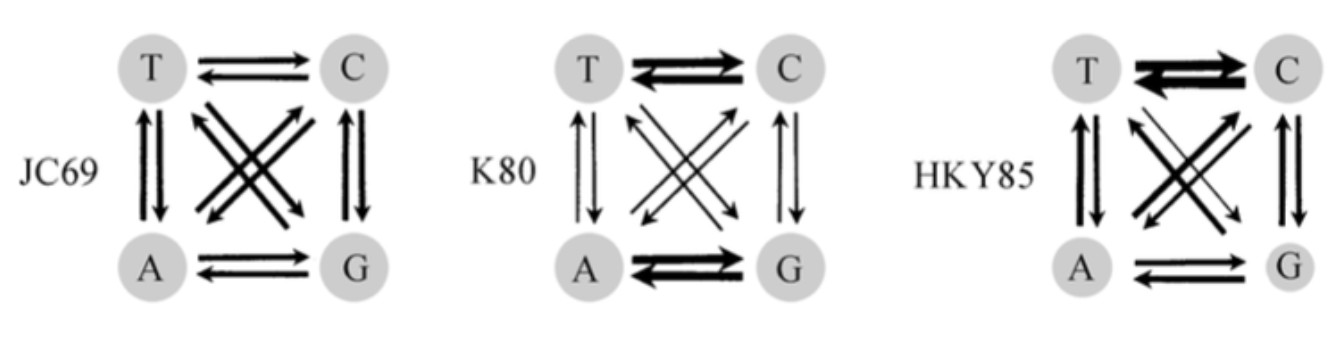
因此为解决p距离的问题,科学家们提出了很多替换模型,最简单的为JC69模型认为不同碱基之间的替换率相同(单参数模型);K80模型认为转换(A→G,G→A,C→T,T→C)和颠换(A→C,A→T,C→G,G→T)频率不同,认为转换频率比颠换频率高(双参数模型);HKY85更复杂, 又认为转换中的T→C的频率高于A→G的频率等。
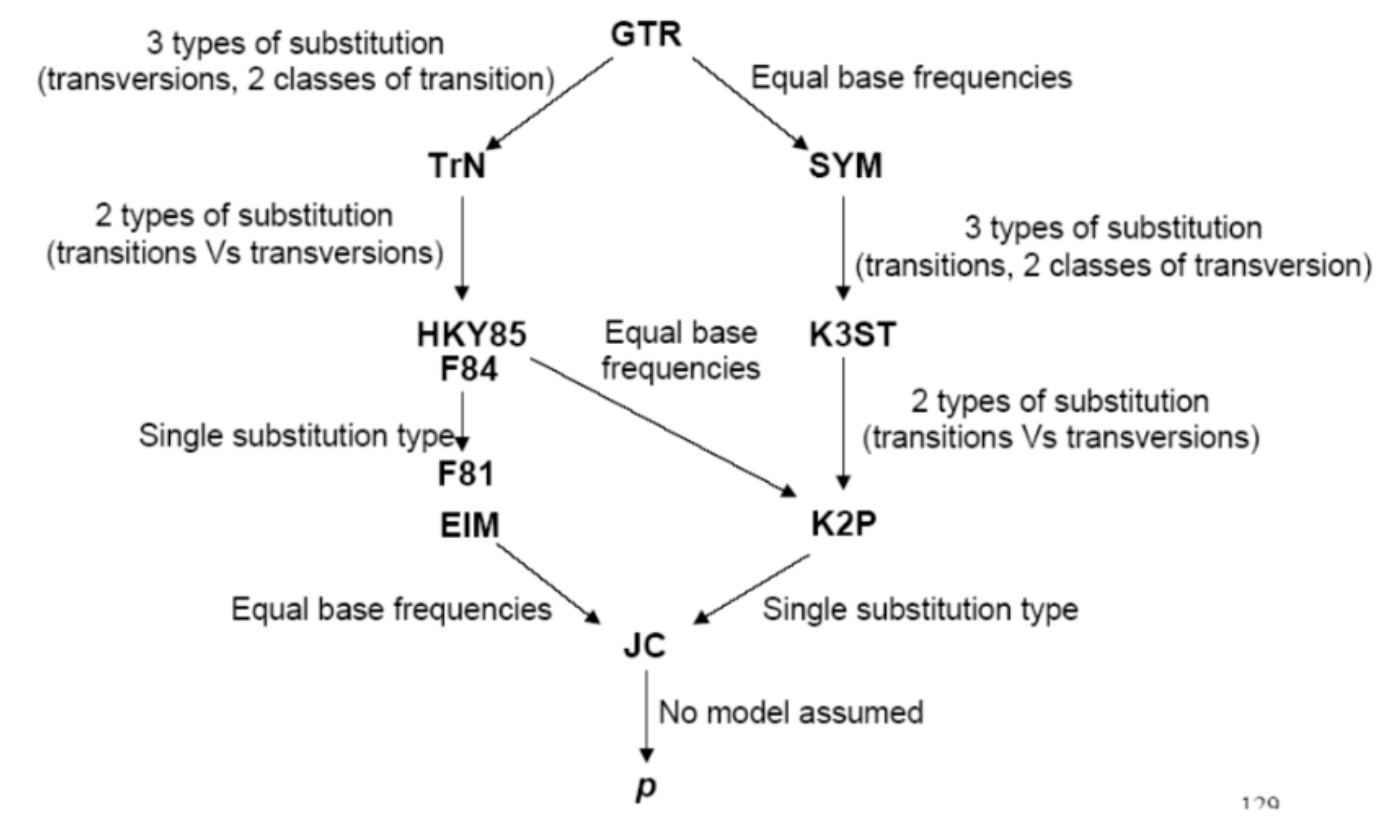
常用的核苷酸替换模型有JC69 、K80 、F81、TN93,HKY和GTR 模型等。其中GTR是所有模型中考虑参数最多最复杂的模型,其他所有模型是GTR模型的特例。上碱基的比例是不等的(两两之间),两两之间的替换速率也是不等的,所有这些参数都考虑进去了。
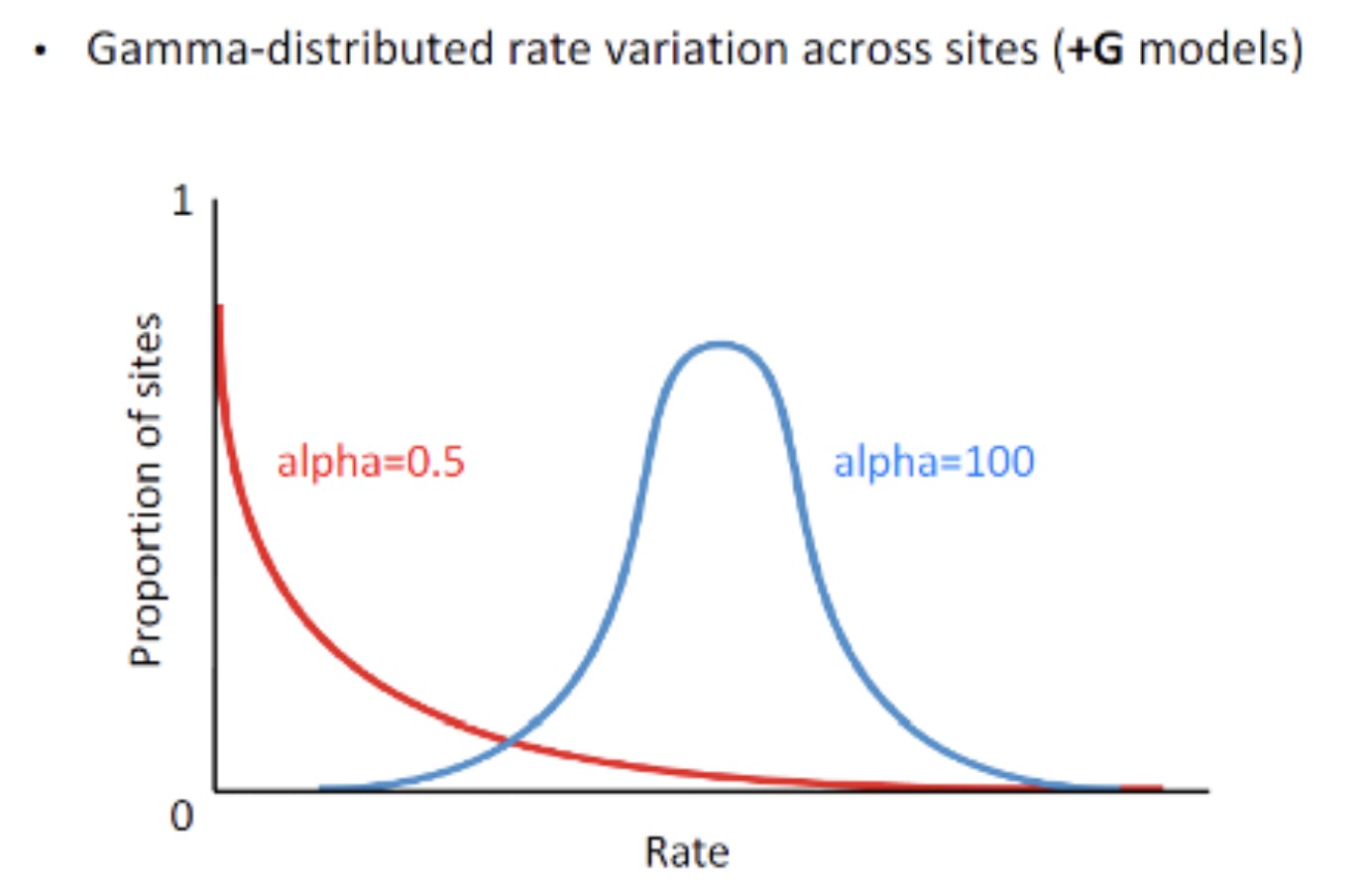
以上模型都是基于假设所有核苷酸位点的替代速率相同,假如:每个核苷酸位点上的替代是随机发生的,则A,T,C,G出现的频率应该大致相等。 实际情况:DNA受到自然选择的压力,各个位点的碱基出现频率并不相等。例如密码子的最后一位比前两位受到的限制少,变化速率更快;再例如编码蛋白关键结构域区域的序列比其他区域受到的自然选择压力更大,该区域替换的频率更低。为了在建树时,表示各个位点的进化速率不同引入gamma分布(gamma distribution (G): gamma distributed rate variation among sites),gamma分布表示碱基位点之间的进化速率异质性。
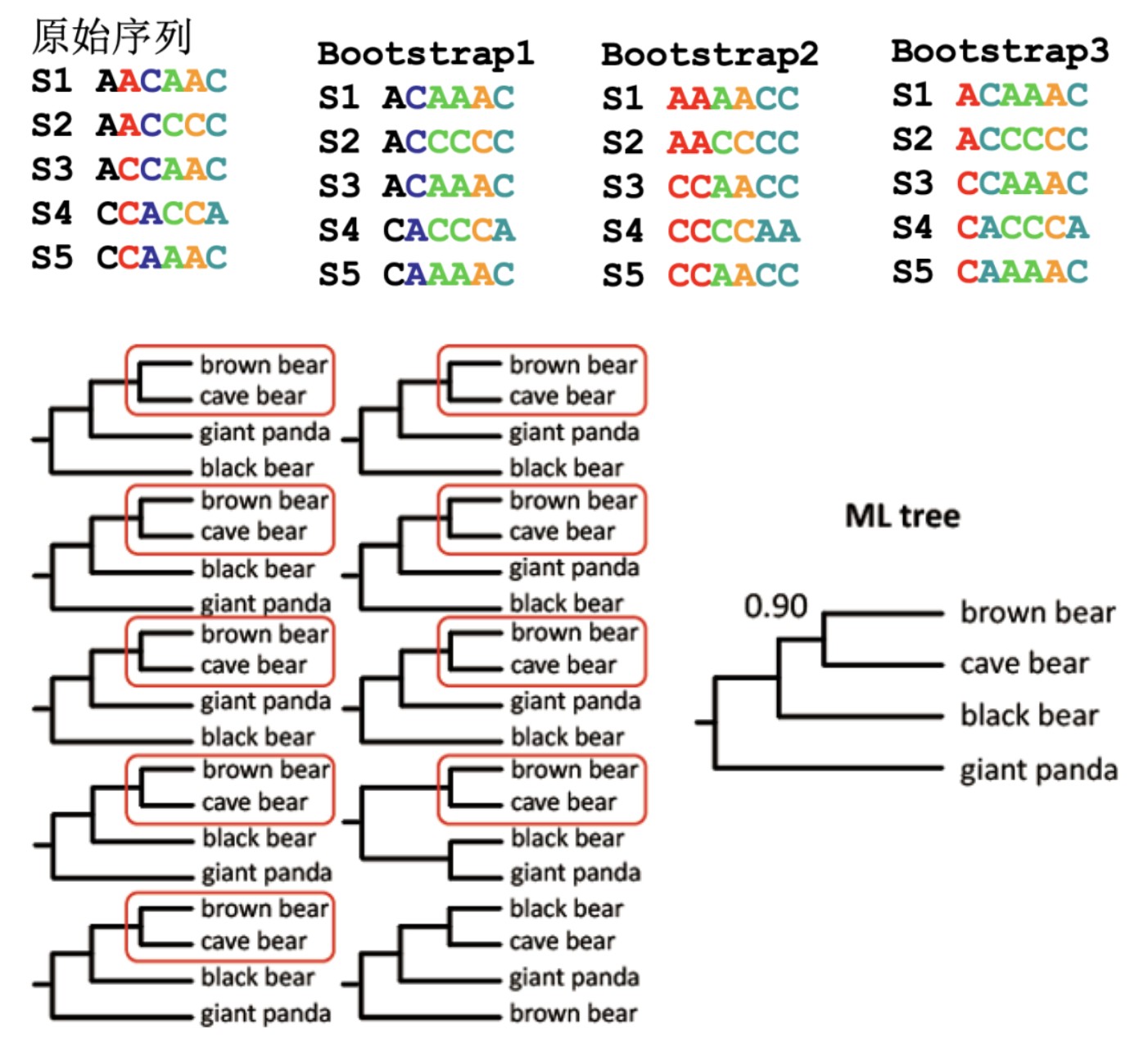
生物的进化历史是唯一的,我们并不能从这么多的模型中确定真实的核苷酸替代过程是依照哪种模型发生的。理论上应该尝试各种模型,根据检验结果选择最合适的模型进行计算。 在系统发育分析中,最大似然法(ML)和贝叶斯法(BI)是可以使用替换模型的两种算法,因此利用ML法或BI法重建系统发育树前,核苷酸替换模型的选择是必不可少的过程。 这里给出检验模型优劣的方法:似然率检验(Likelihood Ratio Test, LRT)用于比较两个模型对数据的拟合优度的一种统计检验方法;AIC值(Akaike Information criterion, AIC)是指Kullback-Leibler (K-L) 距离的估计值,K-L距离定义为一个模型被另一个模型替代后信息的丧失。因此,AIC值较小的模型更好;BIC(Bayesian Information Criterion, BIC):值越小,模型与数据拟合度越好;DT(Decision Tree,DT)将测量系统发生性能的相对分支长度误差整合到模型选择过程的方法。总结:BIC和DT具有较高的准确性和精确性,是目前最佳模型选择标准。对于核苷酸序列模型检验软件:ModelTest, JmodelTest, ModelFinder, Mega, ModelGenerator;对于氨基酸序列模型检验软件:ModelFinder, Protest, Mega, ModelGenerator。 进化树检验:Bootstrap 其中一个衡量树的好坏的方法就是看bootstrap的值,值越大越好。Bootstrap值是指根据所选的统计计算模型,设定初始值1000次,就是把序列的位点有放回的重抽样得到新的序列,再用相同的办法构树,如此让模型计算并绘制1000株系统发育树。如果原来树的分枝在后续构建的树中也出现了,就给这个分枝打上1分,如果没出现就给0分,这样给进化树打分后,每个分枝就都得出分值。系统发育树中每个节点上的数字则代表在1000次进化树分析中出现,有多少次最后一般换算成百分数。一般bootstrap的值>70%,则认为重建的进化树较为可靠。如果bootstrap的值太低,则有可能进化树的拓扑结构有错误,进化树是不可靠的。 因此,一般推荐用两种以上不同的方法构建进化树,如果所得到的进化树类似,且bootstrap值总体较高,则得到的结果较为可靠。通常情况下,只要选择了合适的方法和模型,构出的树均是有意义的,研究者可根据自己研究的需要选择最佳的树进行分析。
小结 比较以上几种主要的构树方法,一般情况下,若有合适的分子进化模型可供选择,用最大似然法构树获得的结果较好;对于近缘物种序列,通常情况下使用最大简约法,因为用的假设最少;而对于远缘物种序列,一般使用邻接法或最大似然法。 对于相似度很低的序列,邻接法往往出现长枝吸引(branch attraction)现象,有时严重干扰进化树的构建。对于各种方法重建进化树的准确性,Hall BG.( Mol Biol Evol 2005, 22(3):792-802)认为贝叶斯法最好,其次是最大似然法,然后是最大简约法。其实如果序列的相似性较高,各种方法都会得到不错的结果,模型间的差别也不大。再有贝叶斯的方法计算量巨大,速度太慢用的也不多。
|
【本文地址】