使用MNE编辑EEG脑电电极(脑电地形图)配置文件 (以SEED数据集的电极排布为例) |
您所在的位置:网站首页 › 绘制亚洲地形图,简单图 › 使用MNE编辑EEG脑电电极(脑电地形图)配置文件 (以SEED数据集的电极排布为例) |
使用MNE编辑EEG脑电电极(脑电地形图)配置文件 (以SEED数据集的电极排布为例)
|
电极位置坐标文件: 链接:https://pan.baidu.com/s/1ddMDSGxbSAPUVkRtlhHIuA?pwd=b3w5 提取码:b3w5 看到私信有人要1020文件,已上传至网盘 在使用MNE进行EEG信号的可视化操作时,往往需要导入对应电极的位置信息,MNE中有内置的常见电极布局系统,通过调用下面指令进行导入: # MNE自带1020系统电极位置 int1020_montage = mne.channels.make_standard_montage('standard_1020') int1020_montage.plot() # 可用的电极布局系统 ''' =================== ===================================================== Kind Description =================== ===================================================== standard_1005 Electrodes are named and positioned according to the international 10-05 system (343+3 locations) standard_1020 Electrodes are named and positioned according to the international 10-20 system (94+3 locations) standard_alphabetic Electrodes are named with LETTER-NUMBER combinations (A1, B2, F4, ...) (65+3 locations) standard_postfixed Electrodes are named according to the international 10-20 system using postfixes for intermediate positions (100+3 locations) standard_prefixed Electrodes are named according to the international 10-20 system using prefixes for intermediate positions (74+3 locations) standard_primed Electrodes are named according to the international 10-20 system using prime marks (' and '') for intermediate positions (100+3 locations) biosemi16 BioSemi cap with 16 electrodes (16+3 locations) biosemi32 BioSemi cap with 32 electrodes (32+3 locations) biosemi64 BioSemi cap with 64 electrodes (64+3 locations) biosemi128 BioSemi cap with 128 electrodes (128+3 locations) biosemi160 BioSemi cap with 160 electrodes (160+3 locations) biosemi256 BioSemi cap with 256 electrodes (256+3 locations) easycap-M1 EasyCap with 10-05 electrode names (74 locations) easycap-M10 EasyCap with numbered electrodes (61 locations) EGI_256 Geodesic Sensor Net (256 locations) GSN-HydroCel-32 HydroCel Geodesic Sensor Net and Cz (33+3 locations) GSN-HydroCel-64_1.0 HydroCel Geodesic Sensor Net (64+3 locations) GSN-HydroCel-65_1.0 HydroCel Geodesic Sensor Net and Cz (65+3 locations) GSN-HydroCel-128 HydroCel Geodesic Sensor Net (128+3 locations) GSN-HydroCel-129 HydroCel Geodesic Sensor Net and Cz (129+3 locations) GSN-HydroCel-256 HydroCel Geodesic Sensor Net (256+3 locations) GSN-HydroCel-257 HydroCel Geodesic Sensor Net and Cz (257+3 locations) mgh60 The (older) 60-channel cap used at MGH (60+3 locations) mgh70 The (newer) 70-channel BrainVision cap used at MGH (70+3 locations) artinis-octamon Artinis OctaMon fNIRS (8 sources, 2 detectors) artinis-brite23 Artinis Brite23 fNIRS (11 sources, 7 detectors) =================== ===================================================== '''之后可通过各类set_montage指令为自己EEG数据匹配上对应的电极位置信息,详细指令可参考MNE官方文档:https://mne.tools/dev/search.html?q=set_montage 但是有时候自己想要的电极位置并不能在上述内置的系统里面找到,或者内置的电极位置有错位(涉及到EEGLAB和MNE数据可能无法兼容性问题),像下图这样,用的是1020系统,但是电极位置排列看上去就很不舒服。这时候就需要自行去编辑电极位置信息,以获得更好的展示效果。
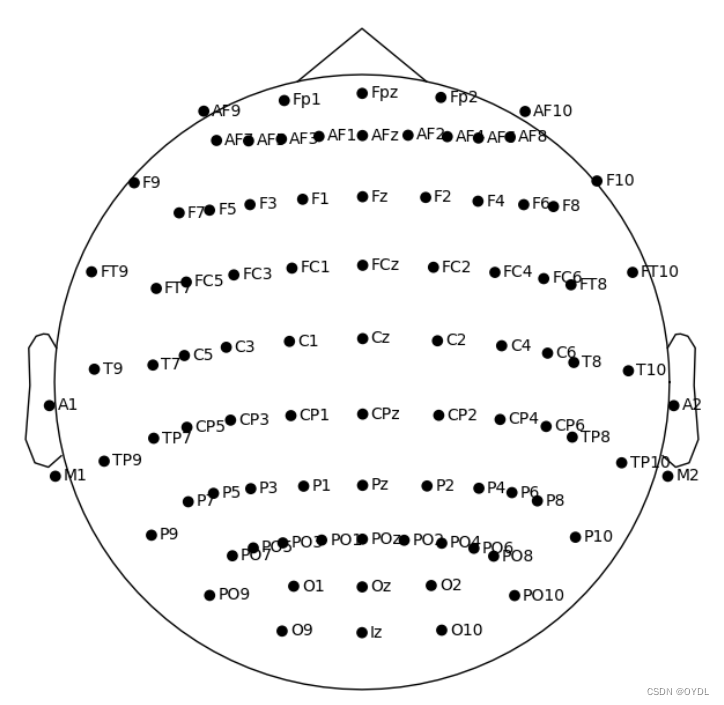
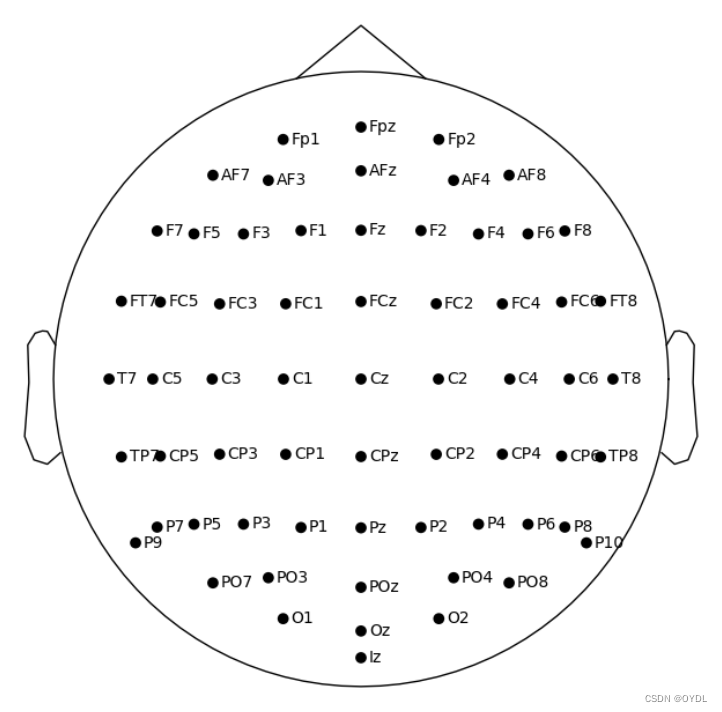
这里以SEED数据集的62电极系统为例 首先,MNE的内置电极位置系统中有一个biosemi64的,其包含的64个电极中,有58个与SEED作用的电极相同,我们只需要在此基础上增加和修改电极位置信息。下图中左图是biosemi64,右图是SEED的电极位置。
相比SEED,biosemi64中缺少了PO5, PO6, CB1, CB2这四个电极,多出了来了其他六个电极(多出来的电极可以不用理会,只要后续不调用对应的电极名称就不会有影响),同时部分电极位置也有些偏差,那么接下来就只需要再加入四个电极的信息并修改一下。直接上代码。 # 读取MNE中biosemi电极位置信息 biosemi_montage = mne.channels.make_standard_montage('biosemi64') print(biosemi_montage.get_positions()) sensor_data = biosemi_montage.get_positions()['ch_pos'] print(sensor_data) sensor_dataframe = pd.DataFrame(sensor_data).T print(sensor_dataframe) sensor_dataframe.to_excel('sensor_dataframe.xlsx') # 获取的除ch_pos外的信息 ''' 'coord_frame': 'unknown', 'nasion': array([ 5.27205792e-18, 8.60992398e-02, -4.01487349e-02]), 'lpa': array([-0.08609924, -0. , -0.04014873]), 'rpa': array([ 0.08609924, 0. , -0.04014873]), 'hsp': None, 'hpi': None ''' # 将获取的电极位置信息修改并补充缺失的电极位置,整合为1020.xlsx data1020 = pd.read_excel('1020.xlsx', index_col=0) channels1020 = np.array(data1020.index) value1020 = np.array(data1020) # 将电极通道名称和对应三维坐标位置存储为字典形式 list_dic = dict(zip(channels1020, value1020)) print(list_dic) # 封装为MNE的格式,参考原biosemi的存储格式 montage_1020 = mne.channels.make_dig_montage(ch_pos=list_dic, nasion=[5.27205792e-18, 8.60992398e-02, -4.01487349e-02], lpa=[-0.08609924, -0., -0.04014873], rpa=[0.08609924, 0., -0.04014873]) # 图示电极位置 montage_1020.plot()在上面的代码中,在获取sensor_dataframe.xlsx文件后,需要在文件中添加缺少的PO5, PO6, CB1, CB2这四个电极的三维坐标信息(多出的6个电极位置信息可删可不删,我个人是直接删掉了),然后再继续运行之后的代码。(注意代码运行顺序) 至于坐标位置怎么确定,只能根据邻近的电极位置信息不断进行调试。下图是获取的sensor_dataframe.xlsx文件内容截图。
然后你就可以获得对应的SEED数据集电极位置信息了。如下面左图(也就是上面代码的输出),右图是SEED官网给出的图。可以看出位置已经很接近了。
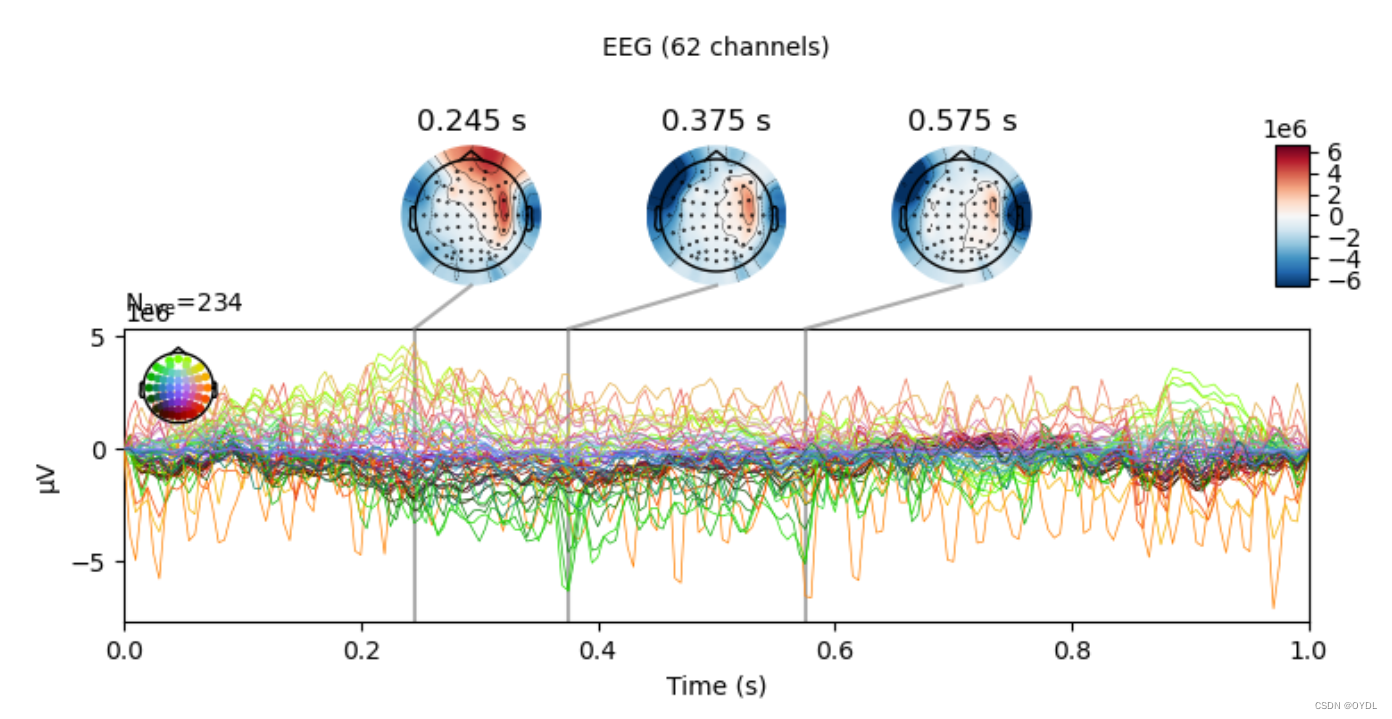
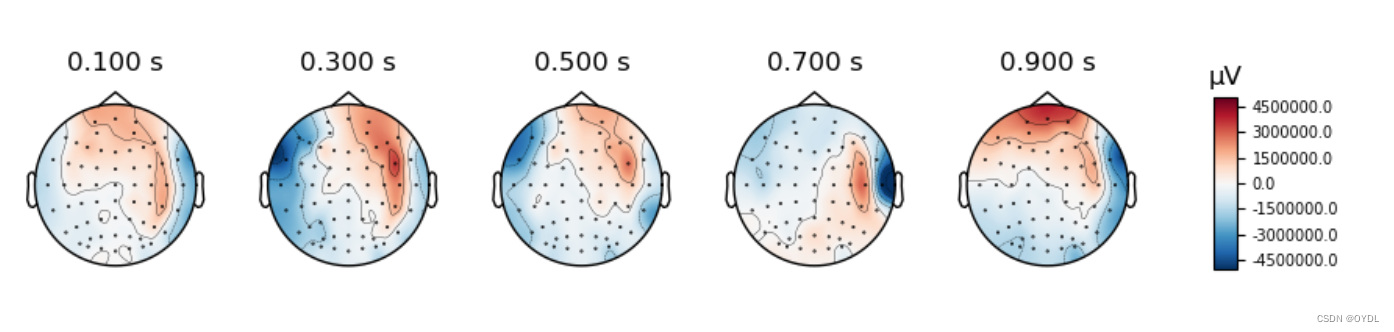
然后你就可以用制作好的montage_1020系统做各种图了,比方说像下图这些。
|
【本文地址】
今日新闻 |
推荐新闻 |