利用深度学习模型基于遗传算法(GA)寻求最优解 |
您所在的位置:网站首页 › 粗糙度数值含义全解图 › 利用深度学习模型基于遗传算法(GA)寻求最优解 |
利用深度学习模型基于遗传算法(GA)寻求最优解
|
文章目录
前言一、各种优化算法的优缺点二、深度学习模型+遗传算法1.思路2.代码部分
三、完整代码四、总结
前言
深度学习模型的训练前面的文章已经记录过,深度学习-LSTM预测未来值,训练好后的模型如何使用呢?其中一个用途就是用来寻求最优解,优化算法的种类也有很多,本文选用比较经典的遗传算法。 一、各种优化算法的优缺点遗传算法(GA):遗传算法具有良好的全局搜索能力,可以快速地将解空间中的全体解搜索出,而不会陷入局部最优解的快速下降陷阱;并且利用它的内在并行性,可以方便地进行分布式计算,加快求解速度。但是遗传算法的局部搜索能力较差,导致单纯的遗传算法比较费时,在进化后期搜索效率较低。在实际应用中,遗传算法容易产生早熟收敛的问题。采用何种选择方法既要使优良个体得以保留,又要维持群体的多样性,一直是遗传算法中较难解决的问题。 粒子群算法(PSO):具有相当快的逼近最优解的速度,可以有效的对系统的参数进行优化,PSO算法的优势在于求解一些连续函数的优化问题。最主要问题的是它容易产生早熟收敛(尤其是在处理复杂的多峰搜索问题中)、局部寻优能力较差等。PSO算法陷入局部最小,主要归咎于种群在搜索空间中多样性的丢失。 蚁群算法(ACO):在求解性能上,具有很强的鲁棒性(对基本蚁群算法模型稍加修改,便可以应用于其他问题)和搜索较好解的能力。蚁群算法收敛速度慢、易陷入局部最优。蚁群算法中初始信息素匮乏。蚁群算法一般需要较长的搜索时间,其复杂度可以反映这一点;而且该方法容易出现停滞现象,即搜索进行到一定程度后,所有个体发现的解完全一致,不能对解空间进一步进行搜索,不利于发现更好的解。 还有模拟退火算法、鱼群算法、鲸鱼优化算法等,可以参考这篇粒子群、遗传、蚁群、模拟退火和鲸群算法优缺点比较。 二、深度学习模型+遗传算法 1.思路整体的思路比较简单,就是将训练好的深度学习模型作为遗传算法的目标函数,关于如何训练存储深度学习模型,可以参考深度学习-LSTM预测未来值。 2.代码部分一、遗传算法的主要实现过程如下: 三、导入训练好的模型,我这里由于没有模型和代码不在同一个文件夹,修改了一下工作路径: folder = "D:\Desktop\First" os.chdir(folder) global model model = load_model('D:\Desktop\First\model.h5')四、这里使用的方法中,没有编码部分,之所以要编码是因为要与十进制相对应,但实际上不编码依旧可以进行对应。例如,以十位二进制数来表示【-1,1】之间的数,十位二进制数对应十进制可以表示0-1023,那么可以把【-1,1】这个区间切割成1023份,一份表示2/1023,就可以精确表示【-1,1】之间的数了。举个例子,0000000011对应十进制为3,占3份,则它在【-1,1】中表示3*([1-(1)]/1023)=0.005865。即基因0000000011表示0.005865。参考:遗传算法详解 附python代码实现。依上述,则有如下代码: '''解码''' def translateDNA(pop): # pop表示种群矩阵,一行表示一个二进制编码表示的DNA,矩阵的行数为种群数目 x_pop = pop[:, 1::2] # 奇数列表示X [::2]表示隔一个取一个 [::-1]可视为翻转操作 y_pop = pop[:, ::2] # 偶数列表示y # pop:(POP_SIZE,DNA_SIZE)*(DNA_SIZE,1) --> (POP_SIZE,1) x = x_pop.dot(2 ** np.arange(DNA_SIZE)[::-1]) / float(2 ** DNA_SIZE - 1) * (X_BOUND[1] - X_BOUND[0]) + X_BOUND[0] y = y_pop.dot(2 ** np.arange(DNA_SIZE)[::-1]) / float(2 ** DNA_SIZE - 1) * (Y_BOUND[1] - Y_BOUND[0]) + Y_BOUND[0] return x, y五、交叉变异部分就是模仿生物学中的父代母代基因对下一代的遗传,以及可能出现的变异,可以参考:遗传算法关于多目标优化python(详解)。 |
【本文地址】
今日新闻 |
推荐新闻 |
 二、首先还是导入所需要的库,并定义一些人为设定的关键参数:
二、首先还是导入所需要的库,并定义一些人为设定的关键参数: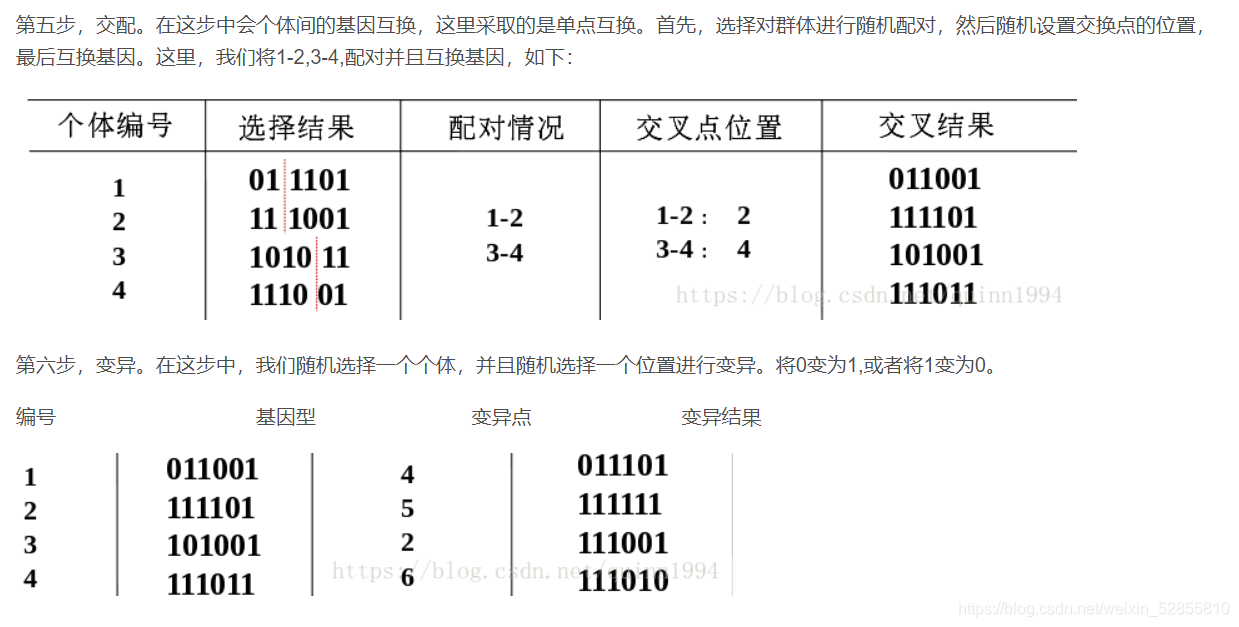 代码:
代码: