GWAS中的effect有什么用?计算PVE和PRS! |
您所在的位置:网站首页 › 拓扑学有什么用 › GWAS中的effect有什么用?计算PVE和PRS! |
GWAS中的effect有什么用?计算PVE和PRS!
|
大家好,我是邓飞,虽然,我早就知道GWAS分析中的effect值,就是数量遗传学的基因中的替换效应,但是一直没有仔细阅读相关材料。今天通过阅读数量遗传学的教程,理解了这个概念,真好。并且通过R语言模拟数据,验证了这个结论,纸上得来终觉浅,绝知此事要躬行! 同时,根据公式推导,可以更贴切的知道BLUP育种值的含义,我们为何要根据育种值进行选择,选择后会发生什么,加性效应,显性效应对于单位点SNP如何计算,对于PRS,MAS,GS的理解都是非常重要的。 1. GWAS中的effect这里,GWAS中的回归系数,effect,beta,都是一个意思。 因为GWAS分析中,单点检测,类似回归分析,effect就是SNP回归系数beta,p值就是SNP的P-value。 比如数据: 这里的M7位点,effect是1.394,p值是0.29。 首先,先看一下加性效应和显性效应的定义:
A2A2的平均值是:20 A1A2的平均值是:17 A2A2的平均值是:10 那么: 平均值是m = (10+20)/2 =15 加性效应的值是a = (20-10)/2 =5 显性效应的值是d = 17-15=2 2.2 期望和方差假定一个位点的次等位基因频率是p,主等位基因频率是q,而且该位点满足哈温平衡,所以: 整体均值为: 一种定义等位基因效应的方法,是利用后代群体的平均表现与随机交配群体均值的离差进行计算。以等位基因A1为例,把它视为配子,与群体中其他配子随机结合产生一个后代群体,其他配子基因型既有A1也有A2,它们的频率分别为p和q。因此,配子A1产生后代群体中的基因型有A1A1和A1A2两种,频率也分别为p和q。根据配子A1后代群体的基因型频率,就能得到后代群体的均值为pa+qd,从中减去随机交配群体的均值μ,就得到等位基因A1的效应 。类似地,我们还可以得到等位基因A2平均效应 。对于复等位基因,可用同样的方法定义它们的平均效应。 ❞ 2.4 替换效应(substitution effect)❝ 2.4 替换效应(substitution effect)❝育种过程中,当选择有利于某个等位基因时,常意味着有利等位基因对另一个不利等位基因的替换。因此,有必要研究等位基因的替代效应(effect of an allele substitution)。假定我们可以把随机挑选的等位基因A2变为A1,中选个体的基因型可能是A1A2也可能是A2A2,频率分别为p和q。把A1A2变为A1A1后,基因型值从d变为a,替换前后的效应变化为a-d;把A2A2变为A1A2后,基因型值从-a变为d,替换前后的效应变化为a+d。因此得到平均基因替换效应的表达式。 「基因平均效应和替换效应的关系:」  ❝ ❝上面资料来源王健康老师的PPT内容:第8章 随机交配群体的遗传分析 ❞3. 用基因型数据计算3.1 基因频率首先,看一下基因频率: p为:0.1693 q为:0.8307
这里,用AA,AT,TT平均表型值计算:
m:2.316 a:2.316 d:1.804 3.3 基因效应和替换效应注意,如果要手动计算的替换效应和回归分析计算的回归系数,需要满足哈温平衡。这里位点不符合哈温平衡,所以手动计算的替换效应和回归分析的beta值有差别。  4. 替换效应和回归系数等价推导 4. 替换效应和回归系数等价推导下面介绍一下相关的推导。 把SNP的分型转为0-1-2的X变量,将表型数据为Y变量,那么回归系数的公式可以推导为替换效应的组成。  上图中,X是编码为0-1-2的SNP,Y是每个基因型0-1-2的表型值。比如: 如果我们对value为Y,SNP为x,计算回归系数:b = cov(X,Y)/var(X),就可以推导为:b = alpha,截距为:u - 2palpha 结论:回归系数就是替换效应。 5 模拟数据演示计算公式:  5.1 小数据演示 5.1 小数据演示我们模拟一个符合哈温平衡的位点,p=0.5,q=0.5,n=12个: A2A2 = 10.7 A1A2 = 18.2 A1A1 = 31 那么加性效应和显性效应为: m = (31+10.7)/2 = 20.85 a = 31-20.85 = 10.15 d = 18.2 -m = -2.65 替换效应为:a + (p - q)  可以看出,计算出的回归系数为:10.16,截距为9.33,结果基本一致。 5.2 大数据演示#假定p为0.8,q为0.2,a=10,m=30,d=5, #那么分型为0的为20,分型为1的为35,分型为2的为40 #那么分型为0的频率为0.64,分型为1的频率为0.32,分型为2的频率为0.04 #总模拟个数为1000,标准差为5 rm(list=ls()) set.seed(123) AA=data.frame(SNP=rep(0,640),y=rnorm(640,20,5)) AT=data.frame(SNP=rep(1,320),y=rnorm(320,35,5)) TT=data.frame(SNP=rep(2,40),y=rnorm(40,40,5)) dd=rbind(AA,AT,TT) head(dd) str(dd) table(dd$SNP) mod=lm(y~SNP,data=dd) summary(mod) ##手动计算 mu=mean(dd$y);mu a=10;d=5;p=0.8;q=0.2 beta=a+(p-q)*d;beta beta_0=mu-2*q*beta;beta_0回归计算的回归系数和截距为: 截距:20.6 回归系数:12.9989 手动计算基因的替换效应: 截距为:20.61 回归系数为:13  一个个体的育种值,就是他的后代群体,相对于整个亲本群体的差异。比如一个个体的育种值是0.5,那就是说他的后代会比群体的整体平均值高0.3,如果育种值是0,那就是后代的平均值和群体一致。所以,我们要选择blup值大的个体,因为它的后代会高于群体的平均值。 因此,基因型A1A1、A1A2和A2A2的育种值分别为A11=2α1, A12=α1 +α2和A22=2α2 。统一起来,各种基因型的育种值表示为: A11 = 2*alpha1 A12 = alpha1 + alpha2 A22 = alpha2 注意,这里的alpha1是等位基因1的平均效应,alpha2是等位基因2的平均效应。 所以,这里,就可以理解为数量遗传学的替换效应就和GWAS分析的效应值联系到了一起。
相关阅读: 1,GWAS学习教程(快来领取 | 飞哥的GWAS分析教程),这个pdf是我将公众号的内容进行了汇总,更方便从头学习GWAS分析,里面配套了数据、代码和讲解,属于干货推荐的Number 1。 2,农学人如何入门数据分析资料汇总(飞哥汇总 | 入门数据分析资源推荐),里面推荐了免费的教程,包括编程、统计和专业书籍。 3,数量遗传学电子书下载(数量遗传学,分享几本书的电子版) 4,R语言电子书线上书籍推荐(学习R语言这几本电子书就够了!) |
【本文地址】
 用R语言拟合模型:
用R语言拟合模型: 下图用GWAS的GLM模型展示,两者结果是一致的。
下图用GWAS的GLM模型展示,两者结果是一致的。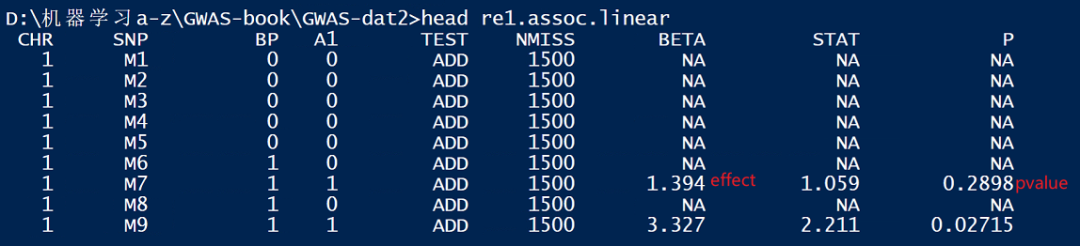

 举个栗子比如:
举个栗子比如: 整体方差:
整体方差:

 也可以根据AA,AT,TT的个数,手动计算:
也可以根据AA,AT,TT的个数,手动计算:
 「计算的结果:」
「计算的结果:」


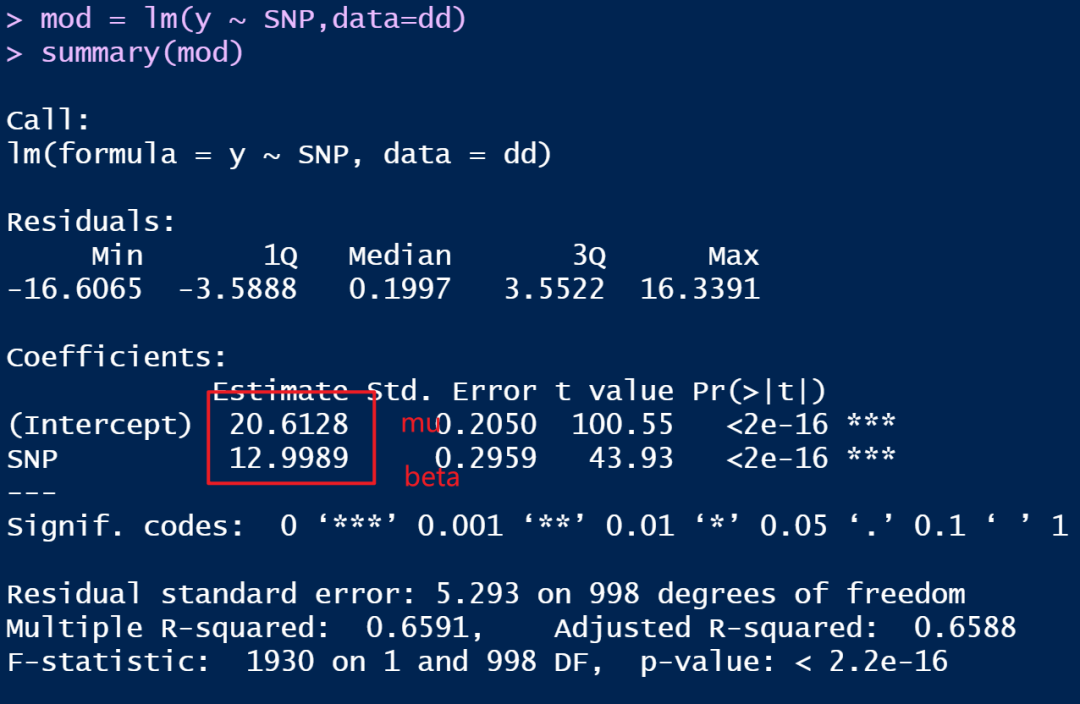
 两者结果完全一致。
两者结果完全一致。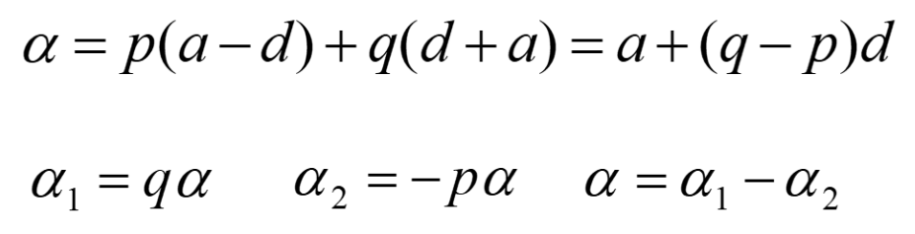 根据上面的公式,我们就可以根据每个位点的效应值,计算单个SNP的育种值,加性效应和显性效应。
根据上面的公式,我们就可以根据每个位点的效应值,计算单个SNP的育种值,加性效应和显性效应。