一文学会PCA/PCoA相关统计检验(PERMANOVA)和可视化 |
您所在的位置:网站首页 › 差异性检验和显著性检验的区别 › 一文学会PCA/PCoA相关统计检验(PERMANOVA)和可视化 |
一文学会PCA/PCoA相关统计检验(PERMANOVA)和可视化
|
方差分析中的元和因素
试验中要考察的指标称为试验指标,影响试验指标的条件称为因素,因素所处的状态称为水平 (通常用于3个或更多水平时;如果只有2个水平考虑T-test);若试验中只有一个因素改变则称为单因素试验,若有两个因素改变则称为双因素试验,若有多个因素改变则称为多因素试验。 方差分析就是对试验数据进行分析,检验方差相等的多个正态总体 均值是否相等,进而判断各因素对试验指标的影响是否显著;根据影响试验指标条件的个数可以区分为单因素方差分析、双因素方差分析和多因素方差分析。(来源于:百度百科) 方差分析中的因素方差分析中的因素通常是人为选定或可控的影响条件,如对样品的人为处理、样品自身的标记属性等。不可控因素如病人的心情、试验操作人的心情等一般不视为因素或不作为关注的因素;(还有一些不可控因素或通常认为不会带来很多影响的因素,如不同的取样时间、不同的RNA提取时间、提取人、细胞所处的分裂周期等;在某些情况下,如果我们记录了这些因素并且关心这些因素时,也会变为方差分析中的因素)。 举个例子,比如病人服用不同浓度药物后基因表达变化试验中: 基因表达是试验指标; 药物浓度是因素,假设有3个水平低浓度、中浓度和高浓度。 这就是单因素方差分析 (one-way ANOVA),比较病人服用不同浓度药物后基因表达的均值是否相等; 如果同时考虑病人的年龄的影响,则 年龄也是因素,有多个水平比如幼年、青年、成年、老年等。 这就是两因素方差分析 (two-way ANOVA),比较用药浓度和年龄对基因表达变化的影响,称为“主效应”影响;有时还需要同时比较浓度+年龄组成的新变量对基因表达变化的影响,称为“交互效应”影响。(如果只是比较浓度+年龄组成的新变量对基因表达变化的影响,就又是单因素方差分析了) 如果再考虑病人的籍贯、药物种类、吃药时间、病人Marker突变等的影响,就是多因素方差分析了。 方差分析中的试验指标试验中要考察的指标称为试验指标。在上面的例子中基因表达是一个试验指标,不过很笼统,默认为是单个基因的表达,称为一元方差分析。 那如果是关注两个基因或所有基因的表达变化整体是否有差异呢? 这就是多元方差分析,每组样本不是只包含一个试验指标而是多个试验指标。 表现在数据形式上: (一元)方差分析是比较多组向量的均值是否存在显著差异。 多元方差分析是比较多组矩阵的均值是否存在显著差异。 因此,比较多组样本整体基因表达的差异、多组样本整体菌群构成的差异,就需要多元方差分析了。 多元方差分析在统计学中,多元方差分析 (MANOVA, multivariate analysis of variance) 是一种对多个分组中检测了多个指标变量 (这里的变量等同于上面的指标;如每个样本中每个物种的丰度信息、每个样本中每个基因的表达信息)的样本整体均值的检验方法 。作为一个多变量过程,它在有两个或多个因变量时使用,并且通常会分别涉及各个因变量的显着性检验。它有助于回答: 自变量 (因素)的变化是否对因变量 (试验指标)有显着影响? 因变量之间有什么关系? 自变量之间有什么关系? 注: 对应上面 - 所有的因素都是自变量 (independent variable),而试验指标是因变量 (dependent variable)。这在看英文文献或不同教程时需要注意描述差异。 多元方差分析 (MANOVA, multivariate analysis of variance)的前提假设可类比于一元方差分析 (观测指标值的独立性、正态性、方差齐性) 数据独立性。 每个分组内的检测指标符合多元正态分布。 每个分组内的检测指标的协方差矩阵一致。 但在很多生物、生态和环境数据集中,多元方差分析的前提假设通常难以满足。 一些鲁棒性更强、对数据分布依赖更少的检验方法被提出来并且获得广泛应用,如ANOSIM (analysis of similarities), PERMANOVA (permutational multivariate analysis of variance) (也称为NPMANOVA, non-parametric MNOAVA), 和Mantel test。这些方法都通过一个样本间的距离矩阵或相似性矩阵构建ANOVA分析类似的统计量,然后对每组的观测结果进行随机置换来计算显著性P-value。对于单因素分析,对数据唯一的假设条件就是观察指标数据存在可置换性 (exchangeability)。 下面我们再介绍如何应用PERMANOVA来检验PcOA等的结果的显著性。 PERMANOVA基本原理PERMANOVA是多元方差分析的非参数变体。它用来比较多组观测样本的统计指标值的异同。 它利用距离矩阵(如欧式距离、Bray-Curtis距离)对总方差进行分解,分析不同分组因素或不同环境因子对样品差异的解释度,并使用置换检验对各个变量解释的统计学意义进行显著性分析。 目的是检测不同分组的响应变量如菌群构成是否有显著差异。因主要用函数adonis进行分析,有时也称为adonis 检验。 The goal of this test is to tell you if there are significant differences in your response variables among your groupings. 原始假设 (null hypothesis)是每组样本在其检测指标构成的检测空间中的中心点 (centroid)和离散度dispersion无差别。 计算出P值小于0.05时拒绝原假设,也就是不同组样品在检测空间的中心点或分布显著不同。 该检验需要预先计算试验样品在检测指标定义的多维空间的距离,如欧式距离、Bray-Curtis距离等。 The null hypothesis that the centroids and dispersion of the groups as defined by measure space are equivalent for all groups. A rejection of the null hypothesis means that either the centroid and/or the spread of the objects is different between the groups. 比如,对宏基因组检测的物种丰度数据进行PCA/NMDS/PCoA降维可视化后,不同组的样品之间存在一些重叠,那怎么判断这些组之间的样品构成是否存在显著差别呢? 这就需要用到PERMANOVA检验了,检验不同组的样品中心点是否重叠。 当然,PERMANOVA并不依赖于某种降维方法,而是依赖于距离矩阵。如果检测出p值大于0.05,表示不同组的物种构成或相对丰度没有显著差异。 PERMANOVA对检测数据的分布没有任何限制,也不受组之间数据协方差不同的影响,对多重共线性和很多0-值不敏感,其依赖的前提假设是: 每个对象的数据具有可交换性 (exchangeable) 可交换的对象(样品)彼此独立 每个样品的检测数据有一致的多变量分布(每组数据的离散程度相近) PERMANOVA分析等同于分组变量为解释变量矩阵的哑变量时的基于距离的冗余分析 (db-RDA)。
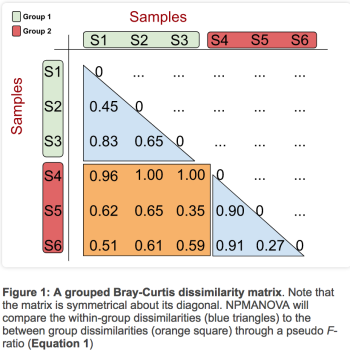
PERMANOVA测试的统计值是伪F值 (pseudo F-ratio),类比于ANOVA分析的F值。它的计算方式是不同组样品之间的距离(或距离的排序)平方和(图中黄色部分)除以同一组样品之间的距离(或距离的排序)平方和(图中蓝色部分),具体如下面公式。 更大的F值表示更强的组分离。通常这个值的显著性要比这个值本身的大小更有意义。
PERMANOVA采用数据置换的方式计算pseudo F-值的统计显著性,比较随机置换数据获得的pseudo F-值是否高于或等于实际观测到的值。如果多于5%随机置换计算的pseudo-F值高于实际观测值,则表示不同组的样品之间不存在显著差异 (p-value > 0.05)。 It is vital that the correct permutational scheme is defined and only exchangeable units are permuted. In nested studies, this would mean restricting permutations to an appropriate subgroup of the data set. At times, exact permutation tests either cannot be done, or are restricted to so few objects, that they are not useful. PERMANOVA 实战 (一)采用vegan包自带的一套数据(也解释了如何自己准备数据)看下PERMANOVA的具体代码和应用。 dune数据集描述dune是一套包含了20个样品和30个物种丰度数据的统计表。其格式是常见OTU表转置后的格式,每一行是一个样品,每一列是一个物种 (检测指标)。 library(vegan) data(dune) dim(dune) ## [1] 20 30 head(dune) ## Achimill Agrostol Airaprae Alopgeni Anthodor Bellpere Bromhord Chenalbu Cirsarve Comapalu Eleopalu Elymrepe Empenigr Hyporadi ## 1 1 0 0 0 0 0 0 0 0 0 0 4 0 0 ## 2 3 0 0 2 0 3 4 0 0 0 0 4 0 0 ## 3 0 4 0 7 0 2 0 0 0 0 0 4 0 0 ## 4 0 8 0 2 0 2 3 0 2 0 0 4 0 0 ## 5 2 0 0 0 4 2 2 0 0 0 0 4 0 0 ## 6 2 0 0 0 3 0 0 0 0 0 0 0 0 0如果我们有一个OTU丰度表,怎么转成这个格式呢? otu_table |
【本文地址】
今日新闻 |
推荐新闻 |