BioinfoTools全网最全的生信零代码网页工具 |
您所在的位置:网站首页 › 壁纸最全的网站 › BioinfoTools全网最全的生信零代码网页工具 |
BioinfoTools全网最全的生信零代码网页工具
|
GEO数据库作为最常用的数据资源,我们经常需要从这里获取想要分析的数据,以及用于验证的外部数据。然而数据下载后通常无法直接使用,需要进行探针合并,重复基因处理,标准化,以及临床信息获取等复杂操作,对于没有编程和生信基础的用户及其不友好。因此我们在BioinfoTools上为用户搭建了一件获取GEO数据的功能模块,将上述处理过程打包合并成一站式流程。接下来我们就演示下如何试用BioinfoTools下载GEO数据。 方法1:优点:操作简单方便,无脑粘贴GSE编号和GPL编号即可 缺点:下载时间较慢,国外服务器不稳定,偶尔下载失败需要重新提交任务 我们以GSE50901为例,进入网站后找到GPL探针编号 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE50901  然后把GSE编号和GPL编号输入到下方位置,点击提交即可 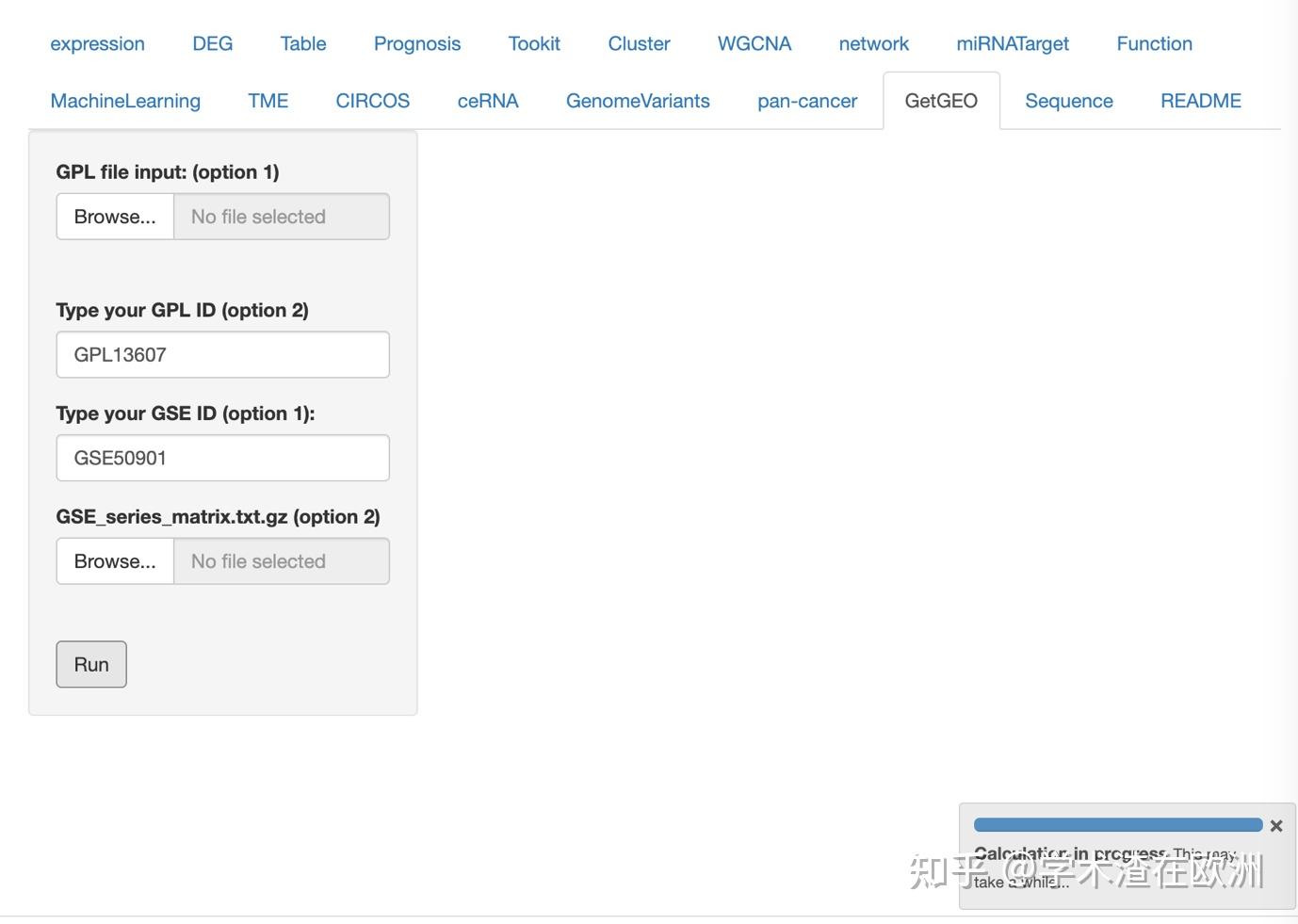 方法1操作运行界面方法2: 方法1操作运行界面方法2:优点:跳过下载时间,直接进入探针合并,标准化等步骤,更稳定,快速 缺点:需要手动下载series文件 这里需要我们手动下载series matrix file,点击下载  手动下载表达谱链接 手动下载表达谱链接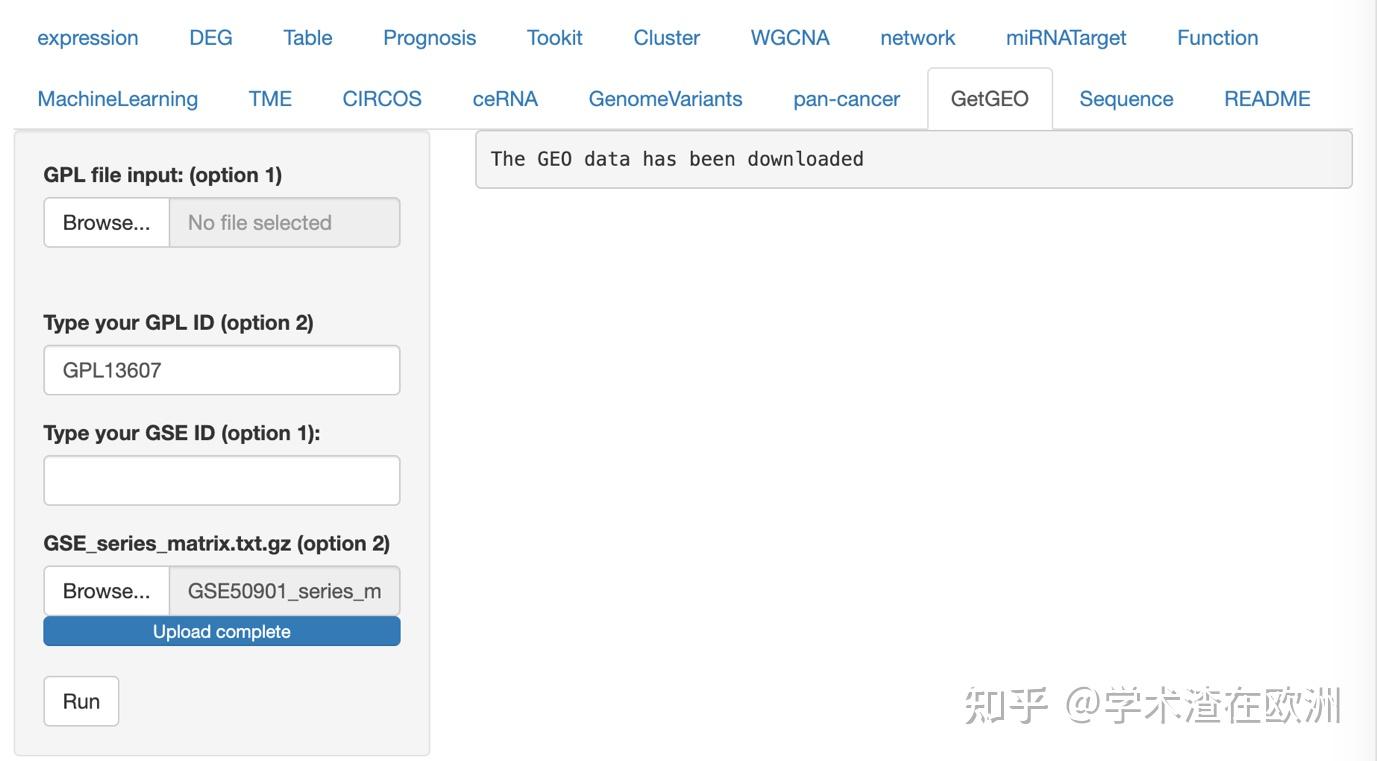 方法2操作运行界面 方法2操作运行界面然后将压缩文件上传到GSE_series_matrix.txt.gz (option2)的位置,点击运行即可。 方法3:优点:更灵活,不局限于GEO数据 缺点:需要手动整理表达谱和探针的输入文件 首先我们需要整理一套表达谱数据,第一列是探针,第二列开始是样本,格式如下  需要整理成上图的表达谱格式 需要整理成上图的表达谱格式注意,series文件最后一行的注释也要删掉哦~~~ 然后我们整理探针文件,需要包含两列,第一列是探针id,第二列是对应的基因名,前两种方法自动注释只能转换成gene symbol,这里可以灵活的选择任何基因名,例如GB_ACC。  需要保留探针id和基因名两列 需要保留探针id和基因名两列下载下来后,我们选择第一列探针id,以及感兴趣的基因名,GPL文件里包含GB_ACC和genename,我们这里如果选择GB_ACC,只需要保留前两列,其他的列删掉即可。  最后把整理好的表达谱上传到expression profile, 把包含两列的探针数据上传到GPL. 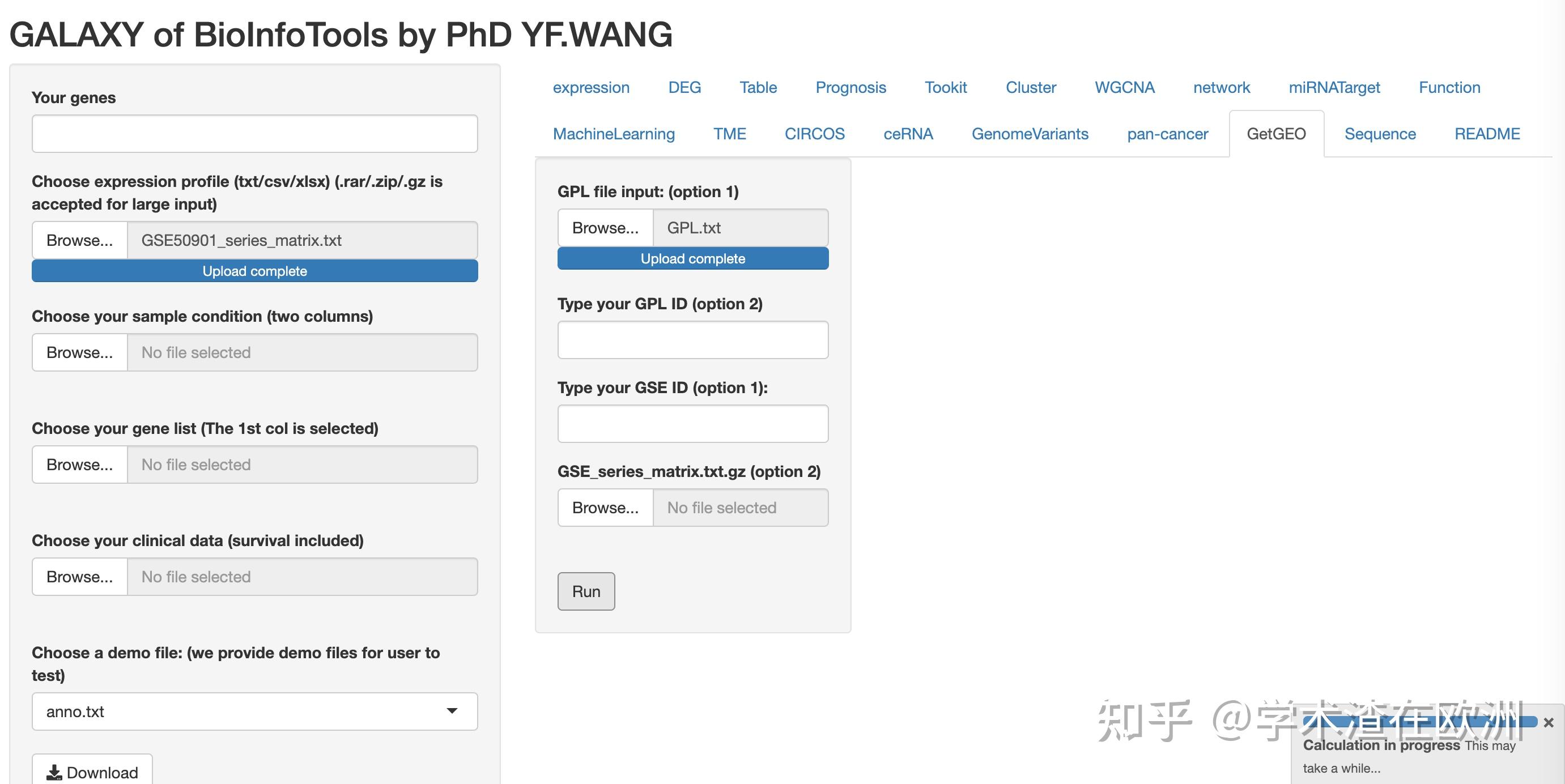 方法3操作运行界面 方法3操作运行界面运行结束后我们得到的结果包括原始数据表达谱,标准化后的表达谱,以及临床信息。 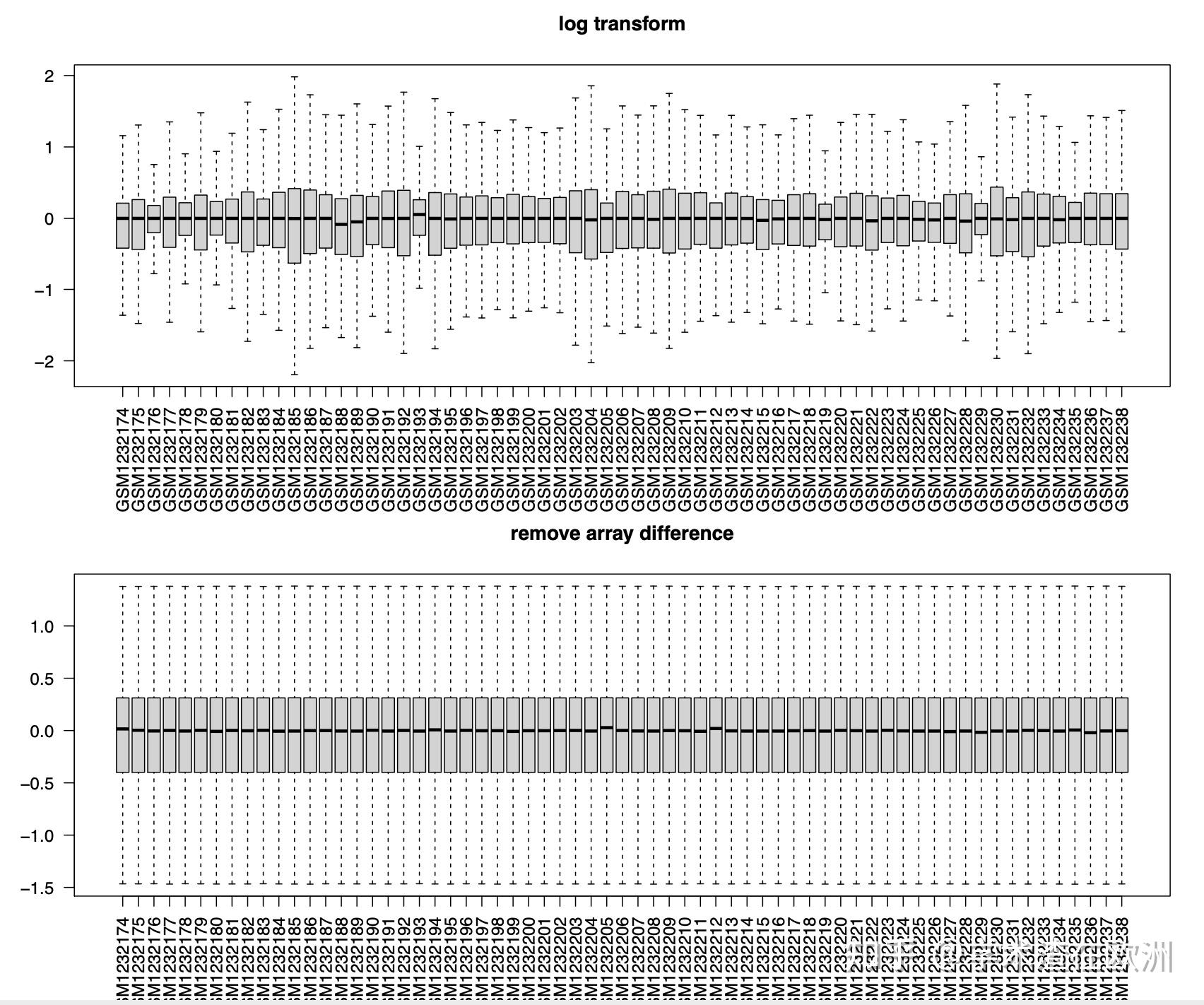 标准化前后对比图 标准化前后对比图 bioinfotools获取的临床信息 bioinfotools获取的临床信息网站获取的临床信息里,很多是从GEO网站看不到的隐藏信息哦,感兴趣的话可以私信作者,告诉你这些临床信息藏在哪里:) 最后再讲个小tips,有时候我们想从探针文件里获取探针id和基因名的时候,发现GPL文件并没有提供基因名,但是提供了探针的序列和基因组坐标。很多时候小伙伴就挠头不知道怎么继续处理了,这里我们可以参考另一个bioinfotools的模块介绍,肯定会对你有帮助的。 https://zhuanlan.zhihu.com/p/620133504 以上就是GEO一键下载模块的教程内容,怎么样,是不是很简单呢。感兴趣的小伙伴快通过下方链接领取账号用起来吧。 http://biowinford.site:3838/trial/ 更多视频教程请跳转B站【学术渣在欧洲的个人空间-哔哩哔哩】 https://b23.tv/ycWnvFe |
【本文地址】
今日新闻 |
推荐新闻 |