为什么要做单细胞测序 |
您所在的位置:网站首页 › 单细胞测序技术定义 › 为什么要做单细胞测序 |
为什么要做单细胞测序
|
普通组织尚且如此,如果研究目标是肿瘤组织这样的细胞突变大熔炉,细胞类型更是多种多样。而组织中的各类细胞往往是离散分布在整个组织中,用常规的切割方法是无法分离的。所以,常规转录组本质上是将组织中的各种细胞混合进行检测,最终得到的是整个组织的均值。
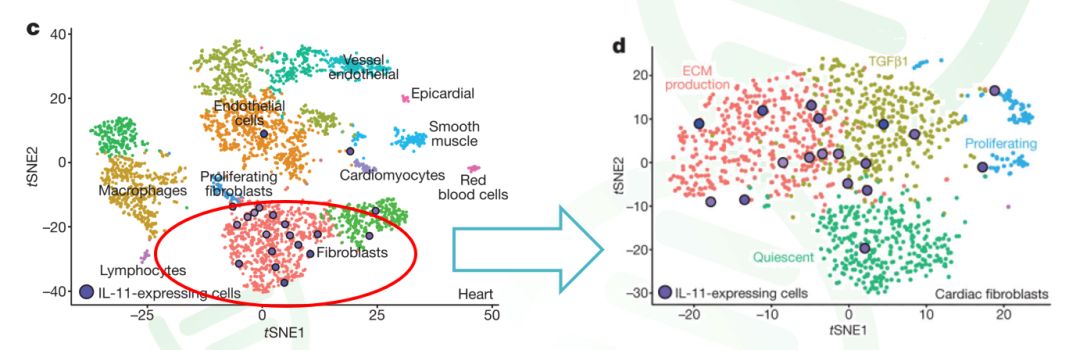
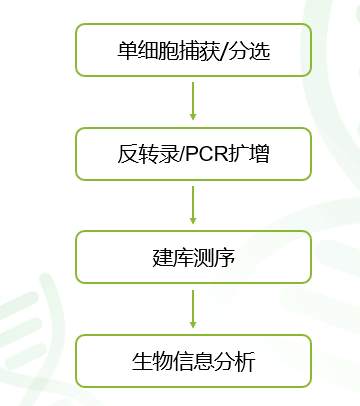
图1 小鼠心脏中的细胞类型(左)以及成纤维细胞可以进一步分为若干子亚群(右) 问题往往就出在这里:常规转录组检测的是样本中所有细胞的均值,而我们研究目标(例如,某种疾病的发生机制)却可能只与其中的某种细胞相关。例如,如果研究心脏纤维变性,最佳方案应该只针对心脏成纤维细胞进行检测和比较,而普通转录组往往是检测整个心脏组织。如果我们研究胰腺的胰岛素分泌变化,那么理论上应该针对胰岛α细胞进行检测比较,而普通转录组可能是对整个胰腺进行检测。 这种期望(研究范围应该限定在特定类型细胞中)与现实(只能检测各种混合细胞的均值)巨大裂隙,导致了以往的发育、疾病、环境应答等的组学研究只能得到一个概要性的结论,而难以在更加精细地在细胞尺度给出一个清晰的解答。要弥补这个裂隙,只能使用单细胞检测的方法。 单细胞转录组测序技术发展史 那么为什么单细胞测序技术为什么这两年才火起来呢?下文,我们以单细胞转录组测序技术为例开展探讨。单细胞转录组主要包括以下四个步骤。其中非常关键的一点就是如何进行单细胞的捕获/分选,这是决定单细胞检测成本和通量的关键步骤。
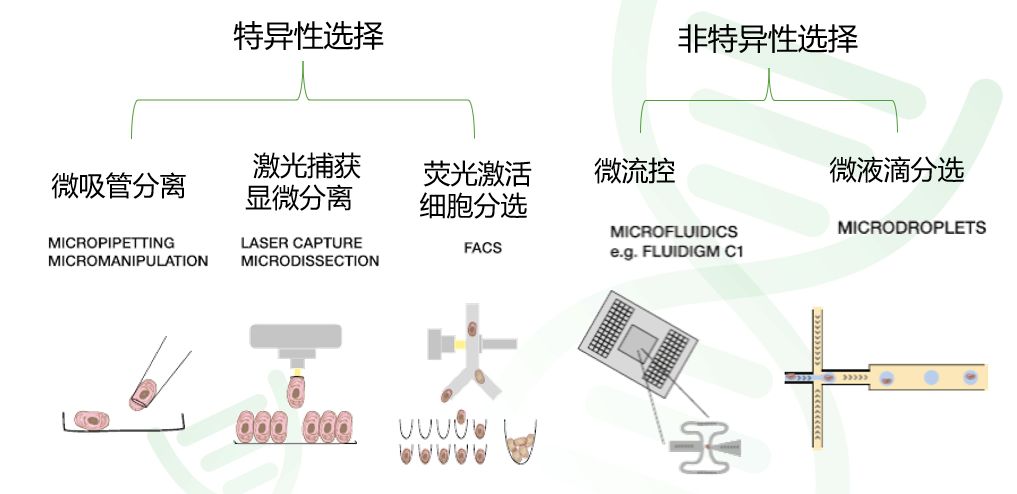
图2 单细胞转录组的主要步骤 在细胞分选的方法里,主要包括特异性分选和非特意性分选两类方法(图3)。两类方法的关系有点类似Qpcr定量(只针对特定目标基因进行检测)和转录组测序(不需要预期目标基因,直接对样本中所有的基因进行检测)。 特异性细胞分选类似Qpcr,该策略是从目标组织/样本中,用特定标志(例如:荧光标记物或细胞形态)对特定目标细胞进行挑选,然后对目标细胞开展测序。所以特异性选择的方法通常通量很低。 非特异性选择的方法则通常都是高通量的方法,一般是用特定的技术随机从样本中(通常为单细胞悬液)捕获的大量细胞单体,然后直接平行对大量细胞进行独立的测序,再从大量单细胞数据中寻找自己感兴趣的细胞类型进行后续分析。这些技术的进一步比较请见表1。
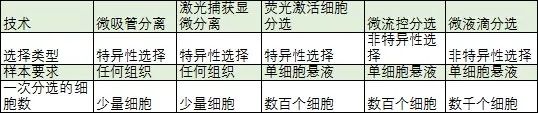
图3 两大类单细胞分选技术示意图 表1 五种常见的单细胞分选技术的比较
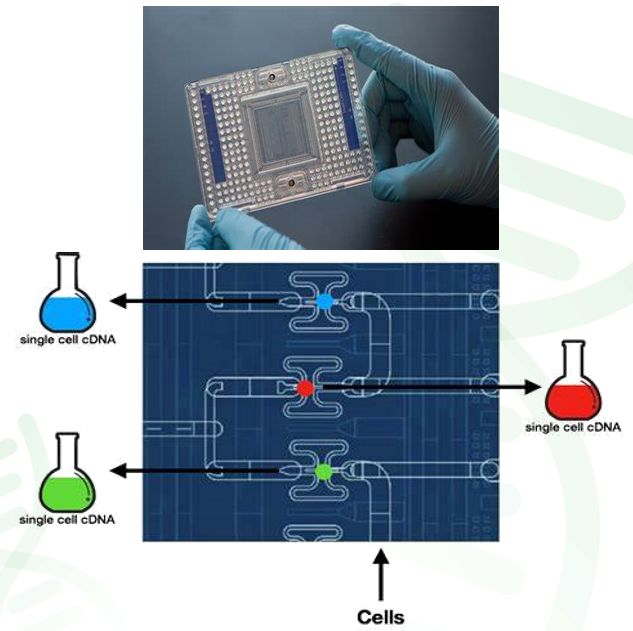
特异性分选类技术 单细胞检测,本质上是对组织以细胞为单位开展调查。早期出现的技术都属于特异性分选技术,通常就是基于荧光分子标记或细胞形态筛选到目标细胞,然后对目标细胞开展扩增和测序。针对目标细胞进行扩增和测序的方法,最有代表性的方法要属SMART-seq。这类策略有很多的局限,主要体现在: 1 细胞分选的效率低 一次分选只能筛选个数有限的一种或几种细胞。而组织中,可能有几十种细胞。 2 细胞分选依赖已知的标记 这意味着很难在已有的研究基础体系之外,发现新的细胞类型。 3 检测成本非常高 因为单细胞研究本身是类似群体调查。如果你要全面调查某个组织,一个组织有十几种甚至几十种细胞,每种细胞你又需要至少独立检测几个到几十个重复。考虑到一个细胞测需要一次单细胞转录组测序(例如,SMART-seq),那么开展一次系统的研究需要投入大量单细胞分选的精力和至少几百个SMART-seq的费用(SMART-seq费用又高于普通转录组)。 这个费用对于很多团队来说是难以承担的,所以早期单细胞转录组技术一直难以广泛开展。 非特异性分选类技术 直到高通量的非特异性分选类的技术出现,才彻底降低了单细胞转录组研究的成本,代表性的方法包括微流控技术和微液滴技术。 Fluidigm技术 微流控代表性的技术是Fluidigm公司的微流控芯片Fluidigm C1。Fluidigm C1芯片就像一个电路,单细胞悬液导入后,单个细胞会被引入独立的腔室。一张芯片有96个腔室,所以一次最多允许96个细胞平行独立进行裂解、反转录和PCR扩增。 反转录扩增过程中,每个细胞的cDNA会被接上特异的接头序列,保证后续混合测序后还能重新独立拆分每个细胞的数据。Fluidigm C1芯片由于可以平行进行96个细胞的建库测序,所以相比SMART-seq类的技术,已经大大降低了成本。 但如果进行目标组织细胞图谱全景扫描这样的研究,一般要求检测至少数千个甚至几十万个细胞。而Fluidigm C1芯片通量只有96个细胞,则意味着要进行多次芯片捕获,这会大大提高芯片耗材的投入成本。因此,我们还需要更高通量的技术。
图4 Fluidigm C1芯片的单细胞转录组原理示意图 10X genomics ScRNA-seq 下来就介绍一下10X genomics单细胞转录组测序的原理(下文简称 10X ScRNA-seq)。相比Fluidigm类技术使用实体芯片作为隔离单个细胞的载体,10X genomics技术则是使用液滴作为隔离单细胞的载体,具体实验过程如下图。
图5 10X genomics ScRNA-seq的原理 如图5上半部部分,展示了10X ScRNA-seq包括了细胞悬液制备、文库构建、测序、数据分析、可视化等步骤。其中文库构建包含了细胞分选这个步骤,是使用10X genomics仪器完成。由于该仪器要求细胞在液相中实现流动分选,所以要求投入的样本必须为细胞悬液(组织样本可以通过胰蛋白酶消化制备为细胞悬液)。文库构建的具体过程可以看图5下半部分,这个过程可以分为4个步骤: A:准备好10X 官方提供的微磁珠。这些磁珠上都连接着后续反转录所需的接头、barcode、UMI等序列。 B:磁珠进入10X genomics芯片。磁珠在可以管道中流动(这里管道中是水),会经过两个“十字路口”。在第一个“十字路口”与另外一个管道交叉。新加入的管道里流动是细胞和反转录扩增所需的试剂。经过这个“十字路口后”,一部分磁珠就吸附上了细胞,且整个液体环境富含后续反应所需的酶。 在第二个“十字路口”,新加入的管道里流动的是油。油和水混合,就会形成乳浊液(油包裹水滴的环境)。由于磁珠是亲水的,所以一个个磁珠以及其吸附的细胞就就被包裹在水滴里。 C:在这些油包裹的水滴里,将完成细胞裂解、反转录、扩增等步骤。 D:每个细胞的cDNA完成扩增,并携带这独一无的接头,等待进行后续测序。 基于这样的步骤,通过合理控制每次输入的细胞数,最终一张10X genomics芯片可以检测的细胞数一般控制在3000~8000个之间。而且因为检测的细胞数巨大,几乎保证每类细胞至少几十个甚至成百上千个重复。那么每个细胞检测的数据量大概只要5万条左右的reads就足够了。 最终均摊下来,每个细胞检测转录组的费用仅仅是几块钱人民币。这相比之前的SMART-seq数千人民币的费用比,已经大大降低,因此10X ScRNA-seq让普通实验室以较少投入开展大规模单细胞转录组研究提供了可能。 小结 在互联时代,速度的提升就是革命性的变化(例如从4G到5G的变化),在生物学领域也是类似的。10X genomics这样类型的技术出现,使万级别的单细胞群体研究成为成本可接受的事情,这就是一种革命性的变化。 当然,与10X genomics同时期出现的以万为单位的高通量技术还有若干种,例如,SPLIT-seq、sci-RNA-seq等。但10X genomics技术还有一个优势就是系统稳定性高,从而保证实验员通过短期培训后就能稳定完成大规模单细胞分离这样复杂的操作,从而保证结果稳定性,这也是这个技术大规模得到应用的重要原因。
当然,检测细胞数的增加也会导致数据分析复杂度的大大提高(相比常规转录组)。后续数据的个性化分析和解读,对10X ScRNA-seq就显得尤为重要。如果你对10X ScRNA-seq数据的分析以及后期个性化挖掘感兴趣,也可以选择参加我们6月20~21号两天的培训班。 详细课表>> 培训地点:基迪奥公司(广州大学城青蓝街) 培训时间:2019年6月20-21日 报名费:2000元(交通和食宿费用自理) 报名费可以转账或现场刷公务卡 客服:020-39341079 报名方式 2、发送姓名、单位、电话到邮箱[email protected],主题注明10x单细胞测序培训班返回搜狐,查看更多 |
【本文地址】
今日新闻 |
推荐新闻 |