单细胞(single cell)分选平台比较(10X Genomics,BD Rhapsody,Fluidigm C1) |
您所在的位置:网站首页 › 单细胞建库原理 › 单细胞(single cell)分选平台比较(10X Genomics,BD Rhapsody,Fluidigm C1) |
单细胞(single cell)分选平台比较(10X Genomics,BD Rhapsody,Fluidigm C1)
|
相关产品
BD Rhapsody 单细胞分析系统[点击打开]
Single Cell Sequencing技术,即利用优化后的高通量测序技术(NGS,Next Generation Sequencing)分析每一个单个细胞的序列信息,从而更高分辨率地揭示细胞间的细胞差异以及其在微环境中的功能情况。 那么问题就来了: 1. 如何获得单个细胞?如果无法获得单个细胞这一切都是不成立的。 2. 如何获得大批量 的单个细胞?单组织细胞群体在10E6以上,如果被检测的测序细胞数量过小精确度不足。 3. 如何低成本地获得大批量单个细胞?单细胞测序成本非常高,尤其是需要测2000~3000个以上的细胞的时候,如果单个细胞的分选成本也很高,技术根本无法普及。 所以,正因为这些问题的存在使得高通量测序在单个细胞测序应用中的普及度一直不足,直到几个**性技术的诞生。
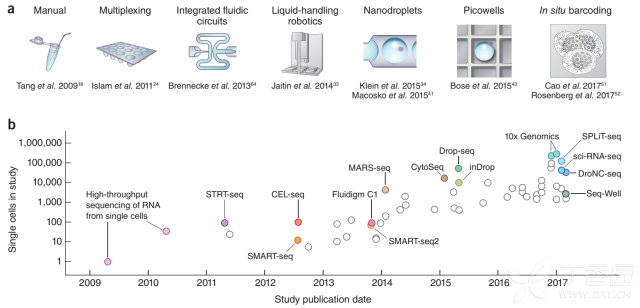
从上图中我们可以看到,2010年-2014年之间,单细胞测序领域采用的主流技术的细胞通量都在100以内,哪怕是最为先进的Fluidigm C1平台也是如此。因此我们在2014-2016年间看到的大量Single-Cell Seq的高分文章的单个细胞测序数量一般都在1000以内,因为这1000个细胞代价极大。 Fluidigm C1平台,是最早被应用于Single Cell领域的商业化分选技术,基于富鲁达(Fluidigm®)的微流控技术,大约在几个小时中可以捕获96个左右的细胞,进行各类的Single-Cell测序建库。当然,通量还是比较小。但不可否认的是,现在很多非RNA类Single Cell测序,仍然在采用这样的技术,如Single Cell DNA-Seq,Single Cell ATAC-Seq等,都是采用此技术进行单细胞分选后再分别用不同试剂盒建库。所以可以说,至少在10X和BD,或者其他企业推出有效的Single DNA测序技术之前,C1仍然会是市场上的重要组成部分。
引用自:http://cn.fluidigm.com/products/c1-system 令人亢奋的是,在2014-2015年间几个**性技术诞生了,仿佛寒武纪大爆发一般,MARS-Seq、CytoSeq、Drop-Seq、inDrop技术在这两年中扎堆出现,真正引爆了Single-Cell这个领域。而在2017-2018年,商业化的测序平台(10X Genomics®, BD Rhapsody®),逐渐浮出水面,才最终让Single-Cell测序技术走进了民用市场。
10X Genomics
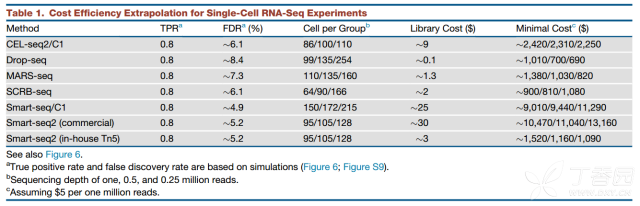
BD Rhapsody 从成本上来说,基于C1类的捕获技术的代价非常大,单细胞捕获成本在9-30美元不等,而且这还是一个没有关税、没有代理商加价的价格。2000-3000细胞需要18000-100000美元不等的捕获费用,可以说非壕不可用的技术。因此暂时就不做详细讲解了。
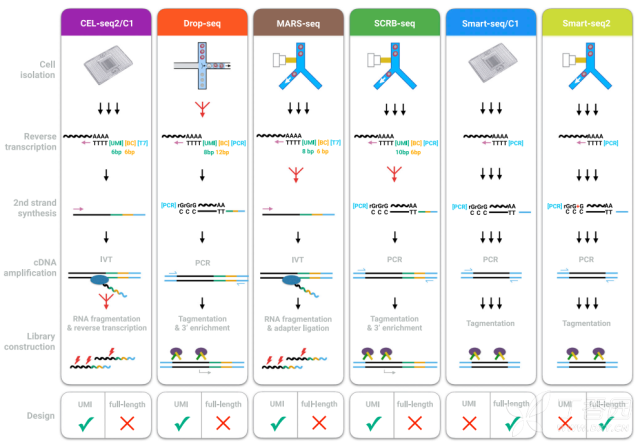
Ziegenhain C, Vieth B, Parekh S, et al. Comparative analysis of single-cell RNA sequencing methods[J]. Molecular cell, 2017, 65(4): 631-643. e4. 主要讲讲两个现在的主流技术,BD Rhapsody以及10X Genomics。从诞生年代来看,两者不分伯仲,10X Genomics起源自Drop-Seq技术,得益于哈佛大学的研究所的工作。
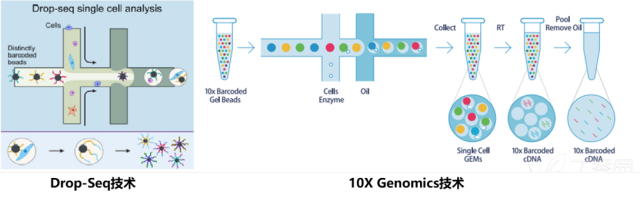
从Drop-Seq技术到10X Genomics技术 BD的Rhapsody平台,最早可以追溯到2015年Single Cell Cytoseq技术,可以说是完全同时代的技术。2017年BD也携一篇研究脑神经的Nature Article将该商业化技术发布了。(Birey F, Andersen J, Makinson C D, et al. Assembly of functionally integrated human forebrain spheroids[J]. Nature, 2017, 545(7652): 54.)
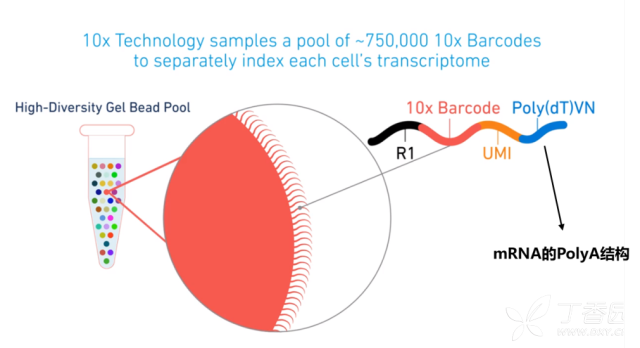
10X Genomics和Drop-Seq具有类似的技术原理。从横向孔道中逐一输入凝胶微珠,第一纵向孔道输入细胞,凝胶微珠与细胞碰撞后会吸附在凝胶微珠上,并通过微流控技术,将之输入到第二纵向孔道,即油相孔道中。这时候,就形成了一个个油滴,最终输出并收集在EP管中。每一个油滴中会落入一个细胞以及一个凝胶微珠,那么在每一个凝胶微珠中上长满了不同的Cell Barcode和UMI Barcode连接形成的序列,再加上一端PolyT的抓手,构成我们的捕获凝胶微珠。而这个凝胶微珠抓手就会使用oligo dT抓住mRNA构建文库。
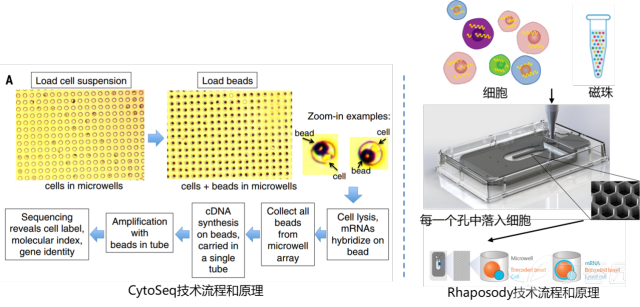
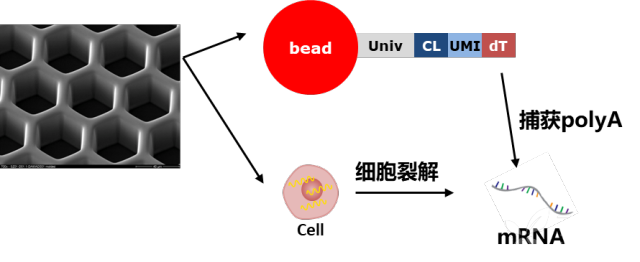
10X Genomics凝胶微珠设计 所以一个油滴=一个单细胞=一个凝胶微珠=一个RNA-Seq,可以说这就是10X的基本技术原理。 然而市场并不只有一家Single Cell的商业化捕获平台,另外还有BD这个老牌的抗体巨头的Rhapsody平台。这个平台又是依托什么技术,和10X Genomics技术存在什么区别呢? BD的技术不再采用利用微流控孔道射出细胞和射出的磁珠碰撞的过程,进行单细胞捕获的技术,转而采用CytoSeq特有的蜂窝板技术。该技术用20W+的微孔(该数量级远大于Input细胞数量),保证单孔中的单细胞捕获。同时避免了10X中存在的概率碰撞影响捕获效率的问题,采用微孔捕获相对会有更好的捕获效率(来自两家企业商业宣传资料比对),保证Input细胞的全面使用。对于Input细胞的量更少的情况,可以基于10E5的细胞总量开始进行前期处理以及后期捕获操作。(烈小冰在之前的实验技术分享里提到过 Single Cell前处理会丢失一些细胞哦!) 而在细胞捕获完成后,进行细胞裂解后,同样的也进行细胞中RNA polyA序列的抓取工作。
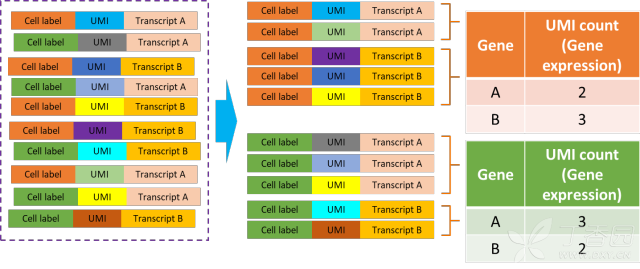
BD Rhapsody 单细胞mRNA抓取过程 所以一个微孔=一个单细胞=一个磁珠=一个RNA-Seq,可以说这就是BD的基本技术原理。 从现有数据来看,我们很难比较两种技术的优劣,但是从现有的文章发表情况来看,两种技术都在主流高IF期刊上有可信的数据发表,并且都与Smart-Seq数据的结果存在比较,因此两种技术都是可用可信的大规模Single Cell RNA-Seq技术。从发布时间来看,10X发布更早,而BD发布在其1年之后,这点上10X Genomics的设备稍占优势。而在捕获效率和有效性来看,BD Rhapsody的捕获效率更高较占优势。 从技术原理上分析两种Single Cell测序技术,都是基于UMI(Unique Molecular Identifier)+ CL(Cell Label)的技术,与传统Smart-Seq + Fluidigm的C1技术比较起来,引入UMI技术(这个我们会在分析部分简单介绍)进行表达定量,使得大规模scRNA-Seq的基因表达定量结果几乎不会受到PCR Bias影响,从而更精确。
图注:如果一个基因具有序列一致的UMI序列的测序Reads,那么这条Reads其实PCR Bias 相对于Smart-Seq技术在细胞数量上的问题,BD和10X这两个平台普遍提供的细胞数量都在1-6K不等的测序细胞量,这个量级的测序细胞量已经可以说能够完全覆盖绝大部分组织的Single Cell群体类型。因此,这两种技术对于基于基因表达进行准确细胞分型上,无论是从细胞数量上来说还是表达检测可靠性来说,都会更实用。同时依托Random Cell Label技术,使得其对于NovaSeq、HiSeq X Ten、Hiseq 4000等设备存在的Index hooping现象,相对于Smart-Seq数据来说,影响几乎可以忽略不计(10X Genomics官方网站曾给出hooping测试结果,显示无影响。https://kb.10xgenomics.com/hc/en-us/articles/115002052846-Index-hopping-on-the-HiSeq-4000-and-its-effect-on-10x-Single-Cell-libraries)。
Ziegenhain C, Vieth B, Parekh S, et al. Comparative analysis of single-cell RNA sequencing methods[J]. Molecular cell, 2017, 65(4): 631-643. e4. |
【本文地址】
今日新闻 |
推荐新闻 |