U |
您所在的位置:网站首页 › cpu尺寸大小一样吗 › U |
U
|
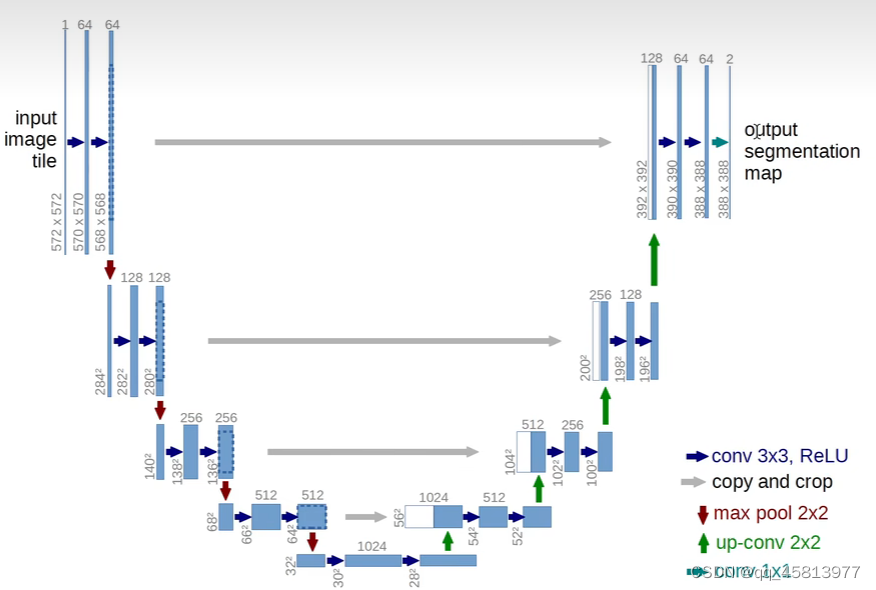
目录 前言 U-net简介 U-net网络架构理解 输入与输出说明 下采样 上采样以及输出 U-net的特点 谷歌Colab复现基于U-Net的眼底图像血管分割 1.环境配置 2.数据获取及其处理 3.代码框架 3.1 数据预处理: 3.2 数据增强: 3.3 U-net网络代码: 3.4 代码运行结果 总结 前言本文只是记录自己的学习过程,并不是面向技术向的硬核文章。 U-net简介U-net首见于这篇论文U-Net: Convolutional Networks for Biomedical,距今7年,算是比较老的模型了,但是它的引用量突破了四万,由此看来,它的重要性不言而喻。U-net初始论文的标题就告诉我们,它是一个用于医学图像分割的卷积神经网络。在医学领域,如果仅仅对图像进行分类,这是不够的。医生在对病人进行医疗诊断时,需要综合更多的信息来得出病情的判断。 所以,U-net做的事就是,在已有图像类别的基础上,对图像进行进一步分类---即像素这一级别的分类(图像分割),定位每个像素的类别,使图像中解剖或病理结构的变化更加清晰,最后输出根据像素点的类别而分割好的图像。 U-net网络架构理解论文中的U-net网络架构如下:
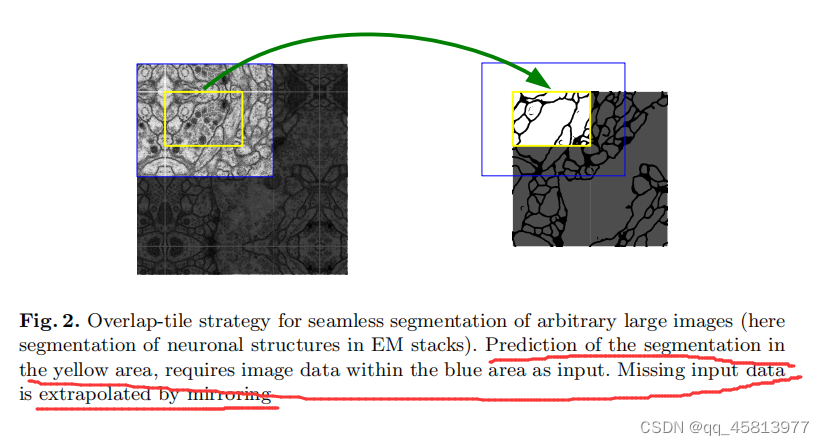
可以看到图中U-net的网络结构呈U形,左半部分我们称之为编码器,右半部分我们称之为解码器。编码器将图片不断压缩,分辨率变得越来越低,解码器将图像分辨率不断还原,分辨率逐步升高。下面我们针对上图,对网络操作流程进行说明。 输入与输出说明我们的输入图像大小为572,572单通道,输出为388,388二通道。图像分割不应该原图和输出图片大小一样吗?实质上,U-net这边做的处理是,根据预先设置的输出图像大小,然后对输入图像进行镜像填充。具体怎么做的呢?
如上图所示,预测的分割区域在黄色区域,蓝色区域作为输入,提供更多的局部信息,但实质上我们是没有那么大的蓝色区域的,所以缺失的数据就以镜像的方式被填充了。这样的操作会带来图像重叠问题,即某一图像的周围可能会和另一张图片重叠。因此作者在卷积时只使用有效部分。 下采样网络左半部分为下采样,一共有四步。 #input 572*572*1 ---(卷积)--->570*570*64(ReLu激活)---(卷积)--->568*568*64(ReLu)激活 下采样 step1:568*568*64---(max pooling)--->284*284*128---(卷积)--->282*282*128(ReLu激活)--- (卷积)--->280*280*128(ReLu激活) 之后的三步与step1类似 上采样以及输出网络右半部分为上采样,同样有四步,与左半部分不同的是,它每次进行上采样采用的都是反卷积(pytorch总的transpose函数即可实现),而且每次都会引入裁剪后高分辨率的图像信息。 step1:28*28*1024---(反卷积)--->56*56*512+左半部分64*64*512---(裁剪合并)=56*56*1024 ---(卷积)--->54*54*512(ReLu激活)---(卷积)--->52*52*512(ReLu激活) 接下来三步与上面一步类似 1*1Conv: 388*388*64--->388*388*2 将前景和背景分割出来,变成一个二分类问题 U-net的特点 U-net是一个完全的卷积层,所以输入和输出图像的大小可以是任意的分为编码器和解码器,编码器将图片不断压缩,分辨率变得越来越低,解码器将图像分辨率不断还原,分辨率逐步升高解码器在解码过程中会用到编码器提取的特征 谷歌Colab复现基于U-Net的眼底图像血管分割在学习过U-net的相关资料后,我准备通过学习代码来加深对U-net的理解,同时记录学习过程中遇到的坑,正好之前浏览博文的时候有过相关收藏,具体链接:基于U-Net的眼底图像血管分割实例。 1.环境配置查看是否有相关的库,具体如下:
由于这个代码已经比较久了,所以里面有些环境并不是很适配,所以在复现的时候遇到了不少坑。笔者在实验过程中发现colab有上面所有的库,只不过需要做些改动。 #首先 colab中的h5py版本是比较高的,到时候导入模型的时候会报解码错误 所以,我们需要执行指令 !pip uninstall h5py !pip install h5py==2.7.1 #其次,colab的tensorflow默认是2.x版本,而这边是1.x版本,所以,我们需要进行tensorflow版本切换 %tensorflow_version 可以查看当前的tf环境,以及可用的版本 %tensorflow_version 1.x 切换到1.x环境 train文件中,导入的Adam应该改为adam_v2 导入的sgd语句变为from tensorflow.keras.optimizers import SGD
使用的数据集为DRIVE数据集,训练图片为40张,测试图片为20张。这个数据集相当小,所以我们需要对其进行数据增强,以得到更好的训练效果。
图一 眼底图
图三 眼球轮廓图 3.代码框架 3.1 数据预处理: #对比度受限的直方图均衡化,用于增强图像的对比度 def clahe_equalized(imgs): assert (len(imgs.shape)==4) #4D arrays assert (imgs.shape[1]==1) #check the channel is 1 clahe = cv2.createCLAHE(clipLimit=2.0, tileGridSize=(8,8)) #限制在2 imgs_equalized = np.empty(imgs.shape) # 创建一个和图像矩阵大小一样的矩阵 for i in range(imgs.shape[0]): imgs_equalized[i,0] = clahe.apply(np.array(imgs[i,0], dtype = np.uint8)) return imgs_equalized #伽马变换用来图像增强,其提升了暗部细节,简单来说就是通过非线性变换 #让图像从暴光强度的线性响应变得更接近人眼感受的响应,即将漂白(相机曝光)或过暗(曝光不足)的图片,进行矫正。 def adjust_gamma(imgs, gamma=1.0): assert (len(imgs.shape)==4) #4D arrays assert (imgs.shape[1]==1) #check the channel is 1 invGamma = 1.0 / gamma table = np.array([((i / 255.0) ** invGamma) * 255 for i in np.arange(0, 256)]).astype("uint8") new_imgs = np.empty(imgs.shape) for i in range(imgs.shape[0]): new_imgs[i,0] = cv2.LUT(np.array(imgs[i,0], dtype = np.uint8), table) return new_imgs 3.2 数据增强: def extract_random(full_imgs,full_masks, patch_h,patch_w, N_patches, inside=True): if (N_patches%full_imgs.shape[0] != 0): print("N_patches: please enter a multiple of 20") exit() # 进行断言判断,满足这些条件才会继续进行下去 assert (len(full_imgs.shape)==4 and len(full_masks.shape)==4) #4D arrays assert (full_imgs.shape[1]==1 or full_imgs.shape[1]==3) #check the channel is 1 or 3 assert (full_masks.shape[1]==1) #masks only black and white assert (full_imgs.shape[2] == full_masks.shape[2] and full_imgs.shape[3] == full_masks.shape[3]) patches = np.empty((N_patches,full_imgs.shape[1],patch_h,patch_w)) patches_masks = np.empty((N_patches,full_masks.shape[1],patch_h,patch_w)) img_h = full_imgs.shape[2] #height of the full image img_w = full_imgs.shape[3] #width of the full image # (0,0) in the center of the image patch_per_img = int(N_patches/full_imgs.shape[0]) #N_patches equally divided in the full images print("patches per full image: " +str(patch_per_img)) iter_tot = 0 #iter over the total numbe rof patches (N_patches) for i in range(full_imgs.shape[0]): #loop over the full images k=0 while k |
【本文地址】


 图二 血管图
图二 血管图