第二章 序列比对 |
您所在的位置:网站首页 › blast怎么比对两个序列 › 第二章 序列比对 |
第二章 序列比对
|
阅读量: 330
主要为基因组测序比对相关知识,部分内容作笔记自查使用。如有错误或遗漏还请海涵,可评论或邮箱联系。最后修改时间:2020-04-16 16:18:55 星期四 Blast局部比对流程
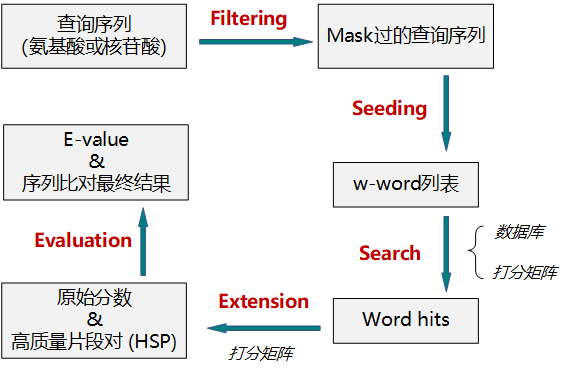
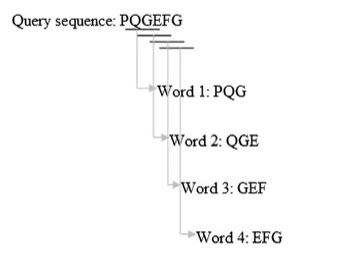
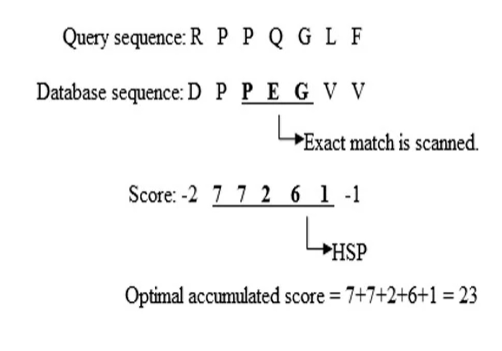
【目的】 防止由于低复杂度和重复片段存在,而产生大量有统计学意义,但无生物学实际意义的比对结果 低复杂度和重复片段: {\left( {CA} \right)_n}(CA)nKLKLKLKLKLKL【方法】 将查询序列中极低复杂度的片段mask掉 mask掩码 核酸残基用{N_s}Ns氨基酸残基用{X_s}Xs【程序参数】-F:开启过滤 二、Seeding【目的】 鸽子洞思想。将查询序列切片成一个一个的w-mers以便快速查找,其中w为切片长度 【方法】 【程序参数】-W 数字:设定切片长度w 三、Search【目的】 拿seeding产生list中的每一个word,去数据库中做匹配。保留匹配值大于设定阈值T的匹配点位置 【方法】 1、 哈希表法:给word设定键值,通过键值直接寻址找匹配 2、 有限自动机法:程序确定word里每一个字符的转换状态,然后去数据库中搜索 四、Extending【目的】 将Search步骤找到的匹配点向前向后延伸,使之成为一个HSP。在扩展后的区域,应用Smith-Waterman动态规划算法,以便确定最终的比对,从而显著降低了计算量 【方法】 设定cutoff值S。如果延伸过程中,比对的打分低于S,我们就停止。下例设定S=0 【目的】 评估找到的比对结果的显著性。如果显著性水平高,说明该比对并不是因为随机原因而比对成功的 【方法】E = kmne{}^{ - \lambda S}E=kmne−λS m:查询序列长度 n:数据库长度 S:HSP的原始得分 通常E蛋白blastx核酸序列转换成蛋白质序列搜索tblastx核酸序列转换成蛋白质序列,再将核算数据库转换成蛋白质数据库搜索蛋白->核酸tblastn蛋白质序列转换成核酸序列搜索 |
【本文地址】
今日新闻 |
推荐新闻 |