生物信息学分析 |
您所在的位置:网站首页 › blast两个序列比对怎么分析 › 生物信息学分析 |
生物信息学分析
|
最全的Linux教程,Linux从入门到精通 ====================== linux从入门到精通(第2版) Linux系统移植 Linux驱动开发入门与实战 LINUX 系统移植 第2版 Linux开源网络全栈详解 从DPDK到OpenFlow
第一份《Linux从入门到精通》466页 ==================== 内容简介 ==== 本书是获得了很多读者好评的Linux经典畅销书**《Linux从入门到精通》的第2版**。本书第1版出版后曾经多次印刷,并被51CTO读书频道评为“最受读者喜爱的原创IT技术图书奖”。本书第﹖版以最新的Ubuntu 12.04为版本,循序渐进地向读者介绍了Linux 的基础应用、系统管理、网络应用、娱乐和办公、程序开发、服务器配置、系统安全等。本书附带1张光盘,内容为本书配套多媒体教学视频。另外,本书还为读者提供了大量的Linux学习资料和Ubuntu安装镜像文件,供读者免费下载。
本书适合广大Linux初中级用户、开源软件爱好者和大专院校的学生阅读,同时也非常适合准备从事Linux平台开发的各类人员。 需要《Linux入门到精通》、《linux系统移植》、《Linux驱动开发入门实战》、《Linux开源网络全栈》电子书籍及教程的工程师朋友们劳烦您转发+评论 网上学习资料一大堆,但如果学到的知识不成体系,遇到问题时只是浅尝辄止,不再深入研究,那么很难做到真正的技术提升。 需要这份系统化的资料的朋友,可以点击这里获取! 一个人可以走的很快,但一群人才能走的更远!不论你是正从事IT行业的老鸟或是对IT行业感兴趣的新人,都欢迎加入我们的的圈子(技术交流、学习资源、职场吐槽、大厂内推、面试辅导),让我们一起学习成长! ###进入解压目录 cd ncbi-blast-2.14.1+ ###查看解压目录内的文件,感兴趣的就读一下README吧。 ls [root@vmgmt ncbi-blast-2.14.1+]# ls bin BLAST_PRIVACY ChangeLog doc LICENSE ncbi_package_info README ###可直接使用的文件在bin目录下 cd bin ###查看目录下的文件 ls [root@vmgmt bin]# ls blastdb_aliastool blast_formatter blastn_vdb blastx deltablast legacy_blast.pl makeprofiledb rpstblastn tblastn_vdb windowmasker blastdbcheck blast_formatter_vdb blastp cleanup-blastdb-volumes.py dustmasker makeblastdb psiblast segmasker tblastx blastdbcmd blastn blast_vdb_cmd convert2blastmask get_species_taxids.sh makembindex rpsblast tblastn update_blastdb.pl ###上面目录的文件默认已经可以直接执行,未配置未系统目录时需要加上指定命令的路径 #如: ./blastn -help [root@vmgmt bin]# ./blastn -help USAGE blastn [-h] [-help] [-import_search_strategy filename] [-export_search_strategy filename] [-task task_name] [-db database_name] [-dbsize num_letters] [-gilist filename] [-seqidlist filename] [-negative_gilist filename] [-negative_seqidlist filename] [-taxids taxids] [-negative_taxids taxids] [-taxidlist filename] [-negative_taxidlist filename] [-entrez_query entrez_query] [-db_soft_mask filtering_algorithm] [-db_hard_mask filtering_algorithm] [-subject subject_input_file] [-subject_loc range] [-query input_file] [-out output_file] [-evalue evalue] [-word_size int_value] [-gapopen open_penalty] [-gapextend extend_penalty] [-perc_identity float_value] [-qcov_hsp_perc float_value] [-max_hsps int_value] [-xdrop_ungap float_value] [-xdrop_gap float_value] [-xdrop_gap_final float_value] [-searchsp int_value] [-penalty penalty] [-reward reward] [-no_greedy] [-min_raw_gapped_score int_value] [-template_type type] [-template_length int_value] [-dust DUST_options] [-filtering_db filtering_database] [-window_masker_taxid window_masker_taxid] [-window_masker_db window_masker_db] [-soft_masking soft_masking] [-ungapped] [-culling_limit int_value] [-best_hit_overhang float_value] [-best_hit_score_edge float_value] [-subject_besthit] [-window_size int_value] [-off_diagonal_range int_value] [-use_index boolean] [-index_name string] [-lcase_masking] [-query_loc range] [-strand strand] [-parse_deflines] [-outfmt format] [-show_gis] [-num_descriptions int_value] [-num_alignments int_value] [-line_length line_length] [-html] [-sorthits sort_hits] [-sorthsps sort_hsps] [-max_target_seqs num_sequences] [-num_threads int_value] [-mt_mode int_value] [-remote] [-version] DESCRIPTION Nucleotide-Nucleotide BLAST 2.14.1+ Use ‘-help’ to print detailed descriptions of command line arguments###如果想直接使用的话注意将本目录加入到profile环境中去(推荐,),或者将本目录下的所有命令复制到/usr/bin下(不建议) vim /etc/profile.d/blast.sh ##前面的/opt根目录请根据自己的实际路径修改 export PATH=“/opt/ncbi-blast-2.14.1+/bin”:$PATH ###配置环境路径生效 source /etc/profile.d/blast.sh #或 source /etc/profile #直接运行命令测试 blastn -h ### 使用方法###基于基于核酸序列-参考核酸序列的比对功能 blastn -db database -query input.fasta -out output -outfmt 6 db 后面接比对的数据库, query后面接核酸序列,out接想要输出的文件名,outfmt输出格式,6表示表格输出,如下:-outfmt alignment view options: 0 = pairwise, 1 = query-anchored showing identities, 2 = query-anchored no identities, 3 = flat query-anchored, show identities, 4 = flat query-anchored, no identities, 5 = XML Blast output, 6 = tabular, 7 = tabular with comment lines, 8 = Text ASN.1, 9 = Binary ASN.1 10 = Comma-separated values ###这个序列比对速度有点慢,建议多核跑加上threads参数, outfmt是大家通用的表格格式,使用6,7都不错。 ###输出后看表格头部,大致字段表示如下: outfmt,格式6输出(无表头,适合文件合并拼接处理)结果如下,没有表头,需要表头说明的采用7输出,从第一列到最后一列解释分别如下: 1、Query id:查询序列ID标识 2、Subject id:比对上的目标序列ID标识 3、% identity:序列比对的一致性百分比 4、alignment length:符合比对的比对区域的长度 5、mismatches:比对区域的错配数 6、gap openings:比对区域的gap数目 7、q. start:比对区域在查询序列(Query id)上的起始位点 8、q. end:比对区域在查询序列(Query id)上的终止位点 9、s. start:比对区域在目标序列(Subject id)上的起始位点 10、s. end:比对区域在目标序列(Subject id)上的终止位点 11、e-value:比对结果的期望值 12、bit score:比对结果的bit score值 一般情况我们看第3、11、12两列,e值越小越可靠 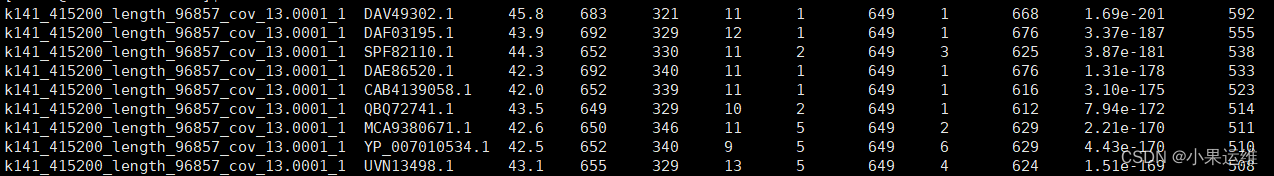 **网上学习资料一大堆,但如果学到的知识不成体系,遇到问题时只是浅尝辄止,不再深入研究,那么很难做到真正的技术提升。** **[需要这份系统化的资料的朋友,可以点击这里获取!](https://bbs.csdn.net/forums/4f45ff00ff254613a03fab5e56a57acb)** **一个人可以走的很快,但一群人才能走的更远!不论你是正从事IT行业的老鸟或是对IT行业感兴趣的新人,都欢迎加入我们的的圈子(技术交流、学习资源、职场吐槽、大厂内推、面试辅导),让我们一起学习成长!** |
【本文地址】
今日新闻 |
推荐新闻 |