基迪奥云平台新上线7个功能注释工具! |
您所在的位置:网站首页 › Nr数据库注释结果分析 › 基迪奥云平台新上线7个功能注释工具! |
基迪奥云平台新上线7个功能注释工具!
|
使用便捷,只需要上传fasta格式的核酸序列或蛋白序列,点击提交,即可进行数据库注释,得到注释总表以及统计图表;使用便宜,仅需要3000奥币/工具,根据奥币与人民币汇率,即只需要150元!比市场上的数据库注释服务收费便宜多了!从此以后自己来分析转录组或其他组学的数据就更简单便捷啦!
今天我们先推出7个基本注释工具:Nr注释、GO功能注释、KEGG功能注释、Swiss-Prot注释、COG注释、KOG注释、Pfam注释。 温馨提醒: 如果你使用了注释工具,扣除了奥币,然而没有得到注释结果,别慌,基迪奥omicshare免费售后。 立即联系OS客服或者发送邮件到[email protected]。 Nr数据库注释 Nr数据库是NCBI的非冗余蛋白序列数据库,是所有测序流程里默认的蛋白比对数据库。对于所有已知的或可能的编码序列,Nr记录中都给出了相应的氨基酸序列(通过已知或可能的读码框推断而来)以及专门蛋白数据库中的序列号和描述信息。 利用基迪奥omicshare云平台的Nr数据库注释工具,用户可以上传至少100 M(根据omicshare会员等级)的核酸或蛋白序列fasta文件,然后选择要比对的物种,点击提交即可。输出结果有序列注释总表、注释结果统计表和统计饼图、E值分布统计表和统计饼图。 1 序列注释总表
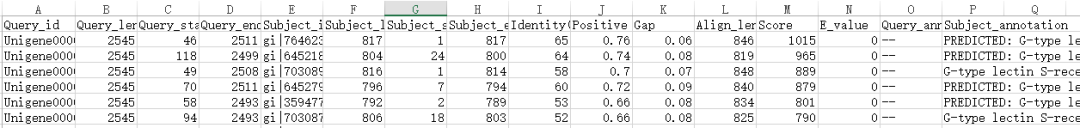
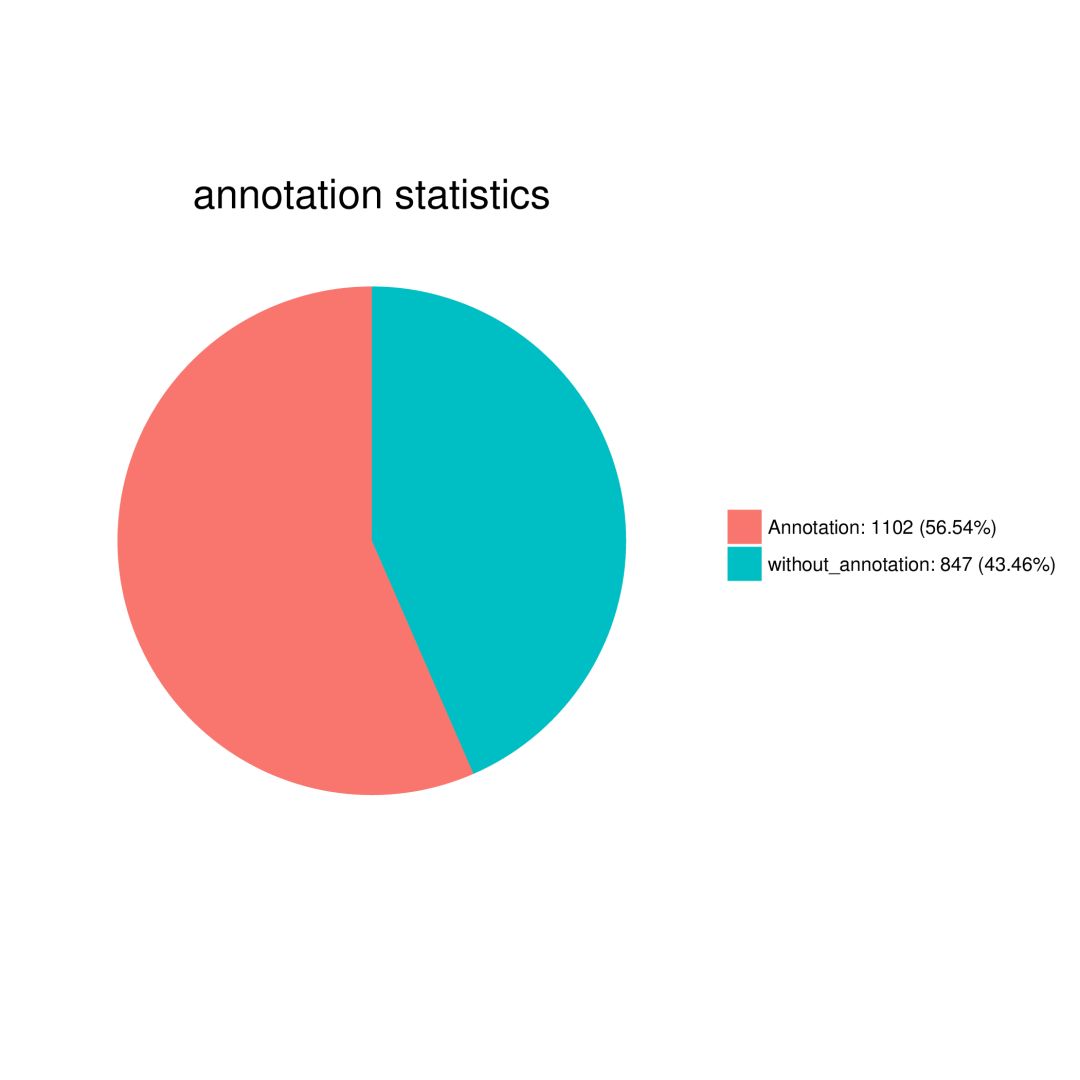
序列注释总表给出了每条序列的Nr比对结果,包括比对上的序列ID、比对位置、比对长度、相似性(identity)、比对E值、比对分数、序列描述信息等。 2 注释结果统计表和统计饼图 统计注释上和没有注释上的序列数目,并画饼图直观展示。
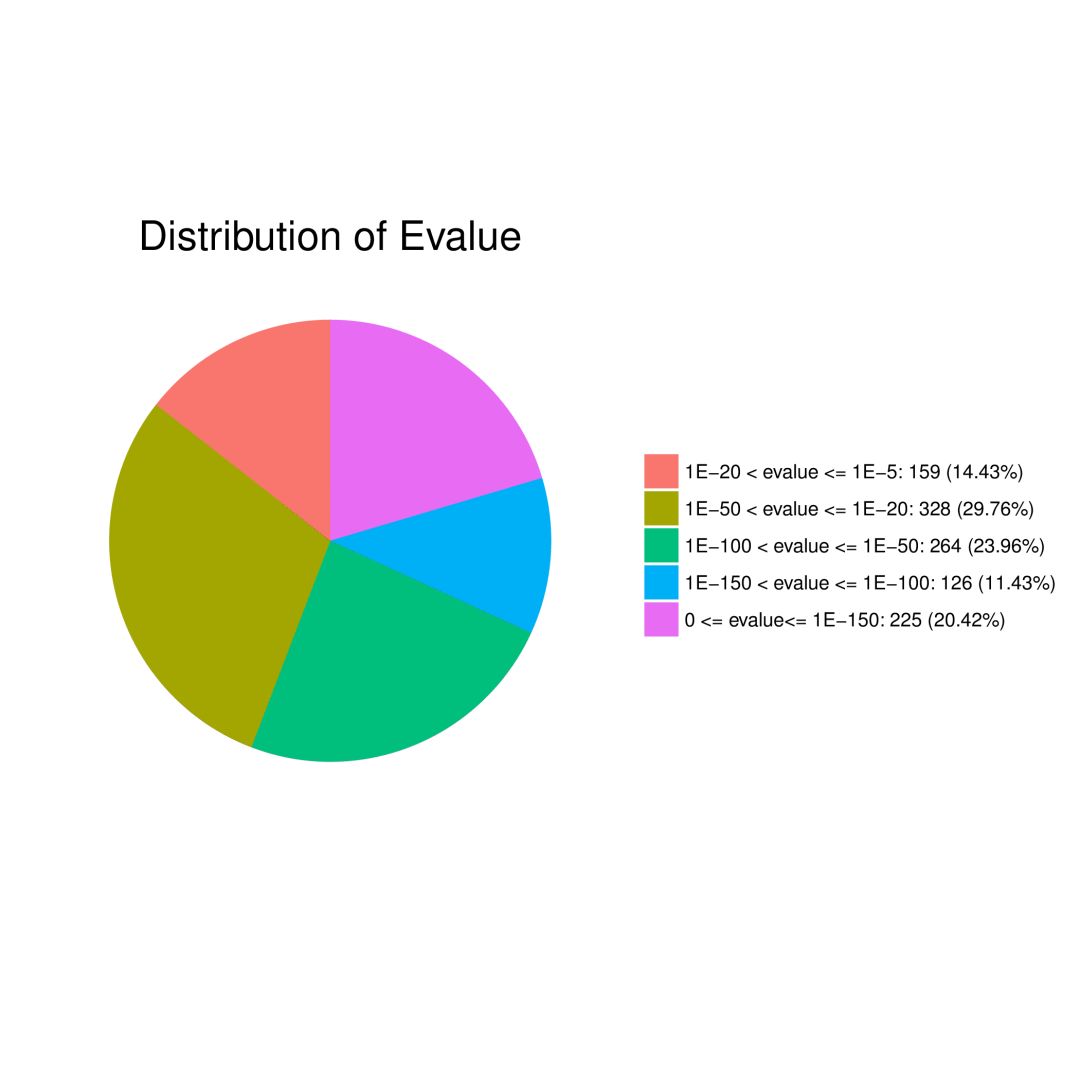
1 E值分布统计表和统计饼图 E值是指期望数据库中具有某一统计学意义配对序列的值,可理解为比对的假阳性率。E值越小,结果越可靠。我们对E值分为5个范围进行统计,并画饼图。
Swiss-Prot数据库注释 Swiss-Prot是另一个著名的蛋白数据库,是经过注释和验证的严格去冗余的蛋白质序列数据库,隶属于UniProt数据库,到2018年底共收录558,898条序列信息。Swiss-Prot可提供蛋白质序列的详尽注释信息,包括蛋白质功能、蛋白质翻译后修饰、结构域和结合位点、二级结构、四级结构、蛋白质缺陷相关疾病等信息。 Swiss-Prot也是测序流程里一般都会有的注释数据库。omicshare云平台的Swiss-Prot数据库注释工具,输出结果也是序列注释总表、注释结果统计表和统计饼图、E值分布统计表和统计饼图。 GO数据库注释 GO数据库(Gene Ontology)是大家非常熟悉的数据库,是研究基因功能的最常用注释数据库。所有文章中在得到测序数据结果后,都会对基因或蛋白进行GO和KEGG功能分析的,所以这个GO数据库注释工具也是非常必要的。 omicshare云平台的GO数据库注释工具,输出结果有基因GO注释统计表、注释结果统计表和统计饼图、GO功能分类统计表和GO二级注释柱状图。GO功能分类统计表给出了每个GO条目下所包含的基因数目及基因ID。GO二级注释柱状图展示了每个二级注释GO 条目下所包含的基因数量。
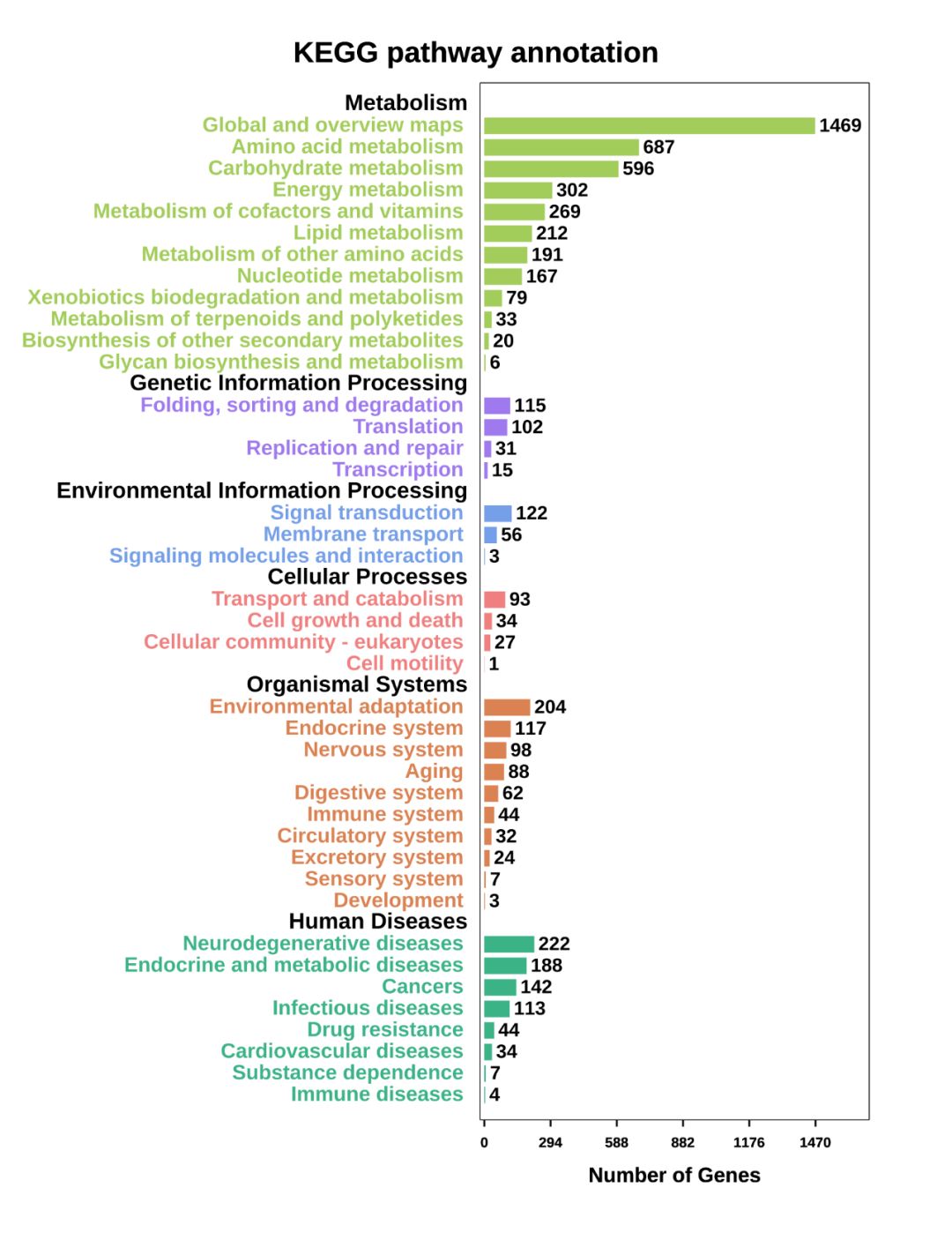
KEGG数据库注释 KEGG数据库也是大家非常熟悉的了,是研究基因在细胞中的代谢通路途径的最常用数据库。所有文章中在得到测序数据结果后,都会对基因或蛋白进行GO和KEGG功能分析的,所以这个KEGG数据库注释工具也是非常必要的。 omicshare云平台的KEGG数据库注释工具,输出结果除了上述三个,还有Pathway注释信息统计表和KEGG注释条形图。Pathway注释信息统计表给出了每条pathway的KEGG ID、所包含的基因数目以及基因ID。KEGG注释条形图给出了KEGG B级分类下每条通路的基因数量。
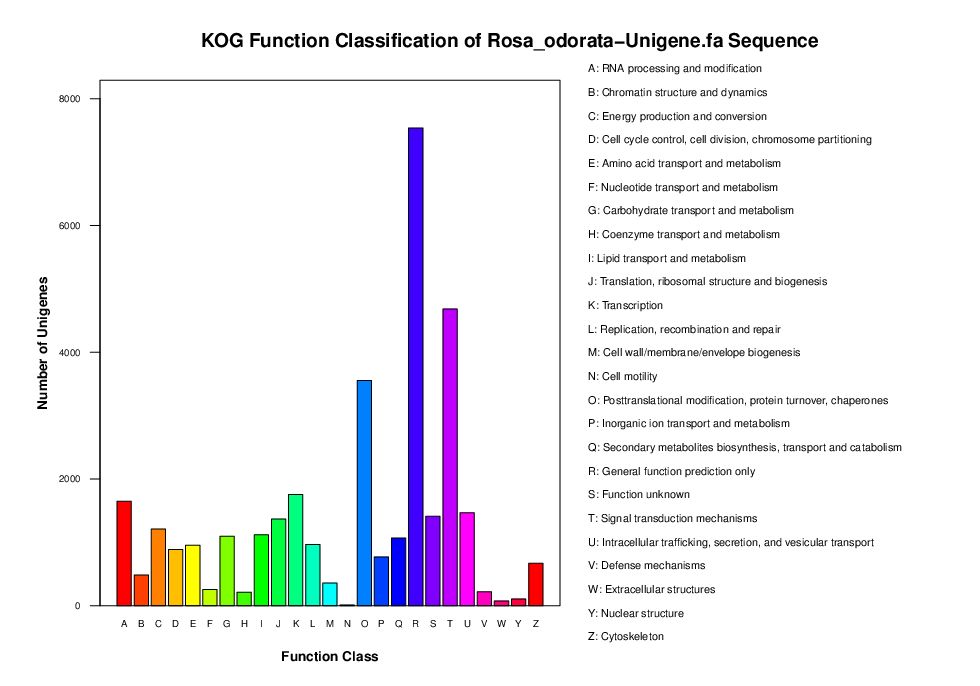
COG/KOG数据库注释 COG蛋白数据库是Cluster of Orthologous Groups of proteins(蛋白相邻类的聚簇)的缩写。构成每个 COG 的蛋白都是被假定为来自于一个祖先蛋白,并且因此或者是 orthologs 或者是 paralogs。 Orthologs 是指来自于不同物种的由垂直家系(物种形成)进化而来的蛋白,并且典型的保留与原始蛋白有相同的功能。 Paralogs是那些在一定物种中的来源于基因复制的蛋白,可能会进化出新的与原来有关的功能。 KOG数据库是COG的真核版本,因此原核生物我们用COG注释,真核生物用KOG注释。COG/KOG数据库包含25大类的功能,每个COG都有一个特定的功能描述。 omicshare云平台的COG/KOG数据库注释工具,输出结果除了上述三个,还有COG/KOG分类统计柱状图,展示了这25大类分别所含的基因数目及基因ID。
Pfam数据库注释 Pfam(Protein families database of alignments and hidden Markov models )数据库基于多重序列比对以及隐马尔可夫模型(HMM)预测的方法,提供了完整准确的蛋白质家族和结构域分类信息,广泛应用于蛋白结构域注释及蛋白家族分析。 omicshare云平台的Pfam 数据库注释工具,使用Pfam_Scan(https://www.ebi.ac.uk/Tools/pfa/pfamscan/)默认参数将目标序列与数据库比对,使用的数据库为2018年9月更新的最新版Pfam 32.0,包含17,929个蛋白家族信息。输出的结果包括注释结果总表和注释结果统计饼图。表格的表头解释在说明文档中都有详细的说明。 不久后,我们将上线另外8个高级注释工具,敬请期待!返回搜狐,查看更多 |
【本文地址】