仙桃学术 |
您所在的位置:网站首页 › 问道手游洛书东海乡12 › 仙桃学术 |
仙桃学术
|
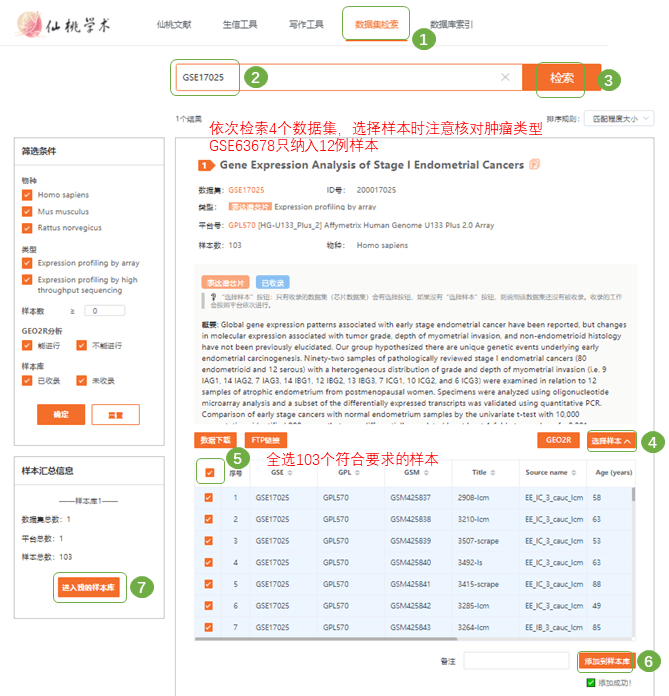
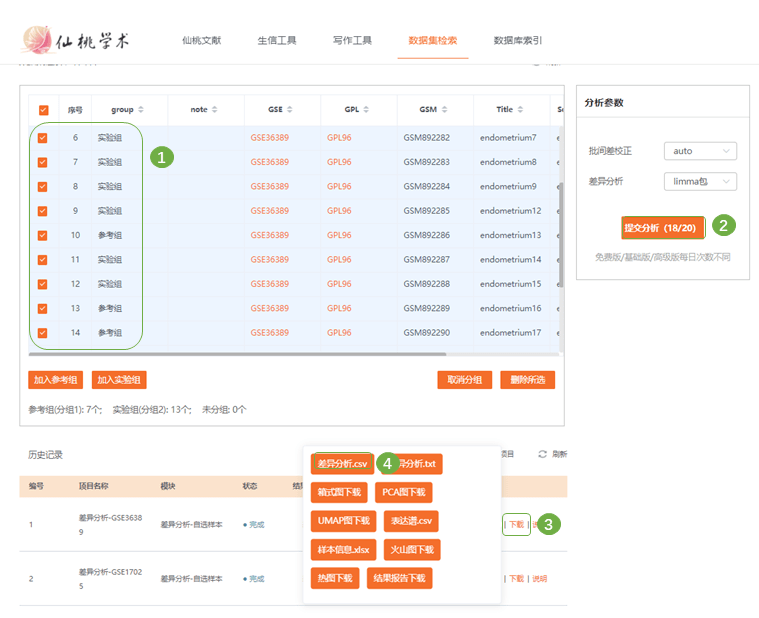
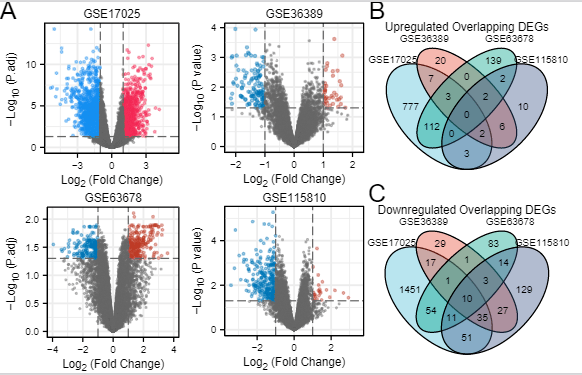
2.使用工具 1)仙桃学术生信工具 https://www.xiantao.love/products 2)Expression Atlas网站 https://www.ebi.ac.uk/gxa/home 3.复现步骤 Fig.1 子宫内膜癌 (EC) 中差异基因鉴定 & Tab.1GEO子宫内膜癌数据集信息 Fig1a为4个GSE各自的差异基因火山图,Fig1b为上调、下调差异基因交集Venn图。进入仙桃数据的“数据集检索”模式,首先检索GSE170,将符合分析目的的103例样本添加至“我的样本库”,进入样本库进行差异分析
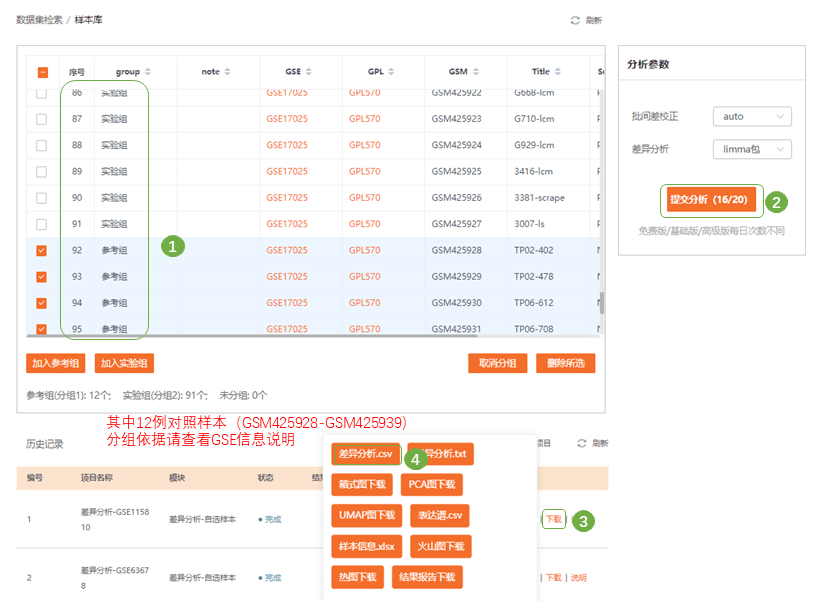
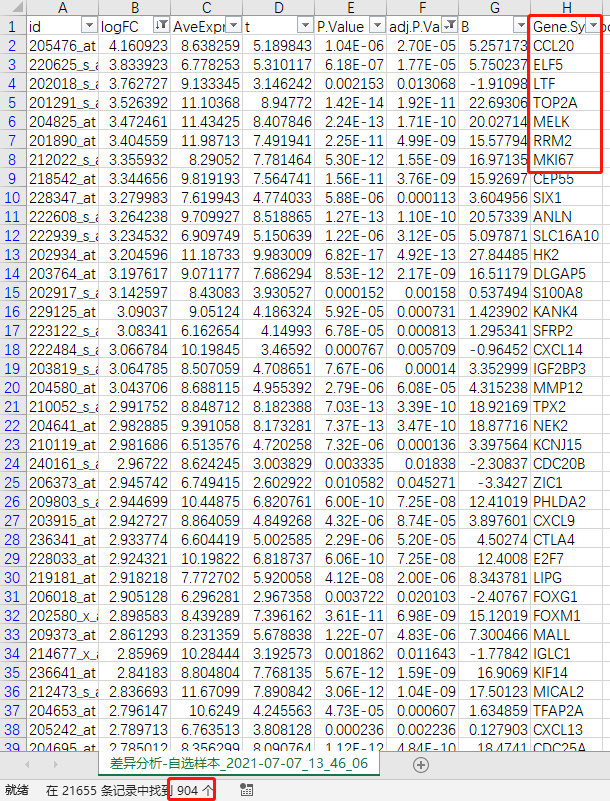
2)查看GEO页面的数据集说明信息,确定分组依据,分别勾选并设定参考组与实验组,提交进行差异分析,分析完成后下载“差异分析.csv”结果表格
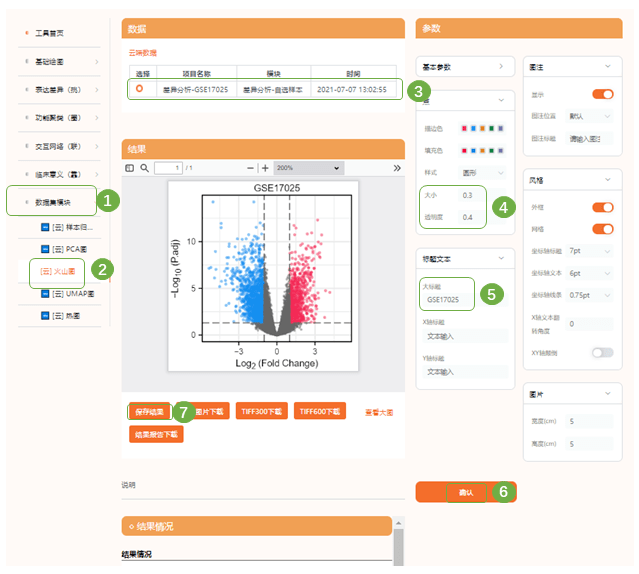
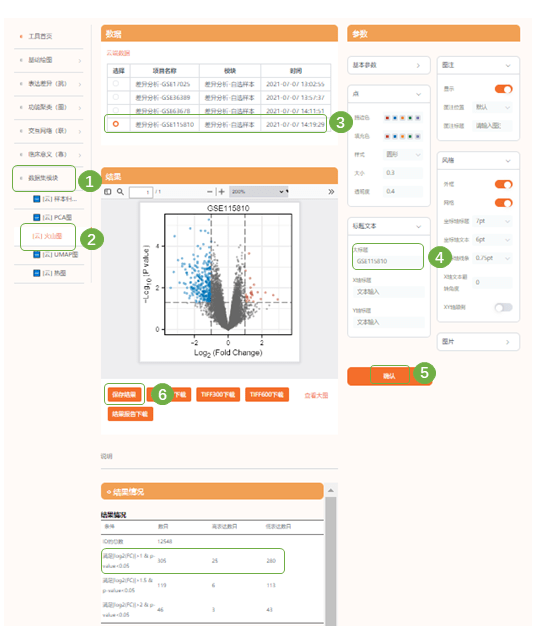
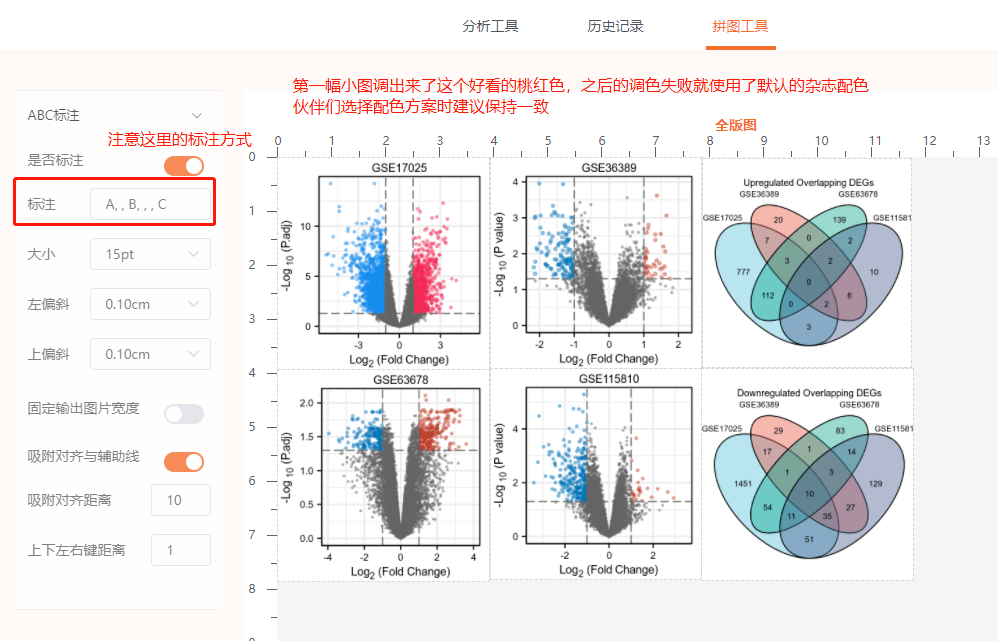
3)接下来进行差异基因可视化——火山图,进入“仙桃学术生信工具——数据集模块——火山图”,已经自动选中了刚才的云端数据(这里说明一下,目前数据集检索模式不支持对不同平台的数据集合并分析,同一平台的数据集是可以合并分析的!),根据图形调整散点大小、透明度等,按数据集名称命名小图;确认提交作图即可
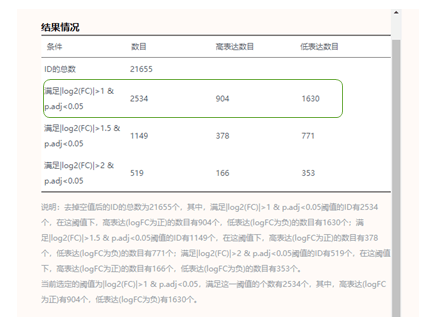
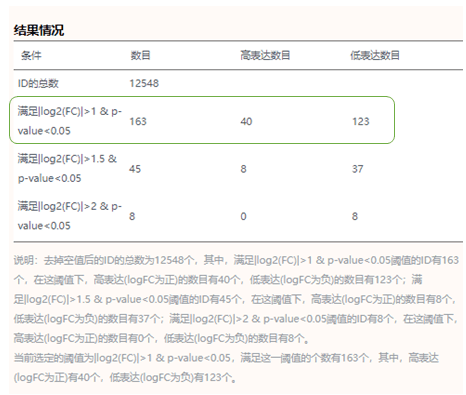
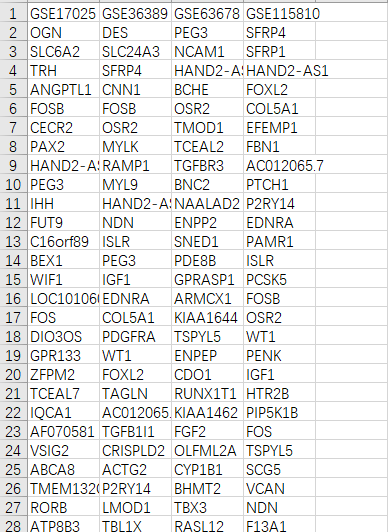
4)查看页面的统计描述结果,接下来我们在最开始下载的差异分析结果表格.csv中提取差异表达的上、下调基因,绘制Venn图
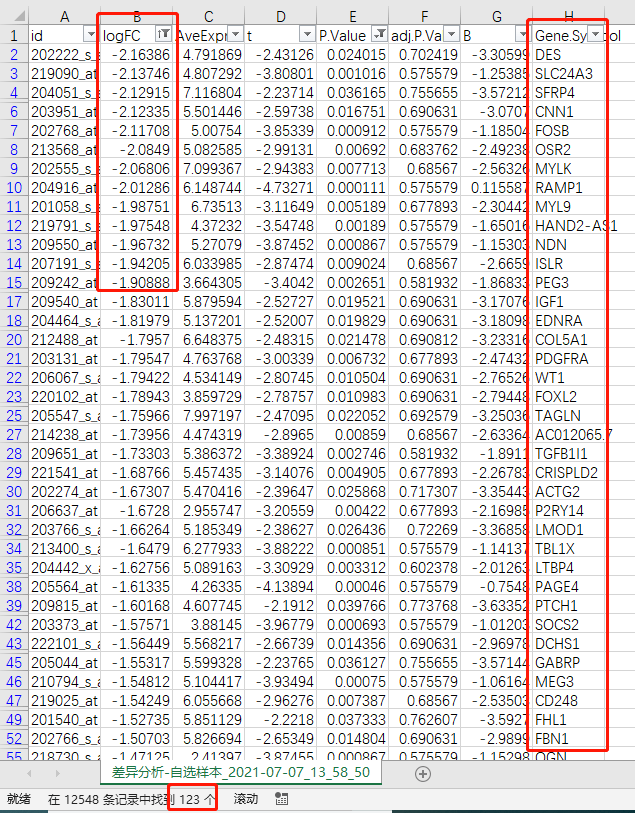
5)使用Excel的筛选功能,按照原文中的阈值adj.P < 0.05 & log|FC| > 1,依次提取logFC > 1 和 logFC < -1的基因
GSE17025 上调DEGs
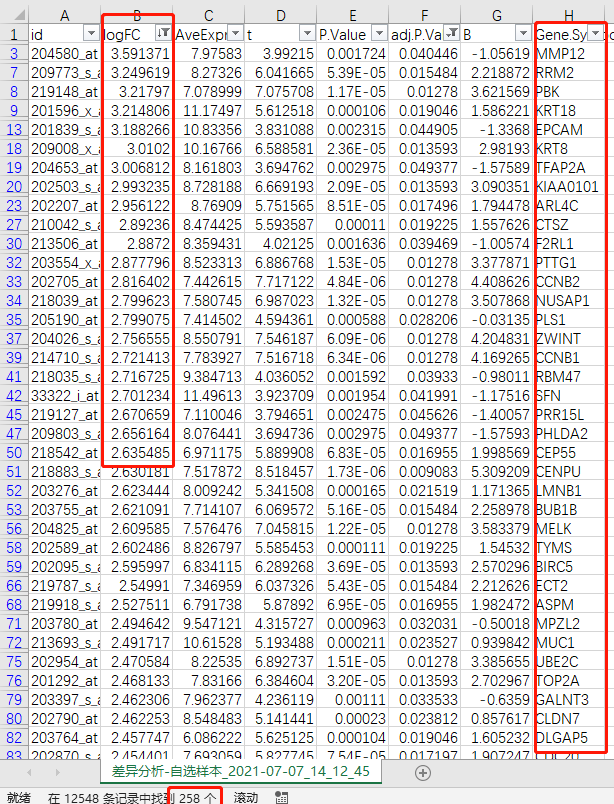
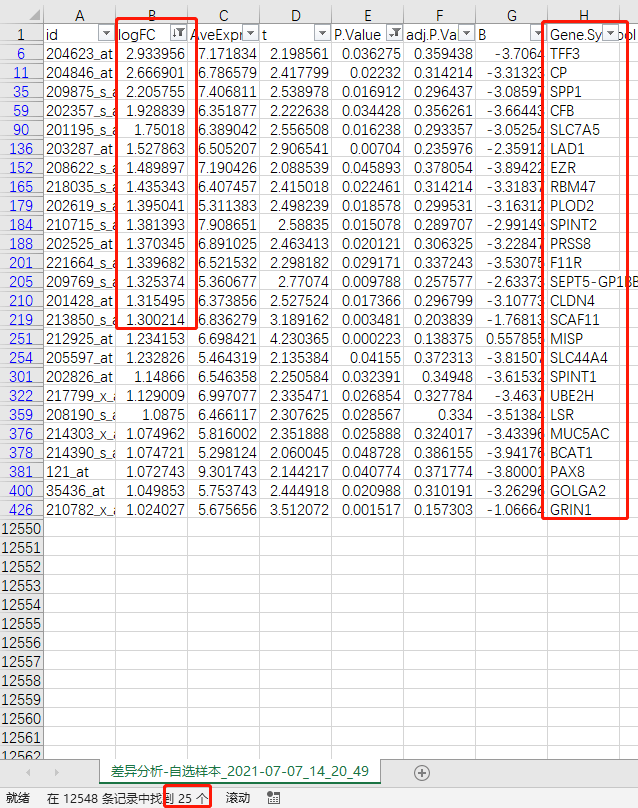
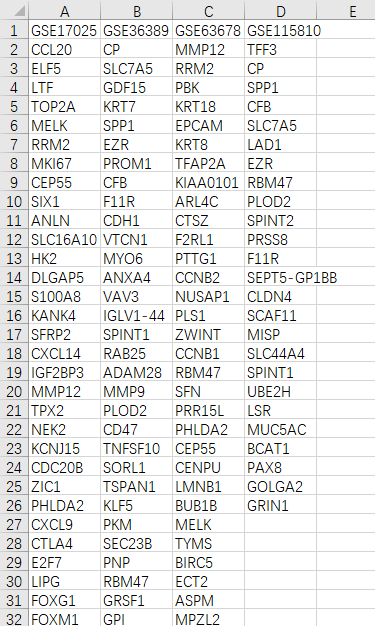
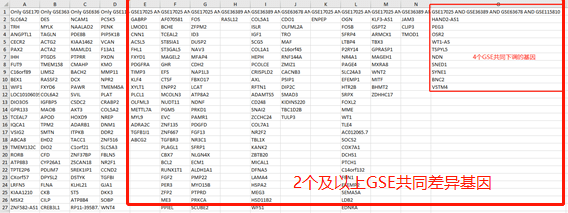
7)差异分析结果表格中同样地提取出各自的上、下调差异表达基因,将4个数据集的DEGs按上调、下调分别整理为1个excel表格
GSE36389 下调DEGs
GSE63678 上调DEGs
整合后的上调DEGs
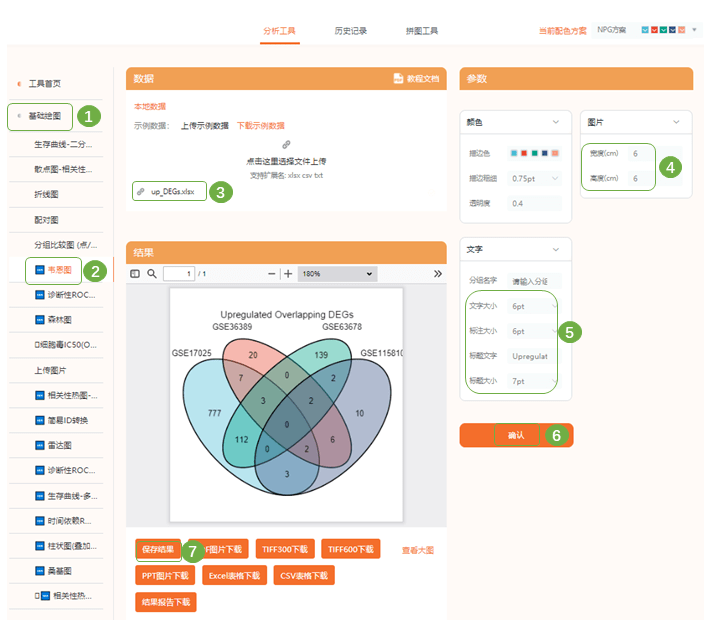
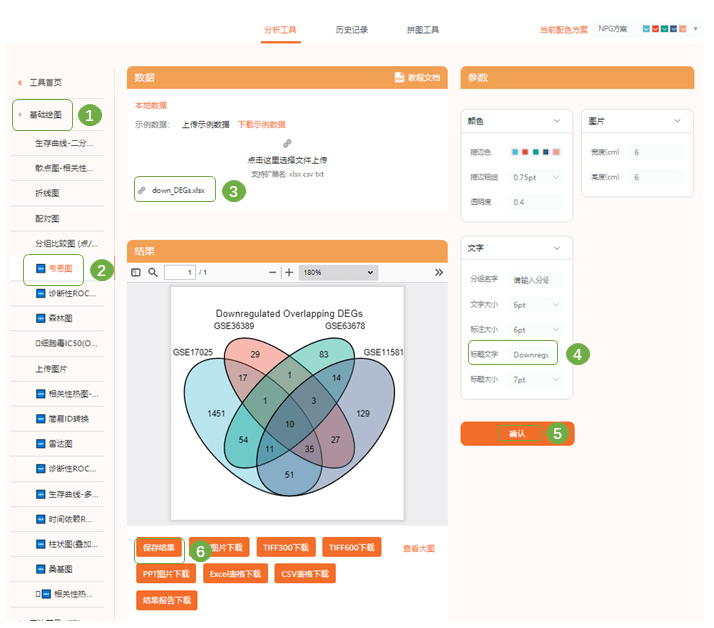
整合后的下调DEGs 8)接下来进入“仙桃学术生信工具—基础绘图—韦恩图“分别绘制4个GSE的上调、下调差异基因交集。可以直接在图片上添加大标题,保存结果
9)进入拼图工具完成拼图,保存图片就得到Fig1啦!注意小图的标注方式~
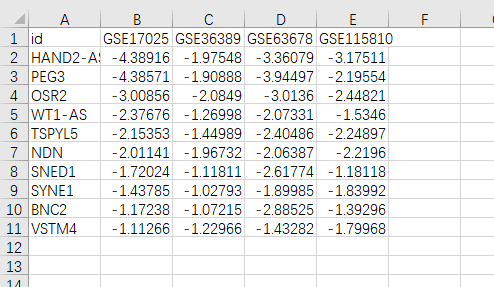
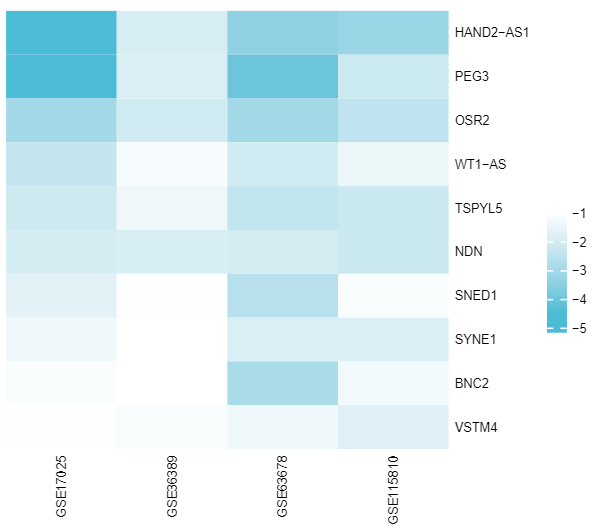
Fig2top20上调、下调DEGs热图 1)Fig1中共获得了28个共同上调基因、94个共同下调基因;作者的差异基因分析使用了GEO2R在线工具,但由于GEO2R不会对探针注释的分子进行去重,因此得到的差异分析结果可能包含不同探针检测到的相同分子,而仙桃学术针对此情况舍弃了重复的分子,故而DEGs数目有所差距,得到的DEGs交集也有出入:没有检测到共同上调基因,仅检测到10个共同下调基因,接下来我们对这10个共同下调基因进行热图分析 2)首先整理数据表格,每个基因的表达值使用“数据集检索“模式分析得到的差异分析结果表格中的log|FC|值即可,整理热图数据如下
复杂热图数据 3)上传数据进行分析,根据原文选择“显示行名与列命“等参数细节,保存结果
Fig3 DEGs GO、KEGG富集分析 1)Fig3中作者对122个DEGs(28个共同上调基因、94个共同下调基因)进行了GO、KEGG富集分析;由于我们的分析得到共同差异基因数据较少,这里选择将2个及以上GSE的共同差异基因列为全部的DEGs,将这些差异整理为富集分析数据格式
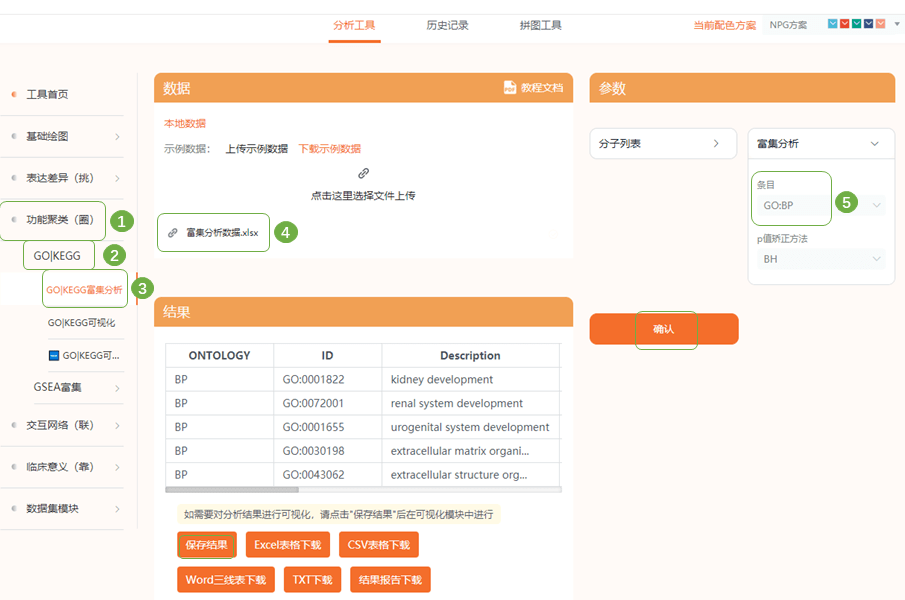
富集分析数据格式 2)进入“仙桃学术生信工具—功能聚类(圈)—GO|KEGG—GO|KEGG富集分析“,上传整理好的数据,依次选择GO:BP, GO:CC, GO:MF, KEGG分析条目,分别提交进行富集分析
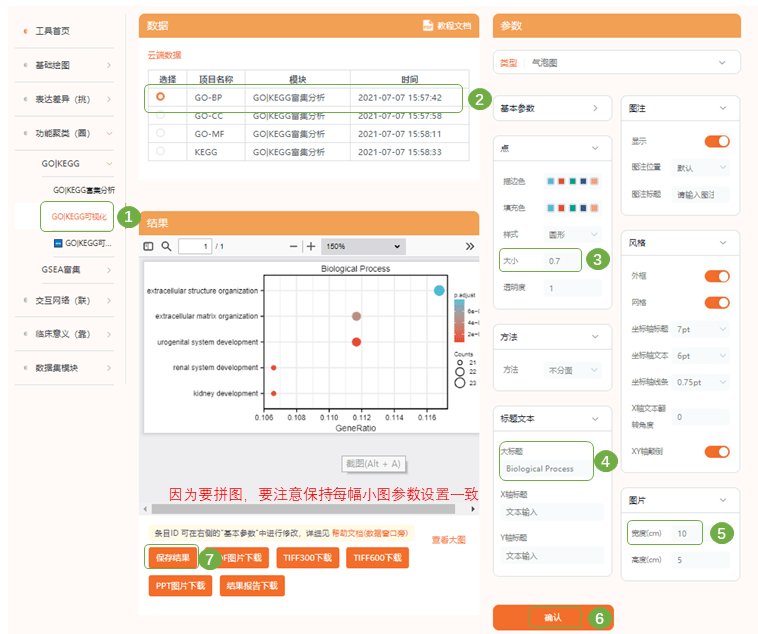
3)进入“GO|KEGG可视化“对刚才的4组数据进行可视化分析,注意每幅小图的参数设置要大体一致
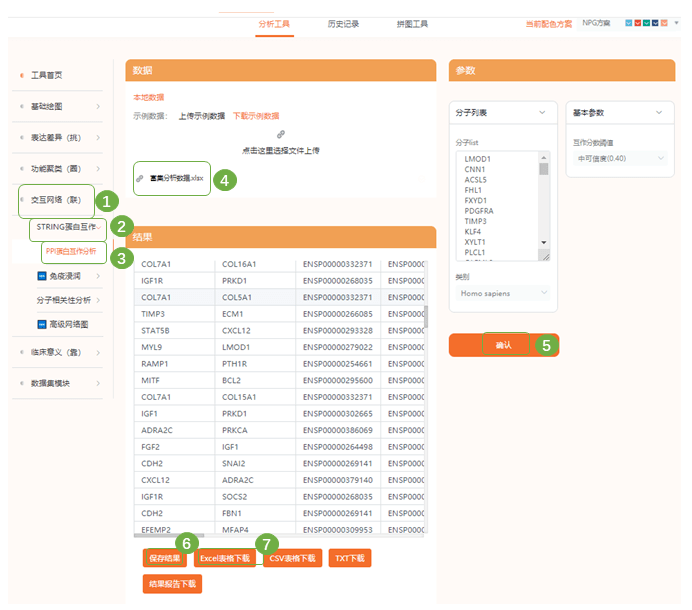
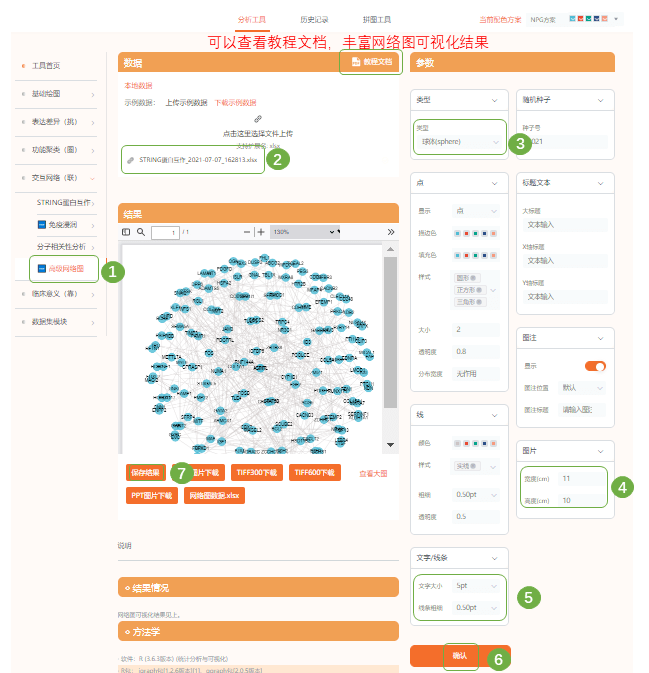
Fig4 DEGs的PPI网络和前10个hub gene的互作网络图 1)接下来作者使用STRING网站对122个DEGs进行了蛋白互作网络分析,进入“交互网路(联)—STRING蛋白互作—PPI蛋白互作分析“,上传富集分析数据,点击确认提交进行分析,保存结果,并下载结果表格
2)进入“高级网络图“,上传刚才下载的结果表的,选择节点呈现方式,台哦正文字大小,提交分析后保存结果
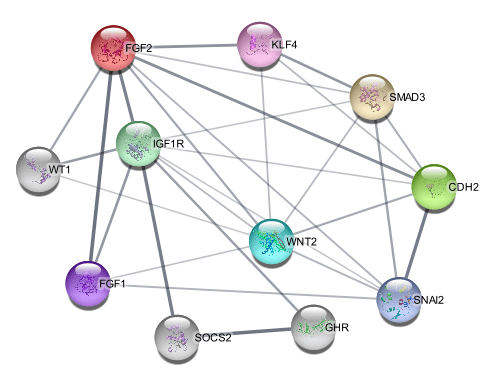
PPI蛋白互作示例数据 此处可以查看教程文档,对可视化网络更好地丰富,还可根据基因表达水平对基因进行色阶差异显示
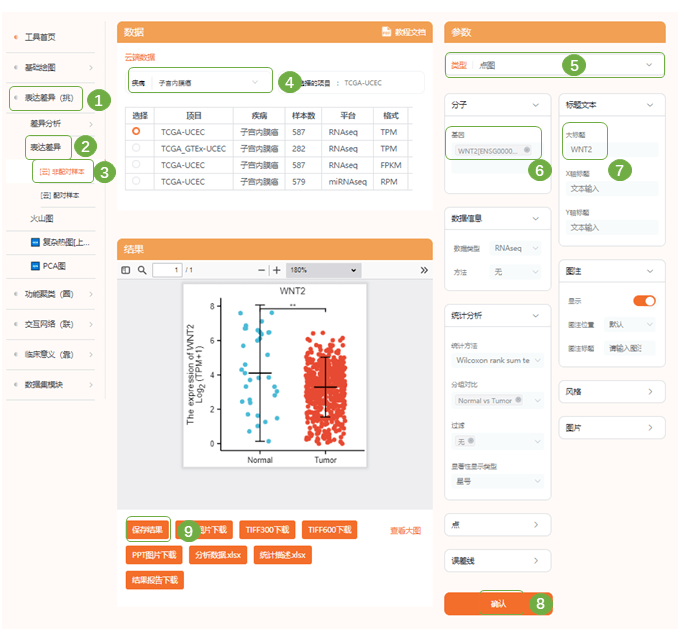
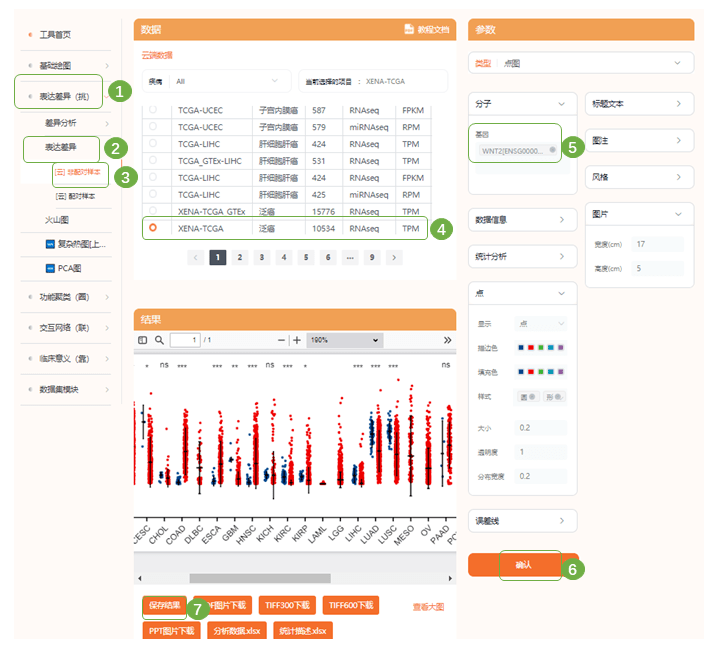
4)可在基础绘图中上传hub gene图,在仙桃学术拼图工具中完成拼图 Fig5 TCGA EC 数据集中9个hub gene表达验证 1)Fig5中作者使用TCGA 子宫内膜癌 (EC) 数据集验证了9个hub gene 的表达水平;进入“表达差异(挑)—表达差异—非配对样本“,选择”子宫内膜癌“,图片类型选择”点图“,依次输入WNT2、IGF1R、SNAJ2、CDH2、SMAD3、FGF2、KLF4、FCF1、SOCS2等9个hub gene进行表达差异分析;保存结果
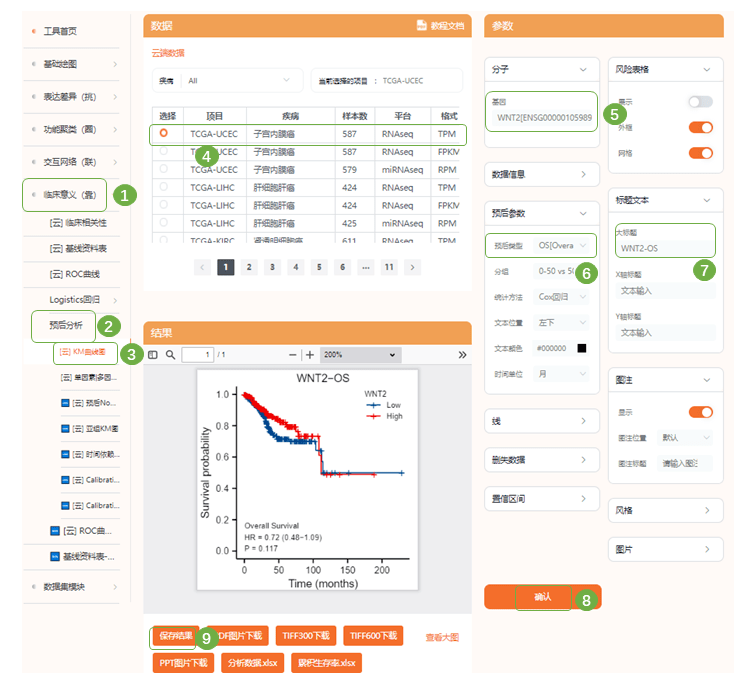
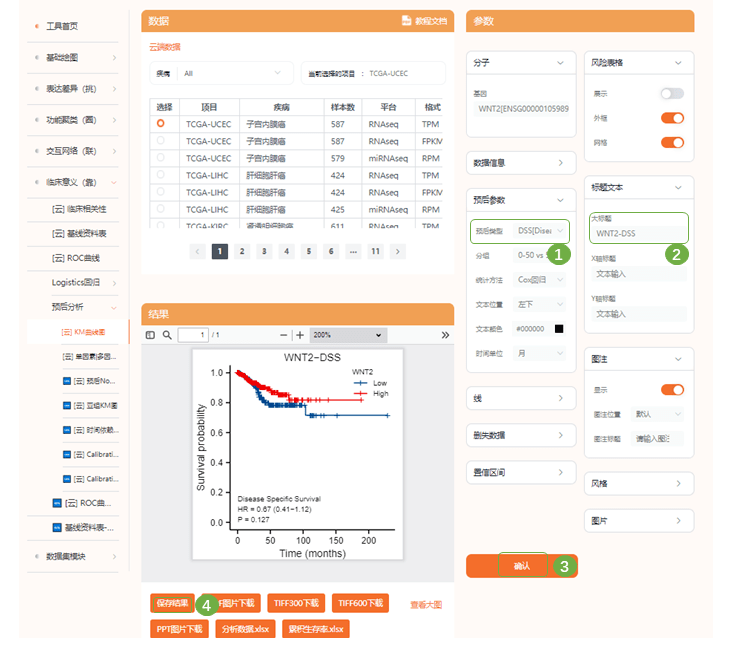
2)在仙桃学术中完成拼图即可 Fig6 EC患者3个hub gene的预后特征 1)接下来作者选择了在EC中低表达的3个基因:FOXL2、NR2F2、TCF4进行了总体存活时间(OS)、无病生存时间(DFS)的GEO、TCGA数据集预后分析;进入“临床意义(靠)—预后分析—KM曲线图“,选择TCGA子宫内膜癌数据集,依次输入目的基因,在预后指标中可选择OS (Overall Survival)、DSS (Disease Specific Survival)、PFI (Progress Free Interval)等多个指标,命名每幅小图的大标题,提交进行分析。保存结果
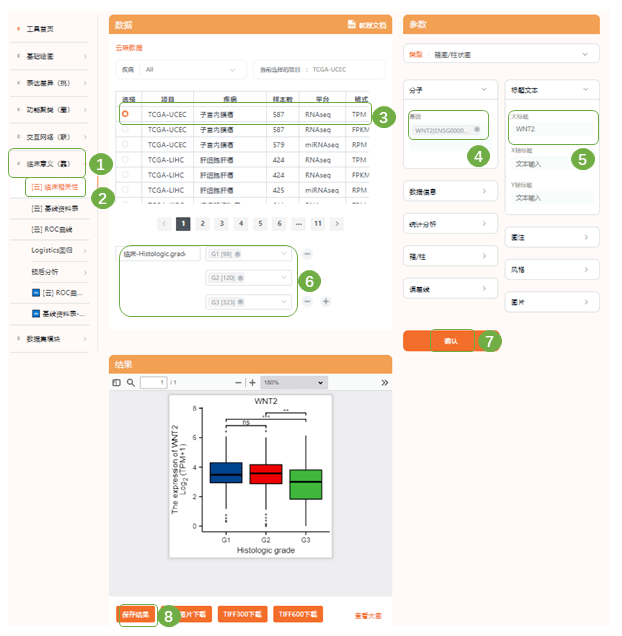
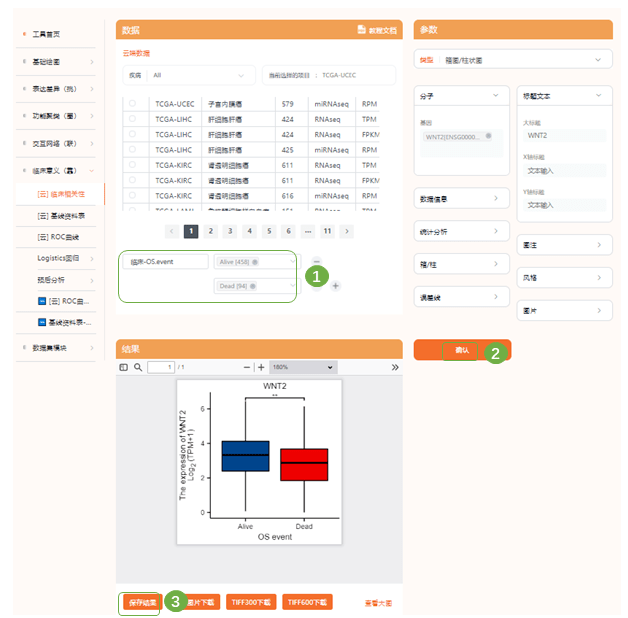
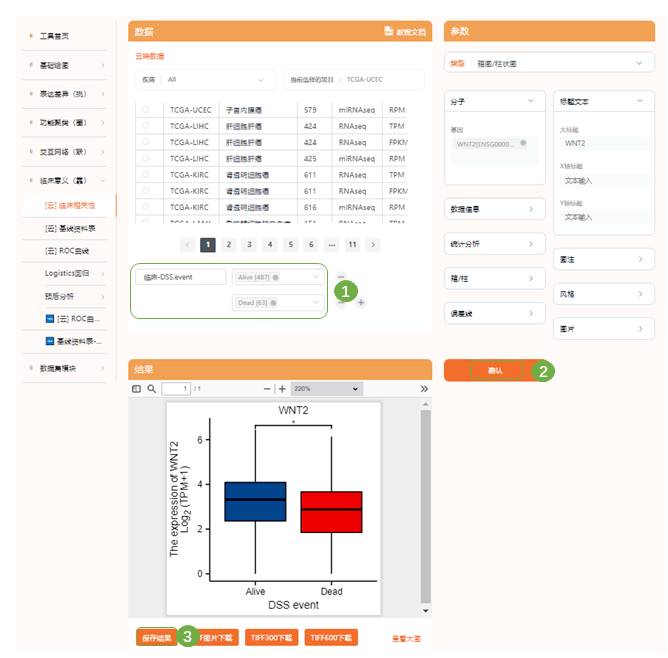
2)同样的,在仙桃学术中完成拼图即可 Fig7 TCGA数据集中FOXL2、NR2F2 和 TCF4 与肿瘤级别、复发和生物标志物、OS 和 DFS关联 1)接下来作者分别分析了3个hub gene表达水平与多种临床病理因素的相关性;进入“临床意义(靠)—临床相关性“,选择TCGA EC数据集,输入分析基因,依次选择不同的病理因素,此处仙桃生信工具可分析的病理因素有Status, clinical stage, primary therapy outcome, race, age, weight, height, BMI, histological type, residual tumor, tumor invasion, menopause status, hormones therapy, diabetes, radiation therapy, surgical approach, OS event, PFI event等20余种指标,依次选择原文中的grade、OS、DFS等,子宫内膜癌尚未收录”肿瘤复发情况“这一病理因素
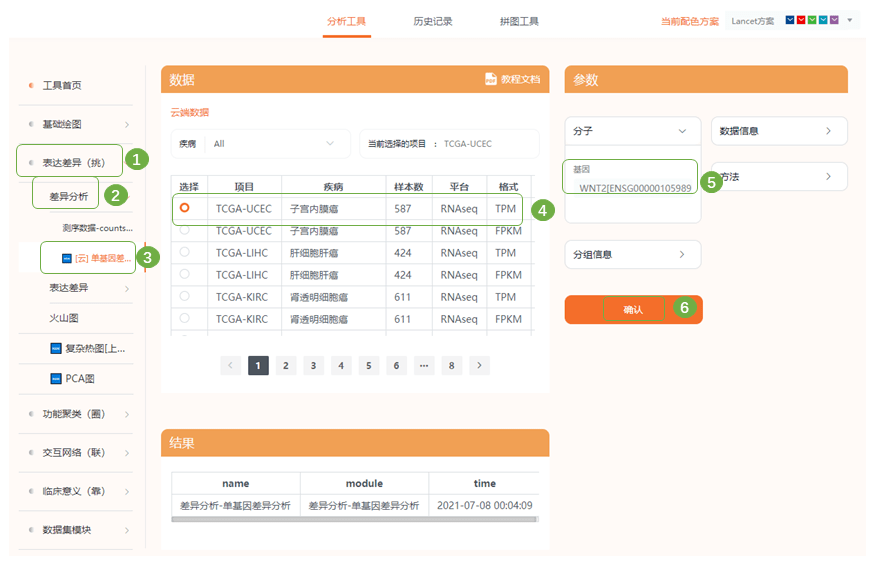
2)进入仙桃学术拼图工具完成拼图即可 Fig8 GSEA富集分析 1)作者使用TCGA_UCEC数据集,根据3个hub gene的表达水平分为高表达组与低表达组,对差异基因进行了GSEA富集分析;进入“表达差异(挑)—差异分析—单基因差异分析“,选择子宫内膜癌数据集,输入分析基因,提交分析
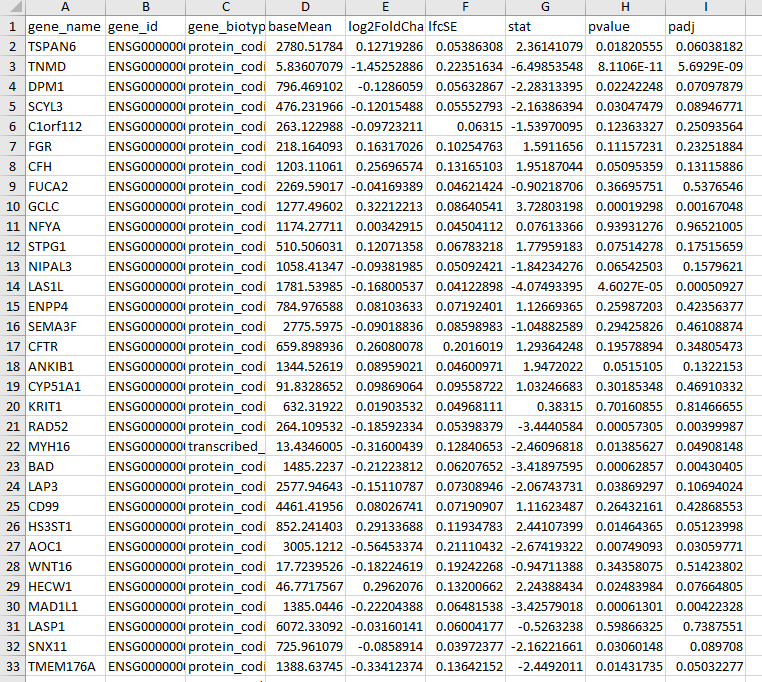
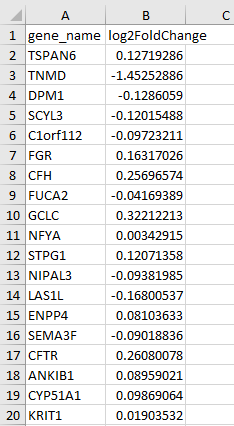
2)分析完成后在“历史记录“中下载结果表格,将数据整理为geneID、表达值(可为logFC)2列格式
单基因差异分析结果表格
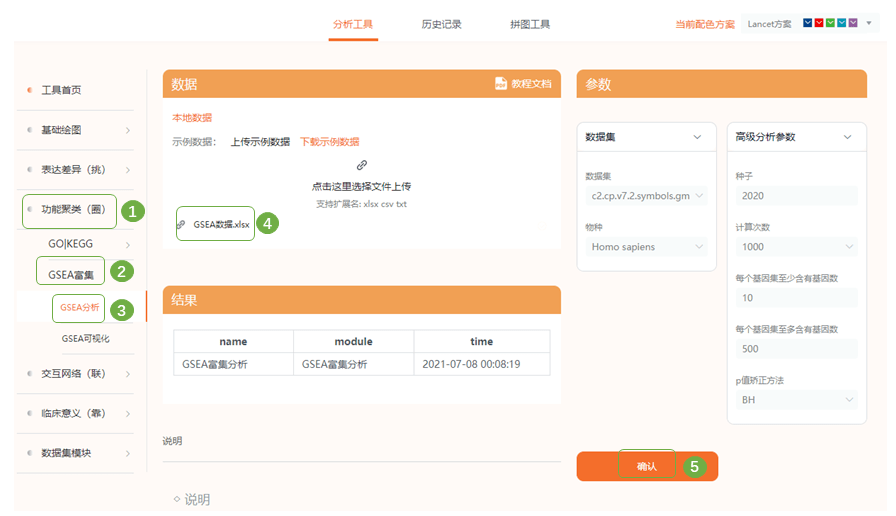
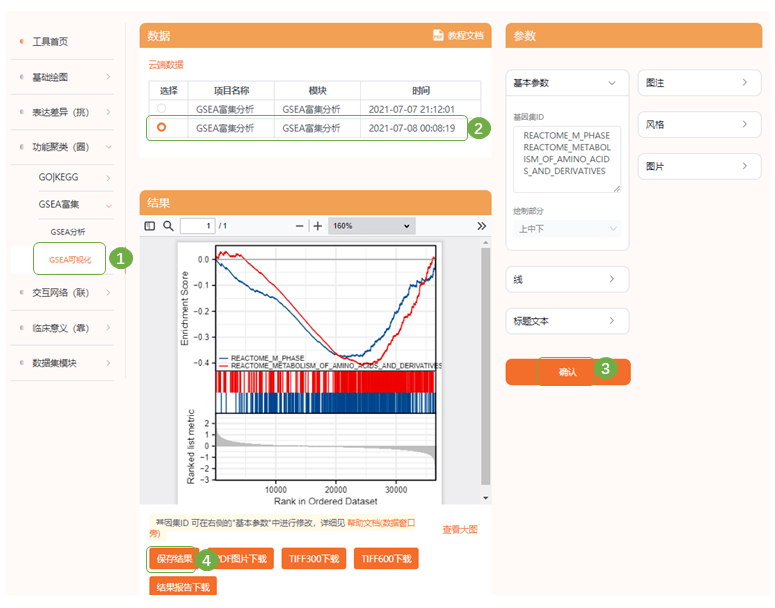
整理后的数据 3)进入“功能聚类(圈)—GSEA富集—GSEA分析“,上传整理好的数据集,提交分析
4)接下来进行GSEA富集分析可视化,选择刚才分析好的数据,提交分析,可以一幅图内展示5条通路,或手动删减通路数目,每次展示1条;保存结果
5)最后完成拼图即可 Fig9 Expression Atlas 分析3个hit gene 1)最后作者使用Expression Atlas数据库分析了3个hub gene在人体多种器官/组织中的基础表达量,发现FOXL2特异性地在女性子宫内膜与卵巢组织中高表达,推测其可能为女性生殖系统特异性基因。这个结论个人觉得与全文联系不是很紧密,我们也可以对hub gene的泛癌表达进行差异分析 2)进入“表达差异(挑)—表达差异—非配对样本“,选择”泛癌“数据集,右侧输入分析基因,点击“确认”提交分析,保存结果
3)对多个hub gene依次完成同样的分析,最后完成拼图即可 本文亮点 1、不同平台的4个GSE分别差异分析后取交集,筛选潜在预后生物标志物 2、TCGA数据集验证GEO数据集分析结果,提高分析结果可信度 3、思路简洁清晰,差异基因功能富集分析提示EC中可能改变的细胞生物学过程,为进一步的基础研究与临床干预提供可能的方向 看完这篇感觉我也可以立马复现了 是的,小编已为你准备了40多篇文献的详细复现操作来让你实操了 扫下方二维码免费领取干货返回搜狐,查看更多 |
【本文地址】
今日新闻 |
推荐新闻 |