深度学习数据标注之 |
您所在的位置:网站首页 › 输出png格式 › 深度学习数据标注之 |
深度学习数据标注之
|
深度学习的标注工具很多,其中labelme很多人都用过。很简单,但是标注之后需要的后处理操作比较繁琐,因此有许多人觉得也不是那么好用。因此,这里稍微分享一下我自己制作数据集的时候如何将json文件提取出png格式的方法吧。 至于labelme具体怎么安装和使用, 安装过程参考:https://github.com/wkentaro/labelme 使用过程参考:https://blog.csdn.net/shwan_ma/article/details/77823281 好,废话少说,先看看需求:  金相显微结构图 金相显微结构图【提出需求】:这是一张普通的金相显微结构图,假如我们现在的任务是需要把图像当中的深灰色区域在图像当中寻找出来,并生成mask,最后对生成的mask逐一筛选出符合所需要条件的部分,这部分是研究中需要用到的。 【设计思路】:那么很明显,我们的设计思路就是让模型能够在复杂的背景环境中正确分辨出来需要的区域,因此,采用labelme对原生数据进行标注,由于原来的图片范围过大,可以考虑将图片分割成几个小的部分,然后再对每个小部分进行标注。 【遇到问题】: 在这里,遇到的第一个问题:labelme标注出来的是只有一个json文件,这个好解决,我们的目的是得到标记的数据,json对我们来说,最终需要转换为单通道的图像(png),而且最好是每个标注的mask都能有单独的png图像,而如果采用这篇博文的做法https://blog.csdn.net/shwan_ma/article/details/77823281v,最终得到的是如下图的结果,从结果来看不是我们期望的。 例如我的文件名是micro,那么在命令行输入: labelme_json_to_dataset micro.json运行过程:  注意在运行之前应当切换到labelme的环境中 注意在运行之前应当切换到labelme的环境中最终得到的是一个文件夹,文件夹的内容是下图的样子:  很显然,我们需要的并不是这样的数据集,而是应该把label.png当中每个不同的mask都提取出来,单独生成一个图像文件。 【解决问题】:(在jupyter notebook中打开) 导入,这里说明的是,由于我自己的环境里没有labelme当中的一些库,所以需要手动添加到系统路径中 # -*- coding:utf-8 -*- #!/usr/bin/env python import argparse import json import matplotlib.pyplot as plt #由于我自己的环境里没有labelme当中的一些库,所以需要手动添加到系统路径中 import sys sys.path.append('C:\\Users\Administrator\Anaconda3\envs\labelme\Lib\site-packages') from labelme import utils import numpy as np from labelme.utils import image打开notebook 的路径是在与micro.json同一路径下,如果不是的话,可以参考我上述代码当中的 sys.path.append('C:\\Users\Administrator\Anaconda3\envs\labelme\Lib\site-packages')将自己的json文件路径也加入到系统路径中去。 接下来,我们需要了解一个文件当中的一个函数,由于我们之前导入了: from labelme.utils import image我们尝试在库文件当中寻找这个文件:  打开后:  image.py当中的内容是这样的:作用是解析原图片数据,并以array形式返回。 有些博客当中的函数是img_b64_to_array,怀疑是因为不同的labelme版本导致。  继续,那么我们首先得需要读入数据,那么有: data = json.load(open(json_file)) # 加载json文件shape.py当中的内容:作用是解析shapes字段信息,最后返回的lbl是mask,而label_name_to_value是对应的label。  为了保存mask信息与对应的label,则有: lbl, lbl_names = utils.labelme_shapes_to_label(img.shape, data['shapes'])# 解析'shapes'中的字段信息,解析出每个对象的mask与对应的label # lbl存储 mask,lbl_names 存储对应的label# lbl 像素取值 0、1、2、3、4 其中0对应背景,1对应第一个对象,2对应第二个对象……以此类推接下来,我们的思路应该是遍历每个不同的像素值(0是背景,可以跳过不取,1、2、3、4,因为我们这张图片除去背景只有4个mask),并单独取出mask,因此有: mask=[] class_id=[] for i in range(1,len(lbl_names)): # 跳过第一个class(因为0默认为背景,跳过不取!) mask.append((lbl==i).astype(np.uint8)) # 举例:当解析出像素值为1,此时对应第一个mask 为0、1组成的(0为背景,1为对象) class_id.append(i) # mask与class_id 对应记录保存可是保存下来的Mask并不是这么容易可以直接输出成一个图片的,如果我们直接用matplotlib输出,则会看到报错: plt.imshow(mask[:,:,0],'gray') 这个报错“TypeError: list indices must be integers or slices, not tuple”,意思是我们需要转换一下,不能用tuple的格式,所以有: mask=np.asarray(mask,np.uint8)这样,就能够在plt中输出图像了:  不对,好像有点不太对劲,对比一下原图(含标注):  有没有发现有什么问题?!是的,方向不太对! 正确的输出应当是:  发现问题了吗?我们应该逆时针旋转90°!因此,将mask逆时针旋转90°的操作是必须的。故而应当改为: mask=np.transpose(np.asarray(mask,np.uint8),[1,2,0])至此,似乎也就可以了?No,实际上我之前也提到过了,这个时候的Mask图片有效区域的像素是以1,2,3,4这样的取值存在的,所以假如我们直接保存出来会是个怎么样的图片呢?看看: #不经过处理直接写入mask import cv2 cv2.imwrite("mask1111.png", mask[:,:,0])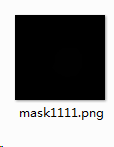 可以看到一片漆黑,果然是因为像素取值过低导致的,虽然背景的像素取值是0,不过Mask也只不过是1,2,3,4,所以最好的思路应当是将Mask进行二值化处理! 这里二值化处理的目的主要是为了可视化Mask,我个人认为对于实际训练的时候的影响是没有的: retval, im_at_fixed = cv2.threshold(mask[:,:,0], 0, 255, cv2.THRESH_BINARY) cv2.imwrite("mask_1111_real.png", im_at_fixed)最终效果:  当然了,mask可不止这一个,得全都输出出来,对吧? for i in range(0,len(class_id)): retval, im_at_fixed = cv2.threshold(mask[:,:,i], 0, 255, cv2.THRESH_BINARY) cv2.imwrite("mask_out_{}.png".format(i), im_at_fixed)看看最终结果:  还是比较理想的吧,所有Mask都分别输出了。输出png的任务基本完成! |
【本文地址】
今日新闻 |
推荐新闻 |