转录组和蛋白组如何关联分析?先从绘制九象限图开始 |
您所在的位置:网站首页 › 绘制时间象限 › 转录组和蛋白组如何关联分析?先从绘制九象限图开始 |
转录组和蛋白组如何关联分析?先从绘制九象限图开始
|
转录组和蛋白组如何关联分析?先从绘制九象限图开始
五种常用蛋白质组学定量分析方法对比 - 知乎 (zhihu.com)
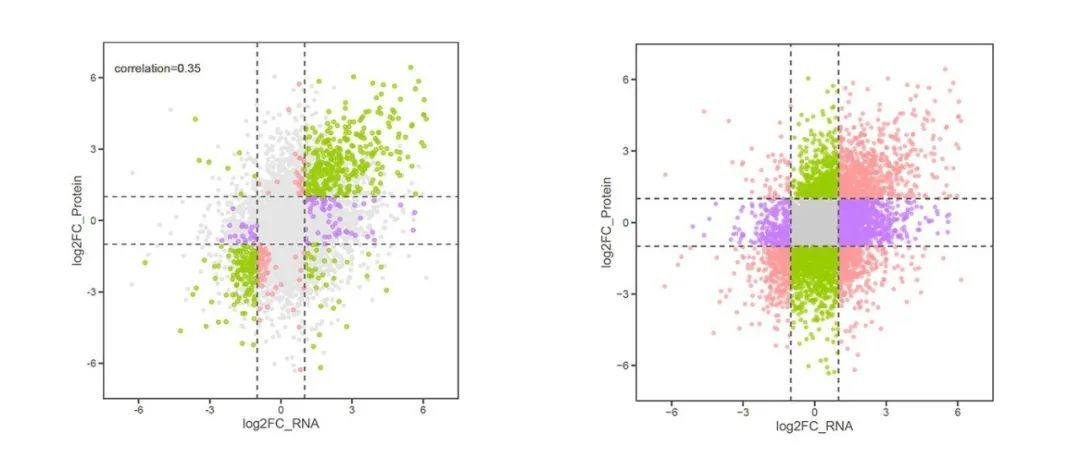
九象限图在多组学关联分析中非常重要,例如我们可以用九象限图展示“转录组+蛋白组”、“转录组+翻译组”等关联分析中不同基因的差异表达情况。通过分析两种组学数据的相关性,我们可进一步缩小候选基因的范围。
当然,之前的微信文章《Graphpad Prism能不能画九象限图?》中介绍过九象限图的画法,下面再为大家介绍一种效果更好看的画法。 1. 数据合并 #读入两个差异分析表(分别是转录组和翻译组)并合并; #设置工作目录; setwd("C:/Users/86136/Desktop/九象限图的绘制") #查看工作目录中的文件; dir() #[1] "protein_exp.csv" "RNA_exp.csv" #读入数据; RNA =read.csv("RNA_exp3.csv",header=T) protein=read.csv("protein_exp3.csv",header=T) #查看数据的维度; dim(RNA) #[1] 10565 5 #查看前6行; head(RNA) dim(protein) #[1] 7028 5 #查看前6行; head(protein)
|
【本文地址】