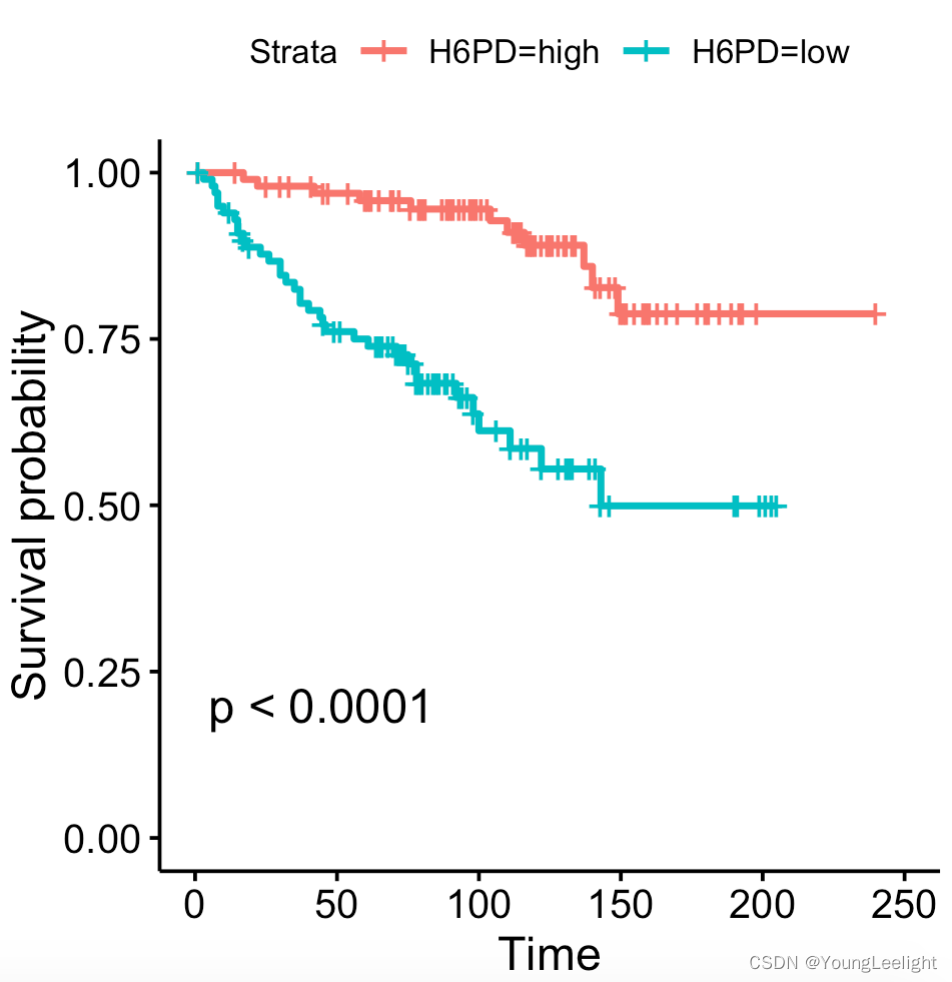
生存分析 存活分析 survival analysis 基因的 高低表达生存分析 按照基因表达量的高低做生存分析 批量基因批量生存分析 做生存分析,已经不需要正常样本的表达矩阵了,所以需要过滤 |
您所在的位置:网站首页 › 生存分析meta提取数据的方法 › 生存分析 存活分析 survival analysis 基因的 高低表达生存分析 按照基因表达量的高低做生存分析 批量基因批量生存分析 做生存分析,已经不需要正常样本的表达矩阵了,所以需要过滤 |
生存分析 存活分析 survival analysis 基因的 高低表达生存分析 按照基因表达量的高低做生存分析 批量基因批量生存分析 做生存分析,已经不需要正常样本的表达矩阵了,所以需要过滤
|
这里做生存分析,已经不需要正常样本的表达矩阵了,所以需要过滤。
而且临床信息,有需要进行整理。
survival analysis only for patients with tumor.
数据准备: 1.phe 临床信息 dataframe格式 。行名顺序要与表达矩阵样本顺序一致 , #####至少包括是否死亡event 生存时间time 以及分类标准(基因高低 肿瘤分期 是否转移等) 2.表达矩阵
临床信息 meta信息 |
【本文地址】
今日新闻 |
推荐新闻 |

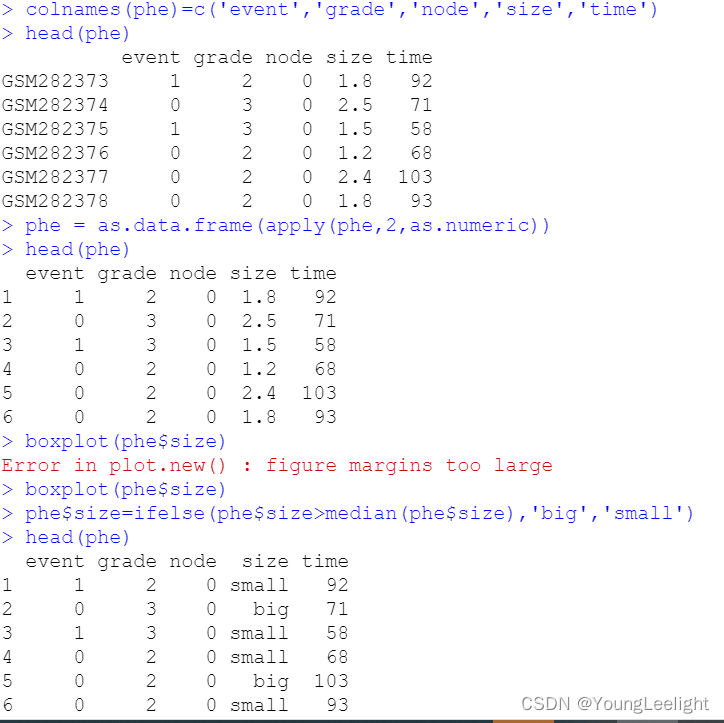 给感兴趣的指标进行赋值
给感兴趣的指标进行赋值 