什么?!一下午搞定一篇4+文章的所有数据,0代码教你复现! |
您所在的位置:网站首页 › 生信发SCI需要提供代码吗 › 什么?!一下午搞定一篇4+文章的所有数据,0代码教你复现! |
什么?!一下午搞定一篇4+文章的所有数据,0代码教你复现!
|
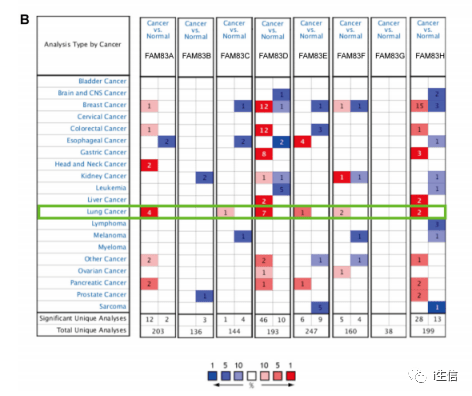
Figure 1B 通过 ONCOMINE 数据库展示 FAM83 家族在多种癌症包括肺癌中的异常表达情况
首先,根据网址https://www.oncomine.org/登陆Oncomine数据库,登陆时需要账号密码,该数据库分为免费版和收费版,一般免费版足够满足日常分析需求了。免费注册仅限非营利性机构邮箱,例如院校或研究机构邮箱。 登陆后直接在搜索框中输入目标基因FAM83A
输入后网页自动刷新就会出现该基因在各种癌症中的表达情况
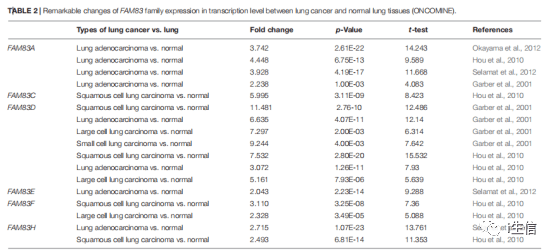
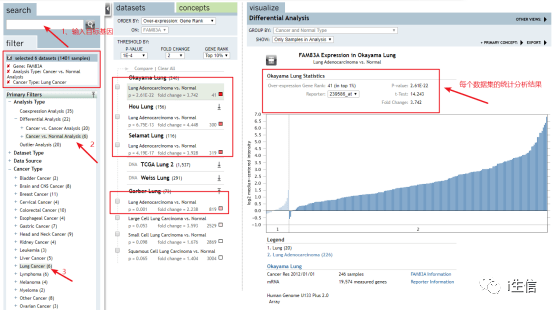
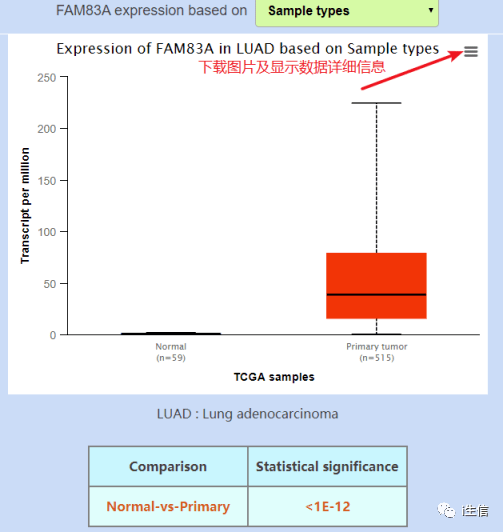
此时FAM83A的表达情况就分析完成了,按照相同的步骤分析完其他基因后,把所有基因的表达图用绘图软件拼在一起就完成了Figure 1。这一步中大部分情况直接使用网站默认的筛选参数就可以,当然也可以根据自己的情况进行调整。 Table 2 肺癌与正常肺组织中 FAM83 家族转录水平的显著变化
这个表格仍然是通过Oncomine数据库中的数据集获得的。首先仍然是登录网站,第一步在搜索框中输入FAM83A,随后选择分析类型Cancer vs. Normal Analysis,再选择分析癌种Lung Cancer。 结果在右侧显示,根据P值进行筛选,癌症组和正常组表达有差异的共有四个数据集,点击每个数据集就可以得到统计分析的各项数值如Fold change、P值、t-test值等等。同样,按照上述步骤完成其他基因的表达分析并将结果整理到表格中就完成了Table 2。
Figure 2 UALCAN 数据库中 FAM83 家族在肺鳞癌和肺腺癌中的表达情况
这一步中作者验证了FAM83家族中每个成员在肺腺癌和肺鳞癌中的表达情况,此处以FAM83A为例展示分析步骤。 首先登录UALCAN网页http://ualcan.path.uab.edu/,点击TCGA analysis
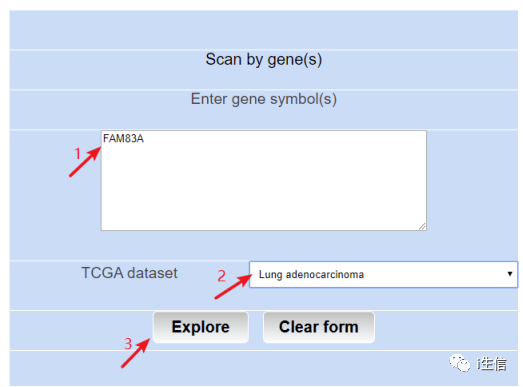
随后将需要分析的目标基因FAM83A输入进去,下一步选择需要分析的癌种,这里选择肺腺癌,最后点击explore。
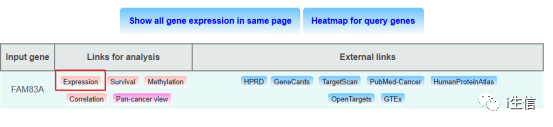
页面刷新后,点击Expression,即显示基因的表达信息。
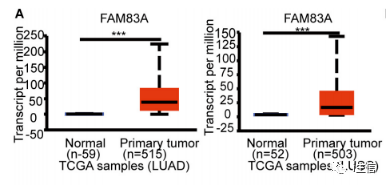
页面刷新后基因在肺癌组和正常组的表达情况就显示了,可以直接下载图片用到论文里,也可以下载后通过修图软件进一步修饰。
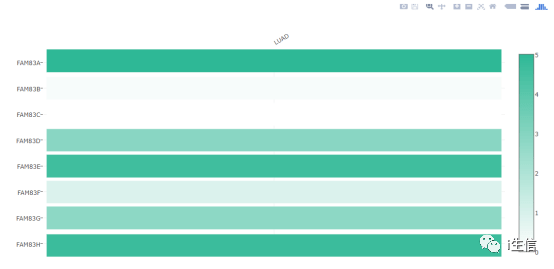
这一步的图就复现出来了,改变癌症类型和目标基因就可以绘制出文章中Figure 2全部的图片。 Figure 3 FAM83 家族在 NSCLC 的相对表达水平热图
首先登录GEPIA网页http://gepia.cancer-pku.cn/,点击Multiple Gene Analysis,随后点击Multiple Gene Comparison。
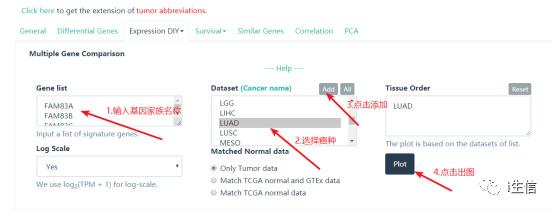
进入新的页面内,首先将目标基因家族全部输入到Gene List 框内,然后选择癌种,点击添加,最后点击Plot就可以了。
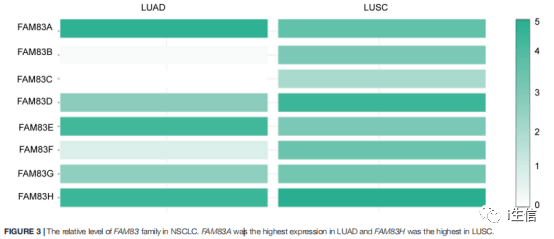
肺腺癌的基因家族表达结果如下图。
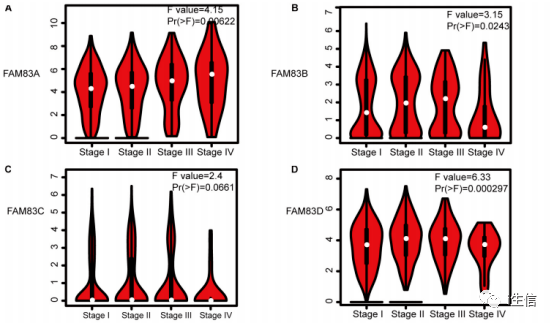
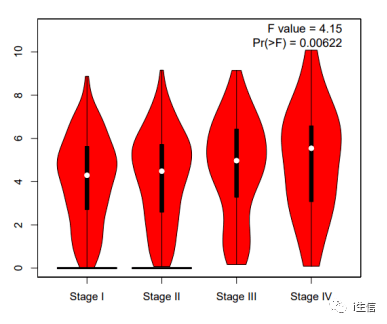
这个图片可以直接下载,按照上述步骤再分析基因家族在肺鳞癌的表达,下载图片并将图片拼合就完成了Figure 3。 Figure 4 不同 FAM83 家族成员 mRNA 表达与肿瘤分期的关系
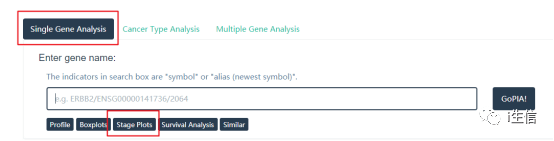
这一步作者分析了FAM83家族所有的成员,我们以FAM83A为例。 首先登录GEPIA网页http://gepia.cancer-pku.cn/,点击Single Gene Analysis,随后点击Stage Plots。
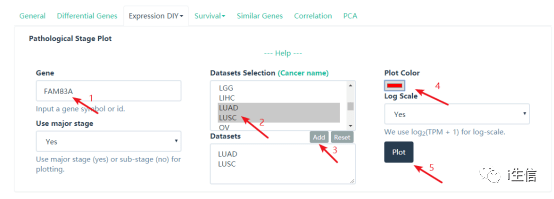
进入新的页面,第一步在输入框内输入研究的基因名称,第二步选择研究的癌种,第三步点击Add,第四步在Plot color下可以选择图形的颜色,设置好后最后点击Plot即可。
FAM83A的mRNA表达与肿瘤分期的关系如下图,按照上述步骤完成其他基因的分析并将图片拼合即可完成Figure 4。
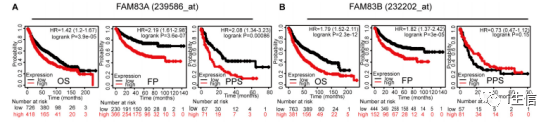
Figure 5 肺癌中 FAM83 家族的预后特征
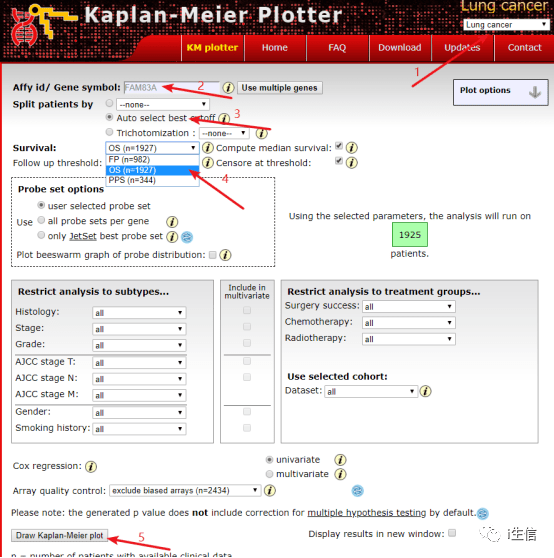
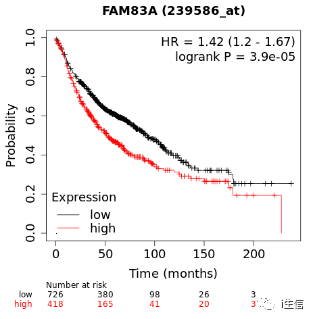
这一步我们仍旧以FAM83A为例进行复现,首先登录Kaplan-Meier plotter数据库网页http://kmplot.com/。 第一步选择癌种,第二步输入研究的目标基因,第三步如何对病人分组选择Auto selcet best cutoff,第四步选择生存分析的类型是OS、FP还是PPS,首先做OS,第五步点击绘图。
FAM83A在肺癌中的OS分析结果如下图。
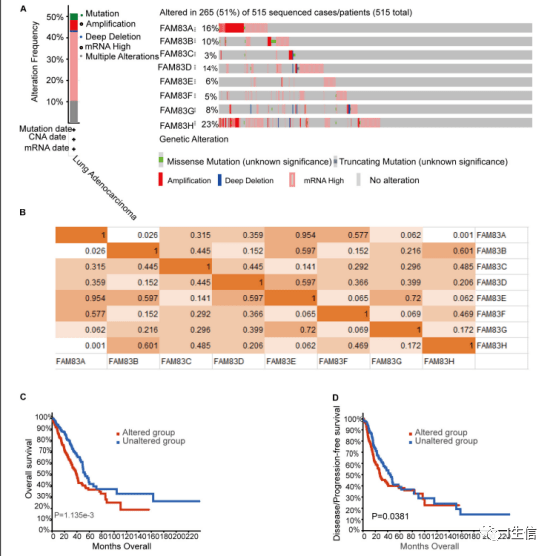
按照上述步骤,再选择分析FAM83A在肺癌中的FP和PPS就完成了这个基因的全部分析,其余基因分析步骤相同。 Figure 6 FAM83 家族基因突变情况及其与预后的关系
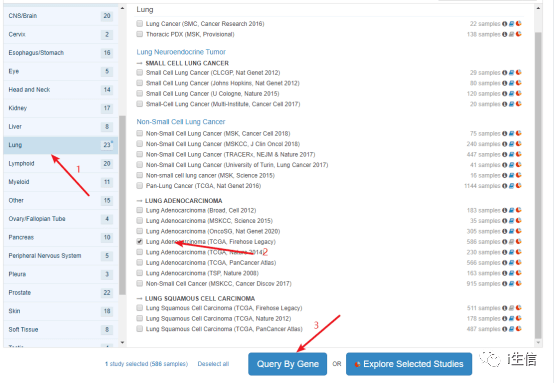
首先登录cBioPortal 官方网站http://www.cbioportal.org/,根据文章里方法部分的介绍,第一步选择研究种类为肺,第二步选择研究的数据集,第三步点击Query By Gene,进入下一个页面。
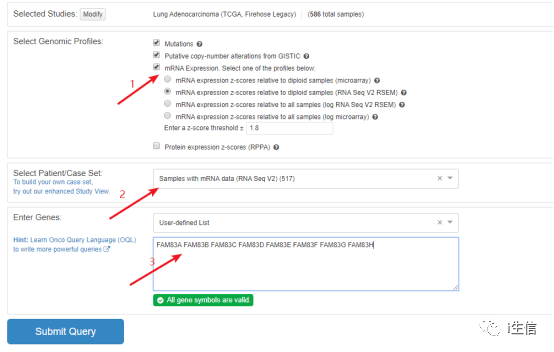
这一步仍旧是按照文章里方法部分介绍的条件来设置参数,第一步按作者的设置调整select Genomic Profiles里的选项,第二步勾选要分析的数据集,第三步将基因家族输入到基因框中,点击submit Query进入结果页面。
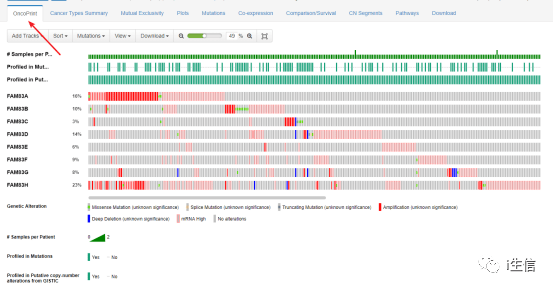
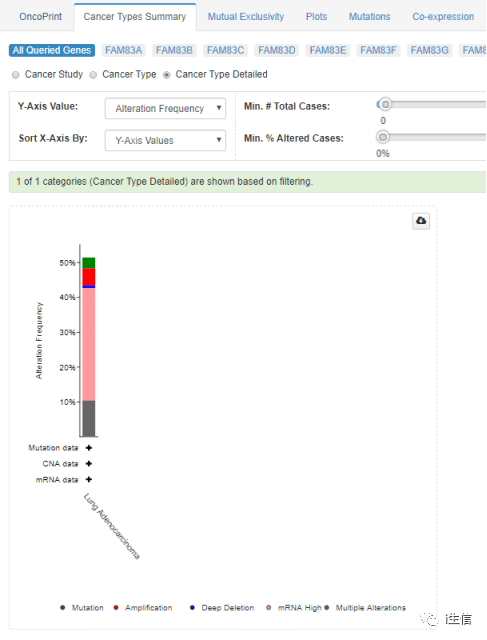
进入新的页面后出来的就是Oncoprint页面上的结果,也就是Figure 6A的内容。
随后点击Cancer Types Summary,就绘制出了Figure 6A的条形图。
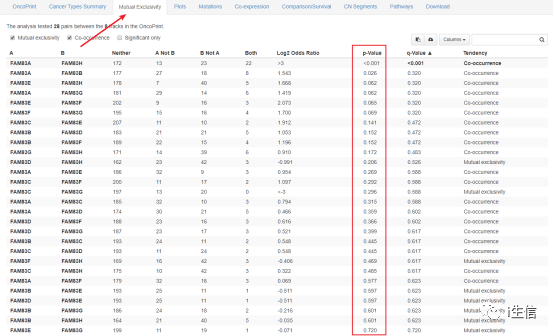
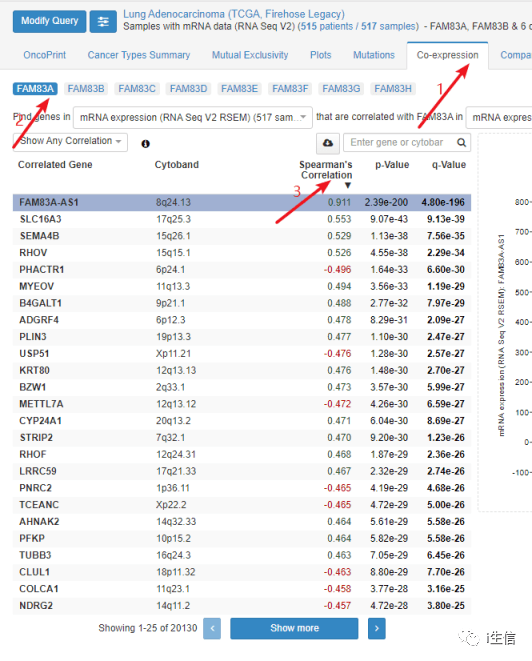
随后是FAM83家族成员之间表达的相关性,点击Mutual Exclusivity,整理表格并将结果中的P值填进去,最后填充颜色就完成了Figure 6B。
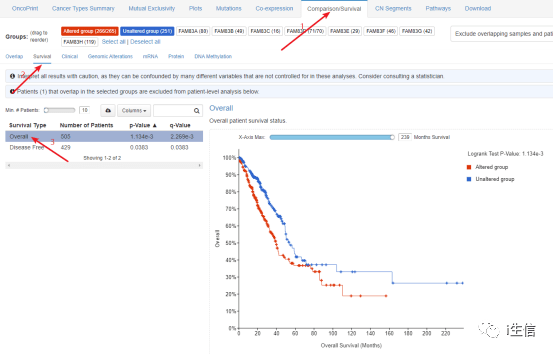
这一步是生存分析,第一步点击Comparion/Survival,第二步点击下面的Survival,第三步选择生存分析的类型就可以绘制出OS或DFS的生存曲线,图片加载出来后直接下载进行拼合即可。
Figure 7 FAM83 家族及其共表达基因的互作网络和富集分析
这一步中,作者首先利用cBiopotal的“共表达”模块分析了与FAM83家族突变显著相关的80个共表达基因,即每个家族基因选取共表达系数最高的前十个基因。这一步可以接着Figure 6的分析继续往下做,选择Co-expression,点击FAM83A,然后根据相关性系数选取前十个基因,家族其他基因相同操作后获得80个共表达基因。
获取共表达基因后进入STRING数据库页面https://string-db.org/
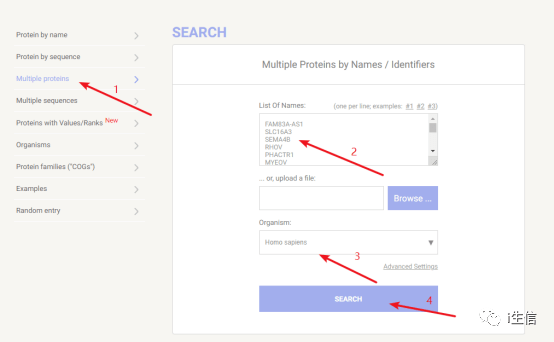
点击SEARCH,进入下一个页面。第一步选择Multiple proteins,第二步将80个基因输入到列表中,第三步选择研究的组织为Homo sapiens,第四步点击SEARCH。
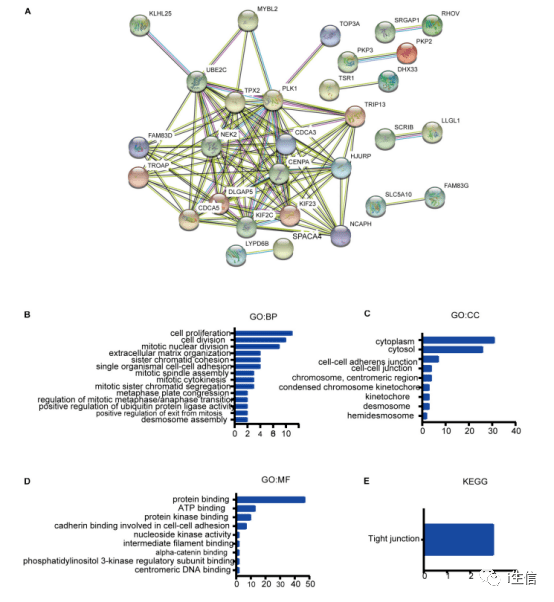
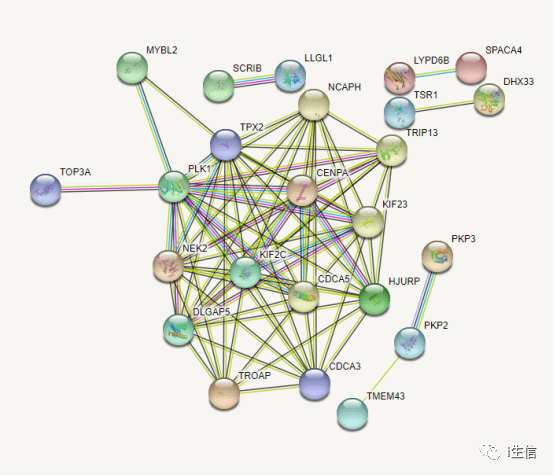
蛋白互作网络如下图,可以根据网页设置调整自己的分析参数。
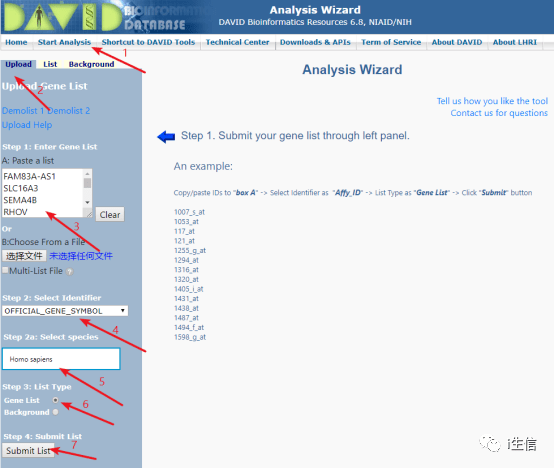
下一步就是共表达基因的GO和KEGG分析,由DAVID数据库完成,首先登陆网页https://david.ncifcrf.gov/。然后按照图片中的步骤选择参数,在第三步中将80个基因输入框中。
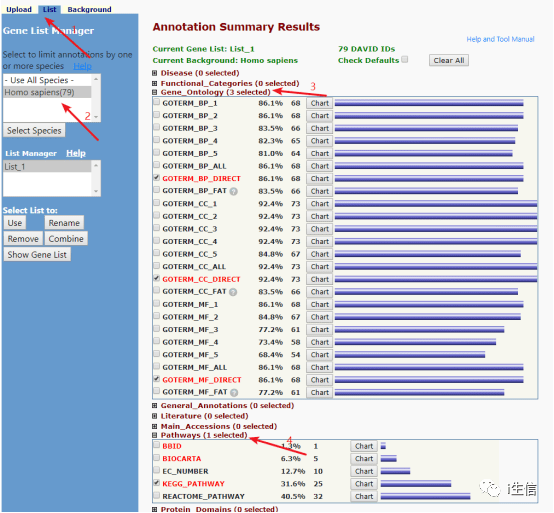
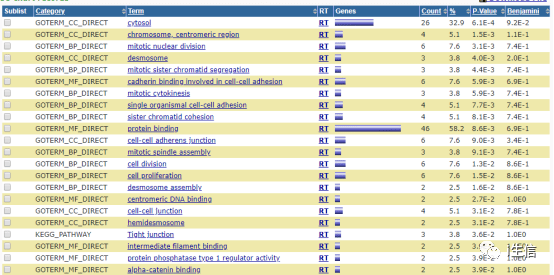
点击submit List后进入下一个页面,按照图片中的步骤选择需要分析的GO和KEGG选项,选择完毕后点击Functional Annotation Chart,即可获得分析结果。
将结果下载并导入到excel中,根据P值和富集的基因数对通路进行筛选并绘图就可以得到Figure 7B-E。
到这里我们已经完成了文章全部的图表,文章分析步骤并不复杂,拿出自己研究的基因快来试试分析吧。
注:本推文未经许可禁止转载。 文献详解 | 单细胞测序数据这样分析,说不定你也能发Science 文献详解 | 10分+期刊单细胞测序肿瘤研究范文,大家赶紧“抄作业” 文献详解丨RNA修饰writer你听说过吗?纯生信发15+文章的新思路 文献解析丨又一篇IF=6.852肿瘤预后纯生信套路文章,巨详细的思路解析,快来复制! 文献详解丨给脑胶质瘤绘制家谱,单细胞测序在肿瘤异质性和发育研究中的大显神通!如果您或者科室有科研上的困扰 扫码备注:科研合作返回搜狐,查看更多 |
【本文地址】
今日新闻 |
推荐新闻 |