无需编程 |
您所在的位置:网站首页 › 瓦时数怎么计算 › 无需编程 |
无需编程
|
如何计算遗传相关? 问题描述 植物或林木育种中,我的数据类型包括基因型(品种),地点(环境),重复(或者无重复),还有一系列性状,比如株高、产量等等,我怎么计算性状间的遗传相关,并且给出显著性? 示例数据 数据描述:location为地点,Genotype为基因型,Rep为重复,DWKPlant, DWKEar, TW, KDM, TKW为性状,想要计算这5个性状之间,两两的遗传相关和表型相关,并给出显著性。  常见误区  直接计算表型值的相关系数r,r这里并不是遗传相关和表型相关,而只是性状间的相关系数 正确做法 运行多性状模型,计算性状间的遗传协方差组分(G12)和遗传方差组分(G11,G22) 图中,黄色的表示方差组分,绿色的表示协方差组分,蓝色的遗传相关值: 计算公式:  Genstat界面  数据  模型1: h4和h5为多性状,固定因子:Rep + Spacing, 随机因子:Fam,矩阵结构:使用us矩阵。option中选择Deviance。  结果: 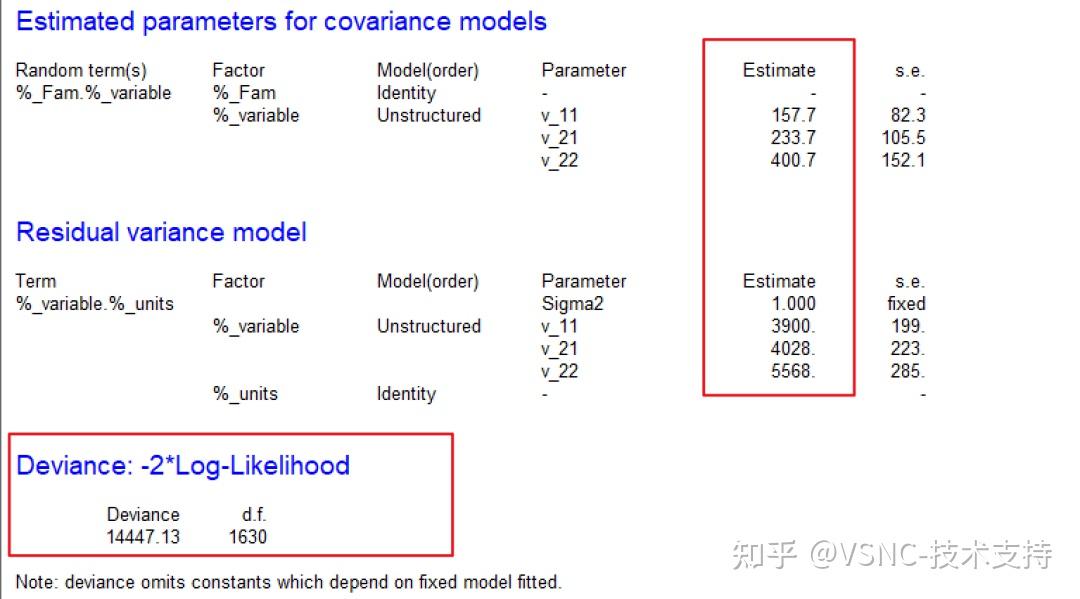 模型2: h4和h5为多性状,固定因子:Rep + Spacing, 随机因子:Fam,矩阵结构:使用Diagonal矩阵。option中选择Deviance。  结果:  这个Deviance,计算方法为:-2* loglikelihood,将结果放到Excel中:  在Excel中,用chisq.dist.rt函数,卡方为模型2-模型1,自由度为模型2-模型1, =CHISQ.DIST.RT(G6-B6,H6-C6) 结果:  与ASReml-R结果比较: library(asreml) mod1 = asreml(cbind(h4,h5) ~ trait + trait:(Spacing + Rep), random = ~ us(trait):Fam, residual = ~ units:us(trait), data=fm) summary(mod1)$varcomp mod2 = asreml(cbind(h4,h5) ~ trait + trait:(Spacing + Rep), random = ~ diag(trait):Fam, residual = ~ units:us(trait), data=fm) summary(mod2)$varcomp mod3 = asreml(cbind(h4,h5) ~ trait + trait:(Spacing + Rep), random = ~ diag(trait):Fam, residual = ~ units:diag(trait), data=fm) summary(mod3)$varcomp ## 遗传相关及显著性检验 vpredict(mod1,h2 ~ V2/sqrt(V1*V3)) lrt.asreml(mod1,mod2,boundary = F) ## 表型相关及显著性检验 vpredict(mod1,h2 ~ (V2+V6)/sqrt((V1+V5)*(V3+V7))) lrt.asreml(mod1,mod3,boundary = F) 结果:  上面的卡方值1133,就是模型2减去模型1的值,自由度就是模型2减去模型1的自由度,是1632-1630=2。  还想了解更多Genstat 功能?请看这里! GenStat是一款拥有强大试验设计、数据分析功能的国际专业统计软件,软件操作简单,国际认可度高,并具有强大的专业分析功能。以Genstat 为主题的“生物统计分析与试验设计”课程应运而生,课程从试验设计、基础统计分析、混合线性模型以及进阶分析,层层递进,结构清晰严密。对于软件的优势功能比如不平衡数据的多重比较、计算遗传力和育种值、基因与环境互作(GbyE,AMMI,GGE模型)都有详细介绍及SCI 案例解析。内容参考如下:   希望大家都能通过Genstat软件学会生物统计与试验设计的方法! |
【本文地址】
今日新闻 |
推荐新闻 |