一步步告诉你NCBI |
您所在的位置:网站首页 › 测序回来的序列怎么对比 › 一步步告诉你NCBI |
一步步告诉你NCBI
|
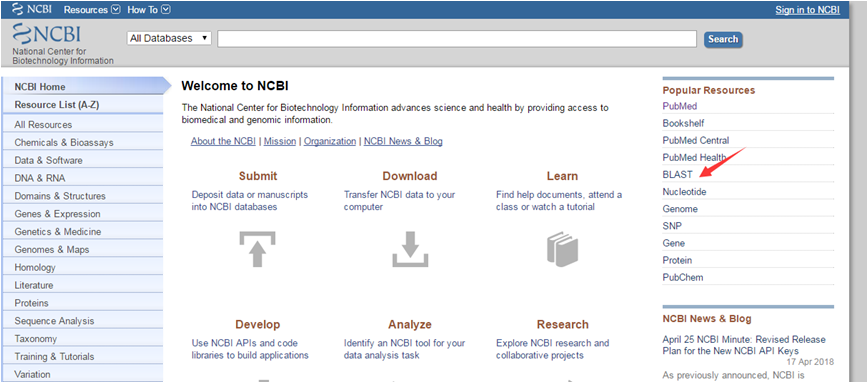
今天给大家分享个简单快速比对测序结果的方法:通过NCBI-BLAST分析测序结果,话不多说我们一起来看看具体的操作方法。(1)打开NCBI(https://www.ncbi.nlm.nih.gov/),找到Popular Resources栏中的BLAST,点击进入;或者直接搜索网址:https://blast.ncbi.nlm.nih.gov/Blast.cgi
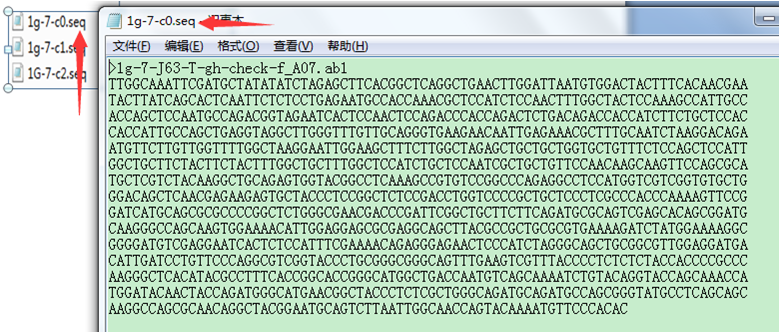
(5)我们将构建好的质粒交给测序公司测序,同时向公司提交测序引物,待测序公司完成测序后公司会反馈给我们测序结果。在测序结果中找到.seq 文件,将文件打开进行序列比对。
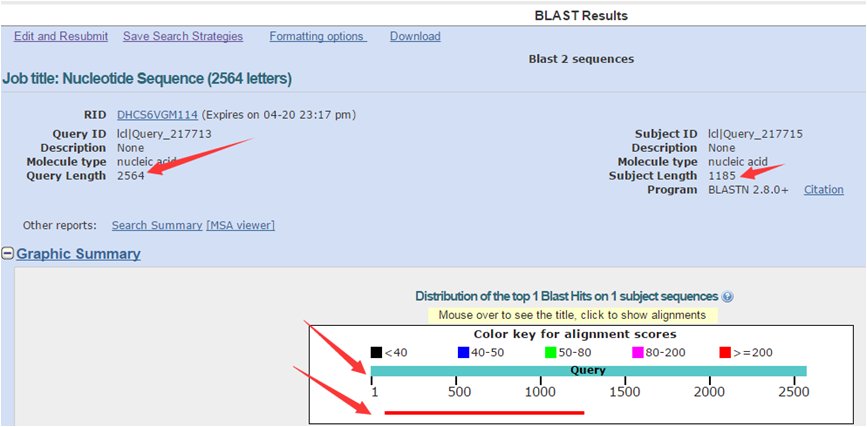
(7)大家可以看到下图:红线代表测序序列,蓝绿色的线是我放在Enter Query Sequence框中的基因A原序列。 (8)比对测序结果比对结果显示两排序列,上排的序列是之前放在“Enter Query Sequence”框中的序列,也就是我们A基因的原序列;下排序列是放在“Enter Subject Sequence”框中的序列,也就是我们的测序序列。大家可以看到,红色箭头处是我标记的碱基缺失或突变的地方,看到这么红色箭头,大家先不要紧张,首先可以看出比对序列结果中出现碱基缺失或者碱基突变的区域大部分在测序结果的后半部分,而之前也有提到过测序仪它能准确测序的部分是整个序列的中间部分,前150 bp和后150 bp(这个长度也不是一定的,可能会有浮动)的测序结果并不准确,所以这些红色箭头的地方不一定就是错误的,还要结合我们第二个引物的测序结果(即:第二个引物测序是将第一个引物测序的后150-200 bp序列包含在其能准确测序的位置上),第二段测序引物的测序结果就不给大家展示了,可以和大家分享的是,将这个基因的3个测序引物的测序结果整合后,我们的这个基因序列与原序列完全一致,也就是说我们的质粒构建成功~~~ |
【本文地址】
今日新闻 |
推荐新闻 |
 (2)选择Nucleotide BLAST 点击进入
(2)选择Nucleotide BLAST 点击进入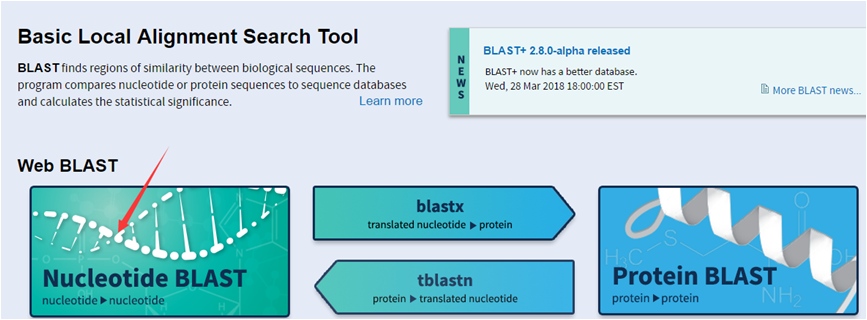 (3)进入BLAST界面后勾选“Align two or more sequences”
(3)进入BLAST界面后勾选“Align two or more sequences”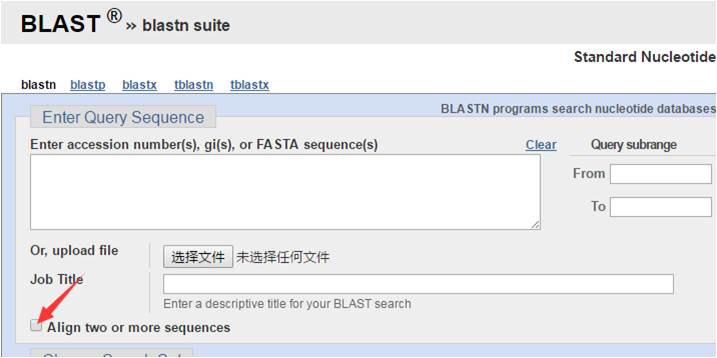
 (4)以基因A为例,我们基因A约2400 bp,测序仪一次不能测得全长,我们我就要设计至少3个引物进行测序,标记处为设计的测序引物。此时需要注意的是引物和引物之间的距离要掌握好,由于测序仪器测出来的序列可能在前150 bp和后150 bp都不准确,所以设计引物是需考虑到这一点,例如我们的第一个引物就不是从起始密码子ATG开始,而是在ATG上游150-200 bp处设计一段引物。同样第二个和第三个引物也要考虑到这点,确保我们的测序结果完全可靠。
(4)以基因A为例,我们基因A约2400 bp,测序仪一次不能测得全长,我们我就要设计至少3个引物进行测序,标记处为设计的测序引物。此时需要注意的是引物和引物之间的距离要掌握好,由于测序仪器测出来的序列可能在前150 bp和后150 bp都不准确,所以设计引物是需考虑到这一点,例如我们的第一个引物就不是从起始密码子ATG开始,而是在ATG上游150-200 bp处设计一段引物。同样第二个和第三个引物也要考虑到这点,确保我们的测序结果完全可靠。
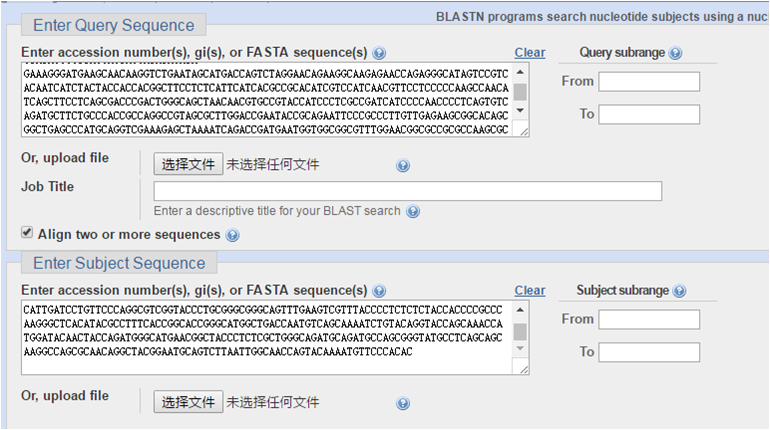
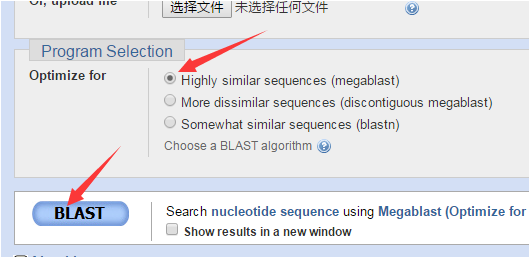
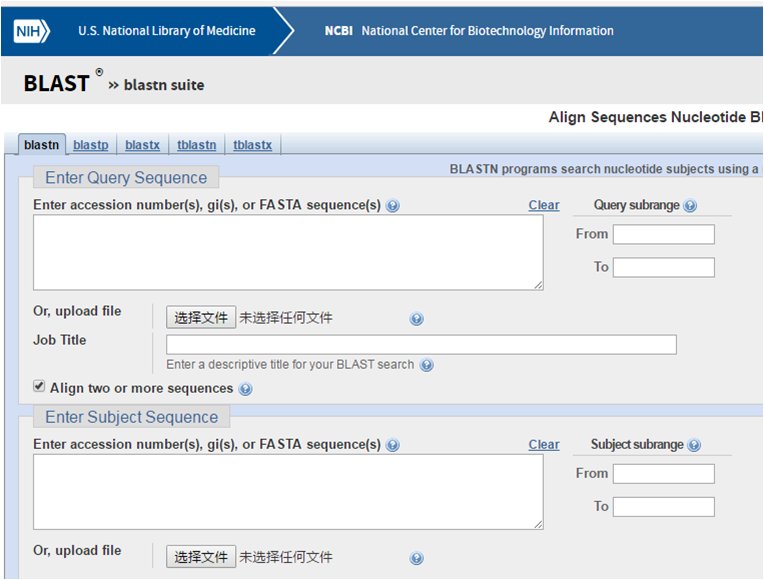
 (6)在BLAST的Enter Query Sequence和Enter Subject Sequence中分别复制粘贴序列源文件和测序文件,个人习惯将基因原序列放在Enter Query Sequence,将测序结果放在Enter Subject Sequence,这样看的方便些。点击BLAST
(6)在BLAST的Enter Query Sequence和Enter Subject Sequence中分别复制粘贴序列源文件和测序文件,个人习惯将基因原序列放在Enter Query Sequence,将测序结果放在Enter Subject Sequence,这样看的方便些。点击BLAST