【吉锐生物】SnapGene 的使用方法 |
您所在的位置:网站首页 › 氨基酸序列表怎么看 › 【吉锐生物】SnapGene 的使用方法 |
【吉锐生物】SnapGene 的使用方法
|
Snapgene 是做分子克隆常用的一个工具软件,学好它可以让我们的实验过程变得更加快捷方便。这里会我们围绕以下 7 个功能为大家做一下讲解: 批量添加不同的酶切位点 对片段进行命名 给质粒图谱增加引物序列 模拟标准限制性克隆 模拟融合克隆 模拟电泳功能 序列比对功能 首先介绍 Snapgene 的主要界面:
最下方的视图栏: (点击下方的图标按钮可以进行相互切换) 最上方的菜单栏:
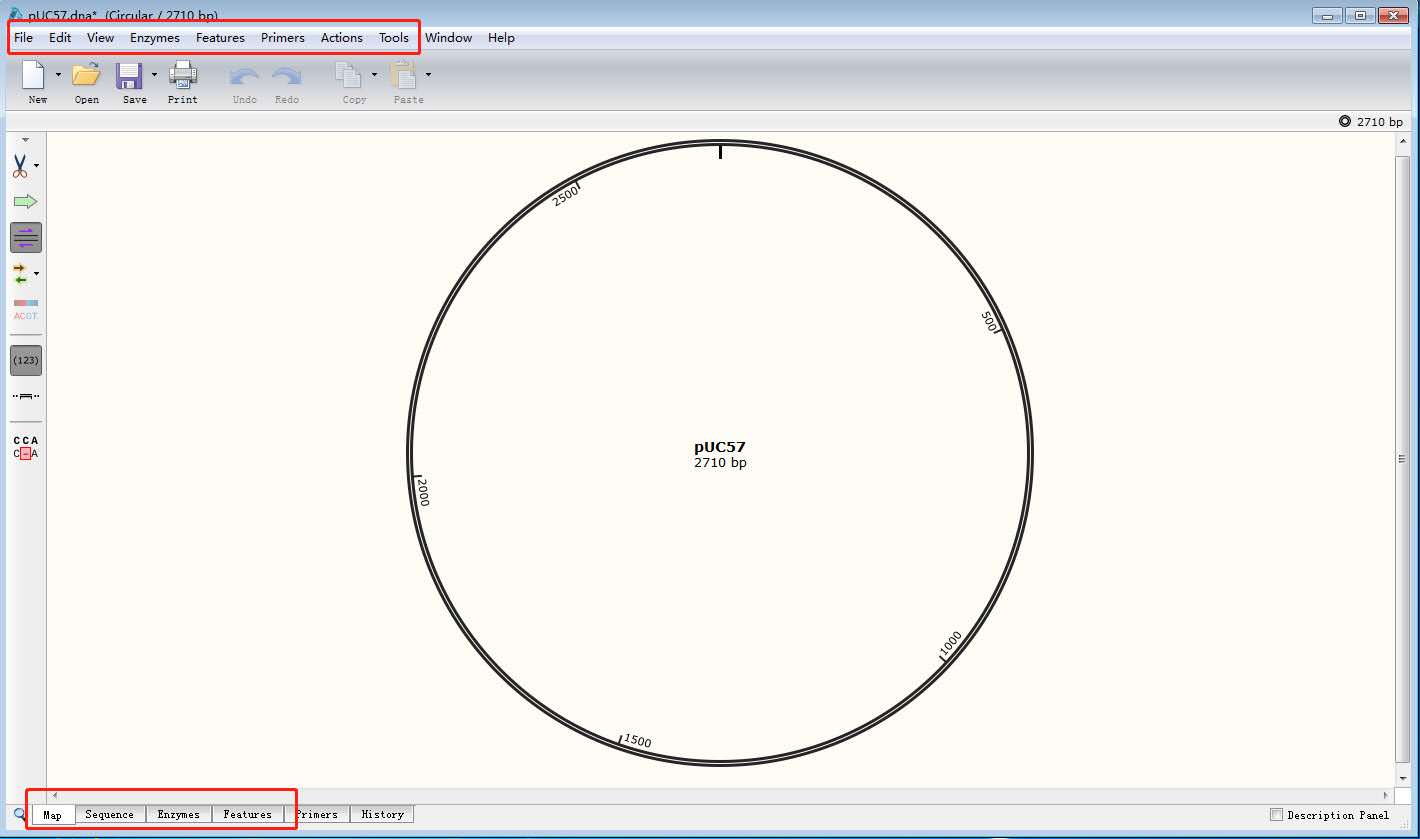
对应的功能如下: Map:显示图谱 Sequence:显示序列信息 File:打开、建立、保存相关文件等 Edit:编辑功能,包括复制选中一些序列等 View:切换几个视图等 Enzymes:显示、选择酶切位点等 Feature:显示、增加(命名)一些片段等 Primers:添加引物等 Action:插入融合一些片段 Tool:模拟电泳、序列比对、查看 DNA 分子量、查询简并密码子、氨基酸等 首先我们介绍这个软件的四个视图:视图一:Map直接打开任一质粒图谱(以 pUC57 为例),点击 Map 按钮,得到如下界面: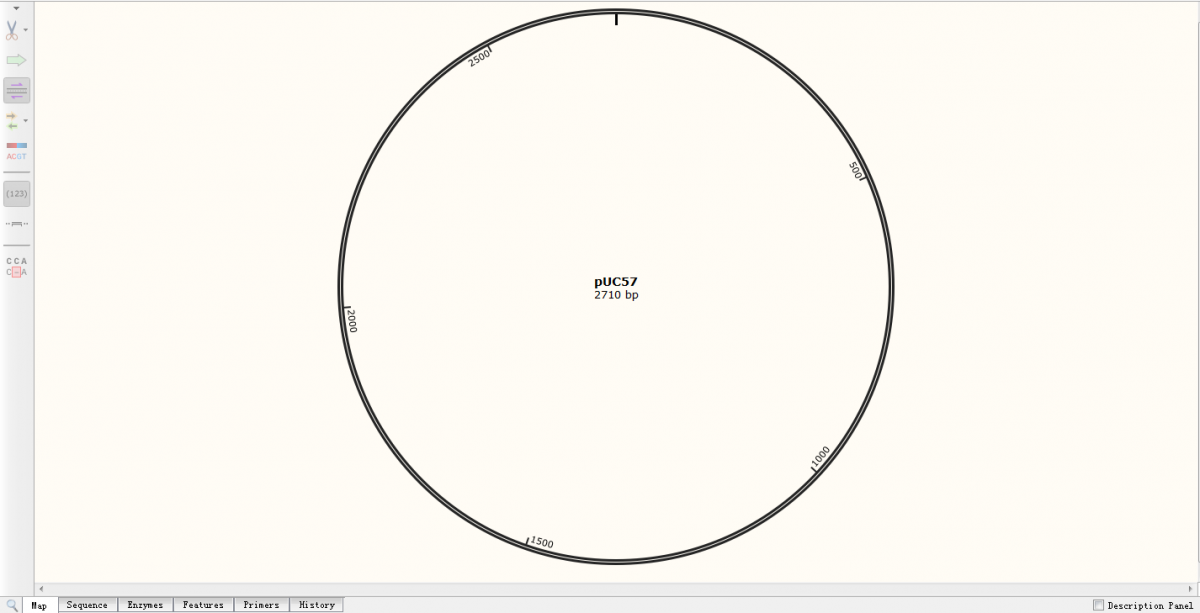
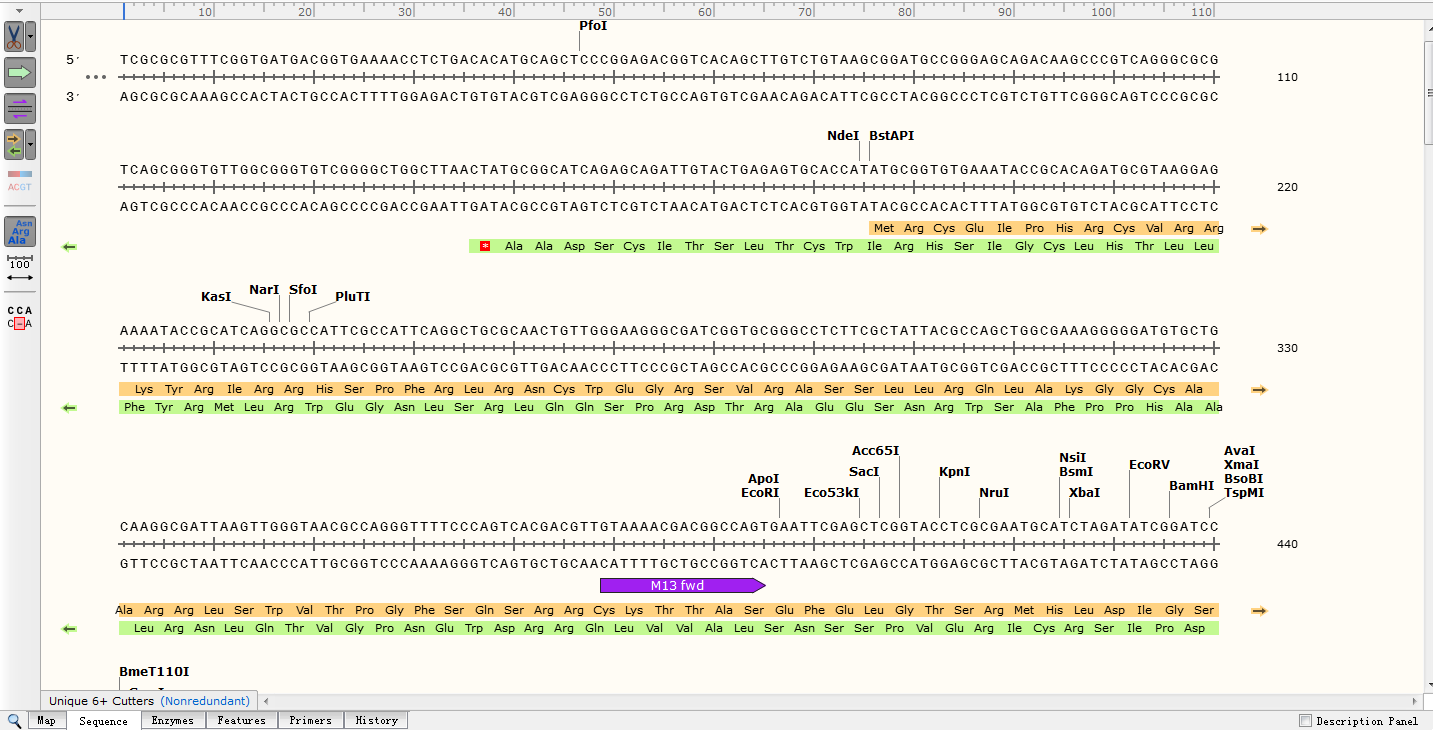
视图二:Sequence点击 Sequence 按钮,得到如下界面:
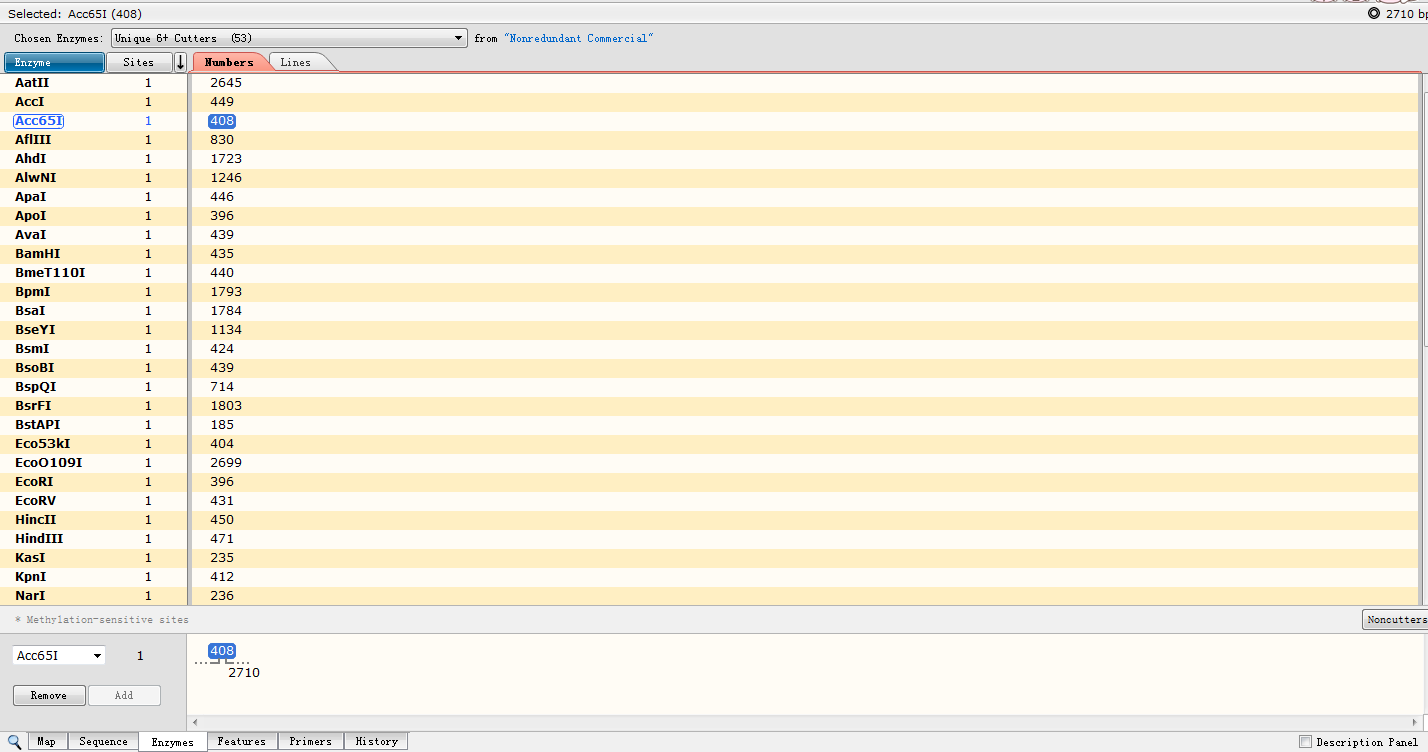
视图三:Enzymes点击 Enzymes 按钮,得到如下界面,可以看到不同酶切位点在序列中位置:
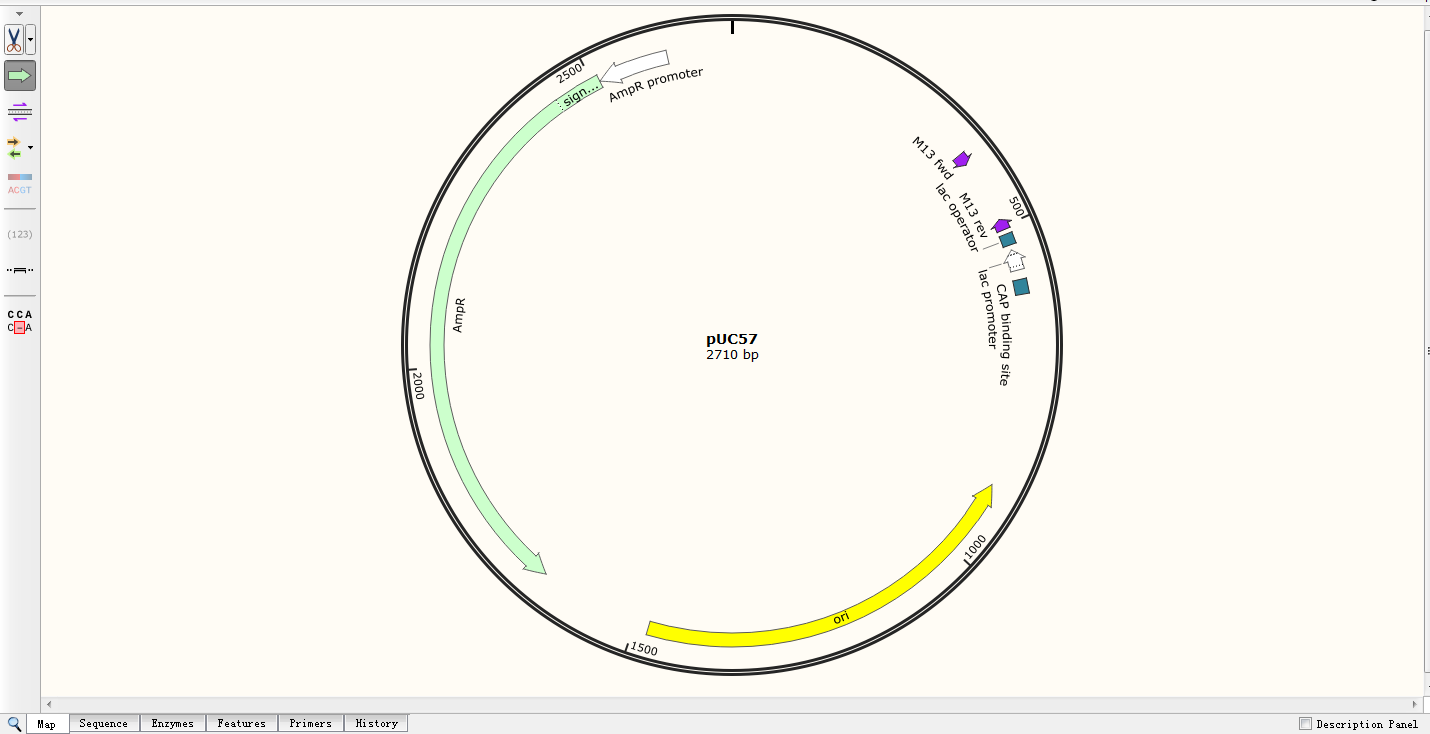
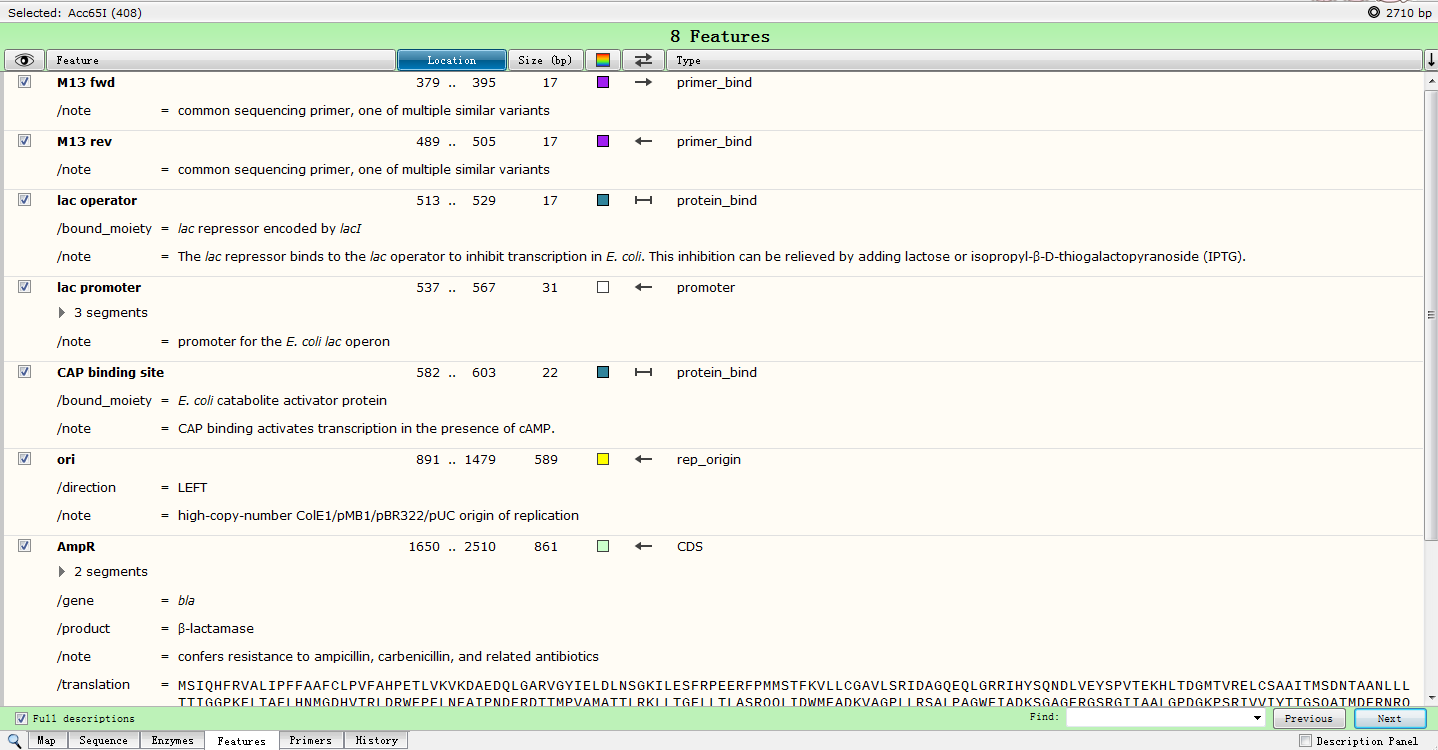
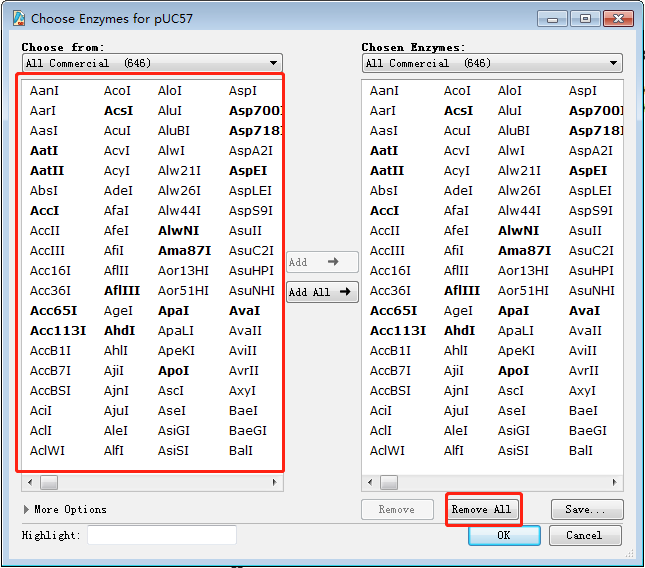
视图四:Features点击 Features 按钮,得到如下界面,显示一些常用元件(含元件的位置、大小、功能等信息): 这次我们所讲的这些功能,主要是在最上方的菜单栏进行操作完成:一、批量添加不同的酶切位点1.点击菜单栏 Enzymes→ChooseEnzymes,可以有选择的显示不同的酶切位点。2.点击 RemoveAll,清空右侧的内切酶,在左侧框内选择需要显示的内切酶进行添加。 二、对片段进行命名1.在 sequence 视图,选中需要命名的序列,点击菜单栏 Feature→Add Feature,弹出以下窗口: 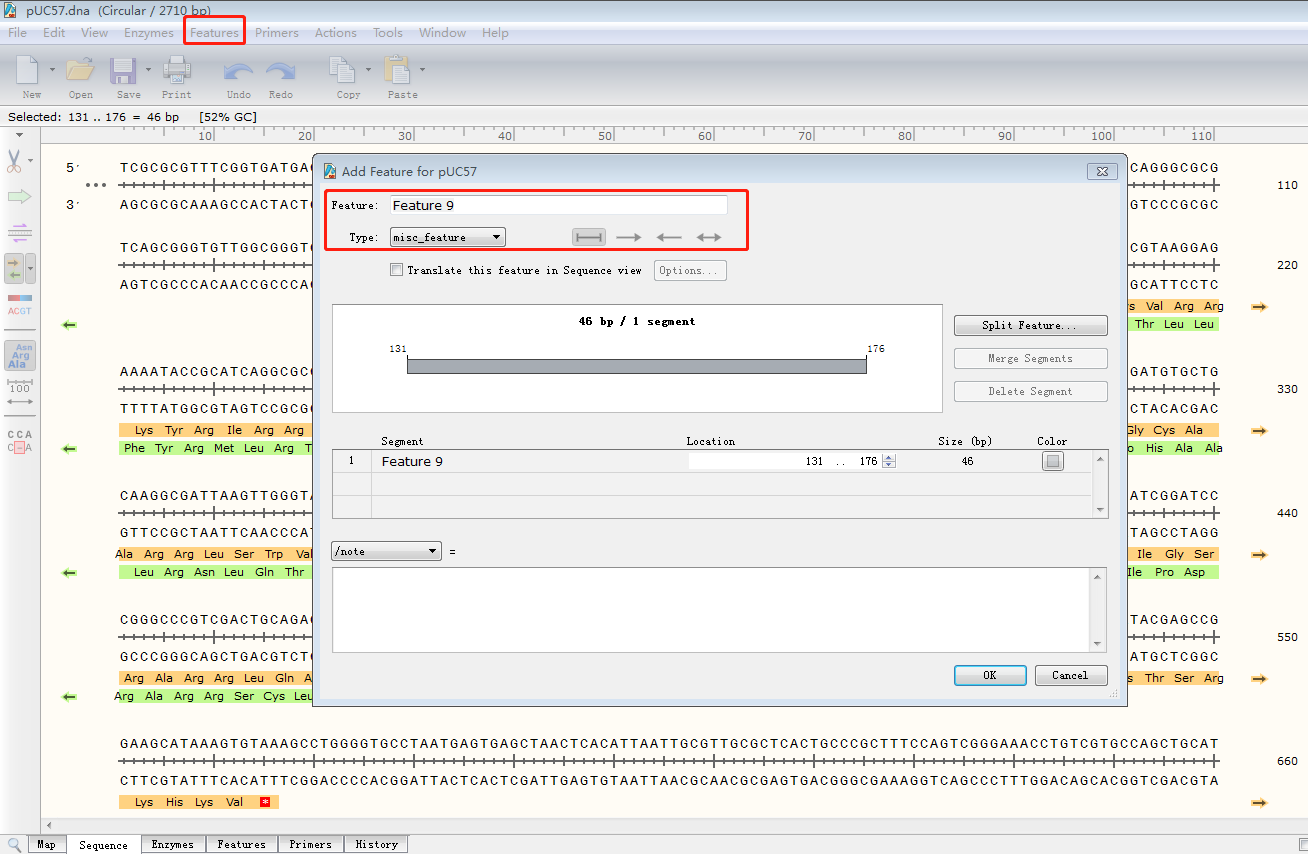
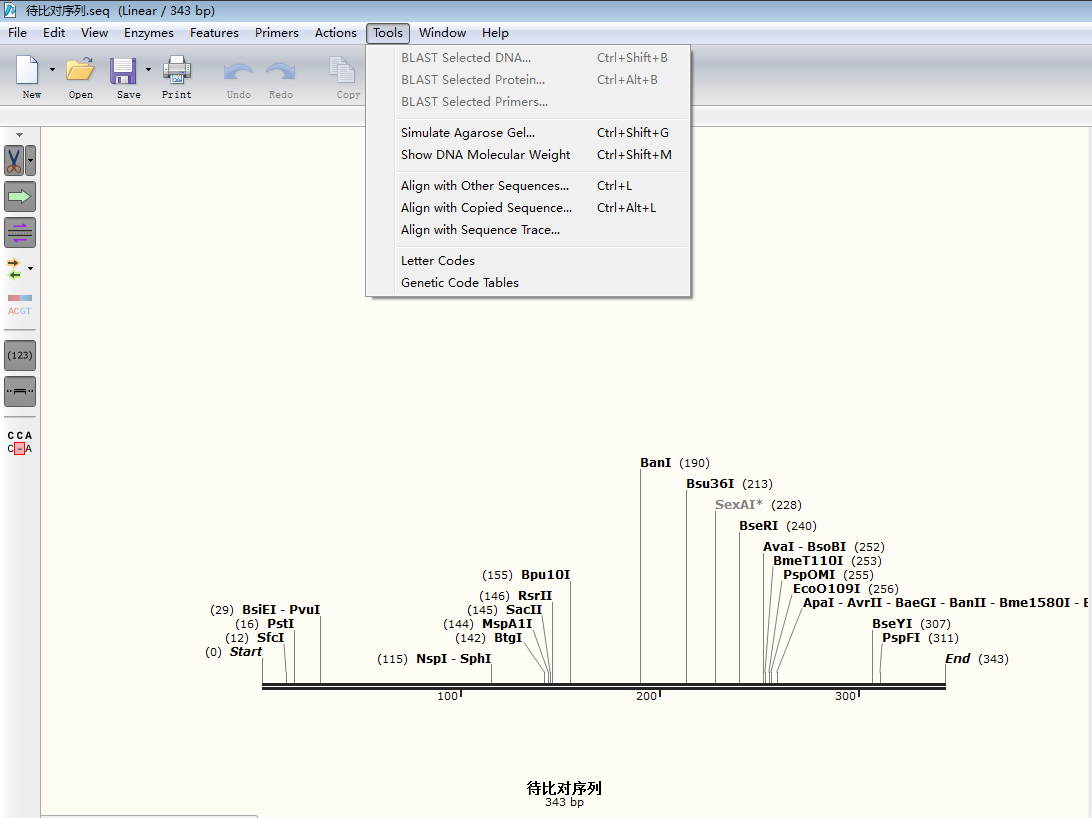
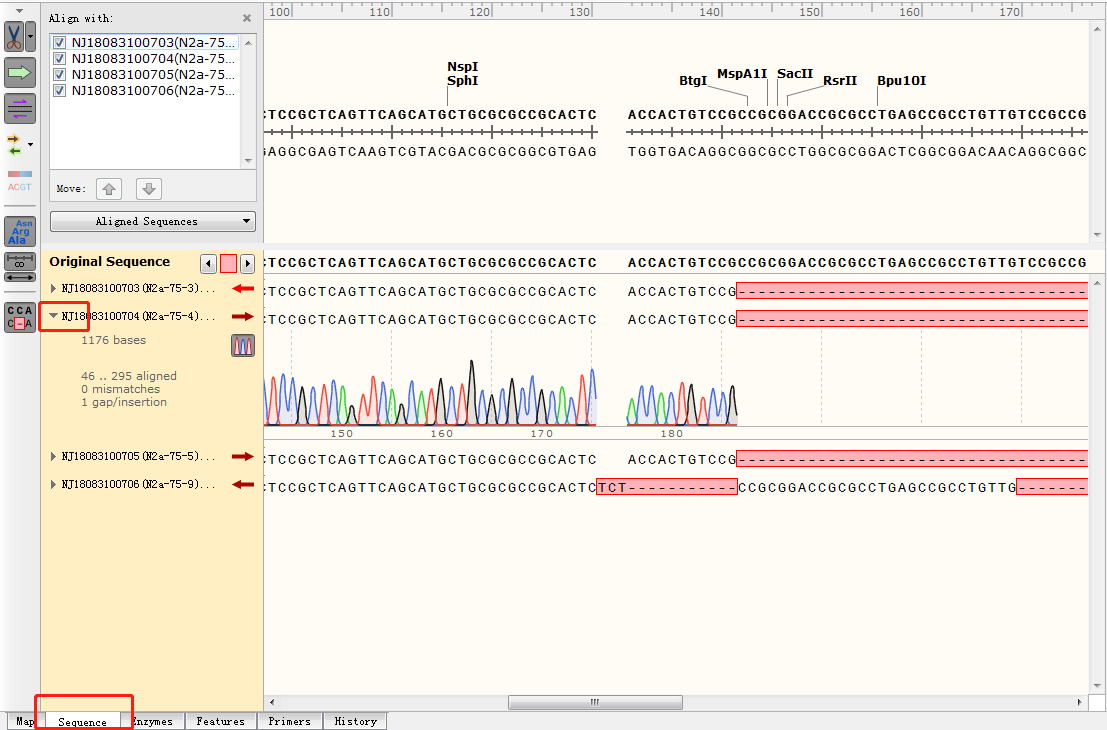
Feature:给该片段命名。 Type:选择该片段的类型,点击右侧箭头选择不同的阅读方向。 Color:选择颜色。 三、给质粒图谱增加引物序列1.点击菜单栏 Edit→Find(或 Ctrl+F),输入引物序列,找到质粒序列对应位置。2.点击 Primers→Addprimer,弹出该界面: 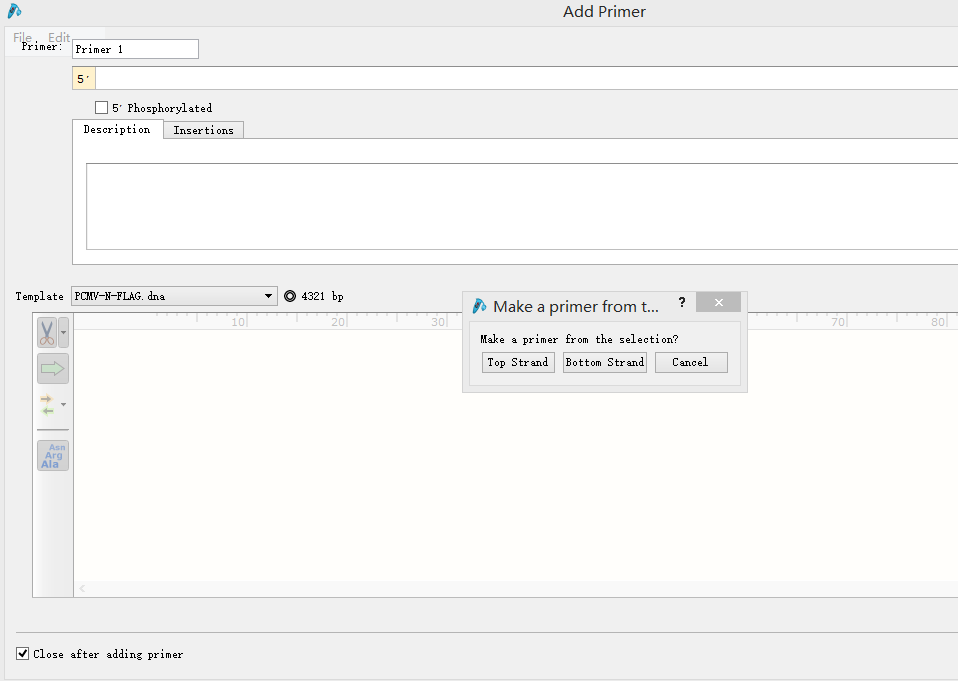 3.按上下游引物选择 TopStrand 还是 BottomStrand,在 Primer 处可给该引物命名,随后即可显示该引物在图谱 Map 中的位置,也可以将常用的通用引物建到一个 txt 文档中,利用 importprimer from a list 进行添加。 3.按上下游引物选择 TopStrand 还是 BottomStrand,在 Primer 处可给该引物命名,随后即可显示该引物在图谱 Map 中的位置,也可以将常用的通用引物建到一个 txt 文档中,利用 importprimer from a list 进行添加。四、模拟标准限制性克隆1.打开被插入片段的质粒图谱,选择合适的两个酶切位点,如 xbal 和 EcoRV,点击菜单栏 Actions→InsertFragment,点击 xbal+Shift 键 +EcoRV。 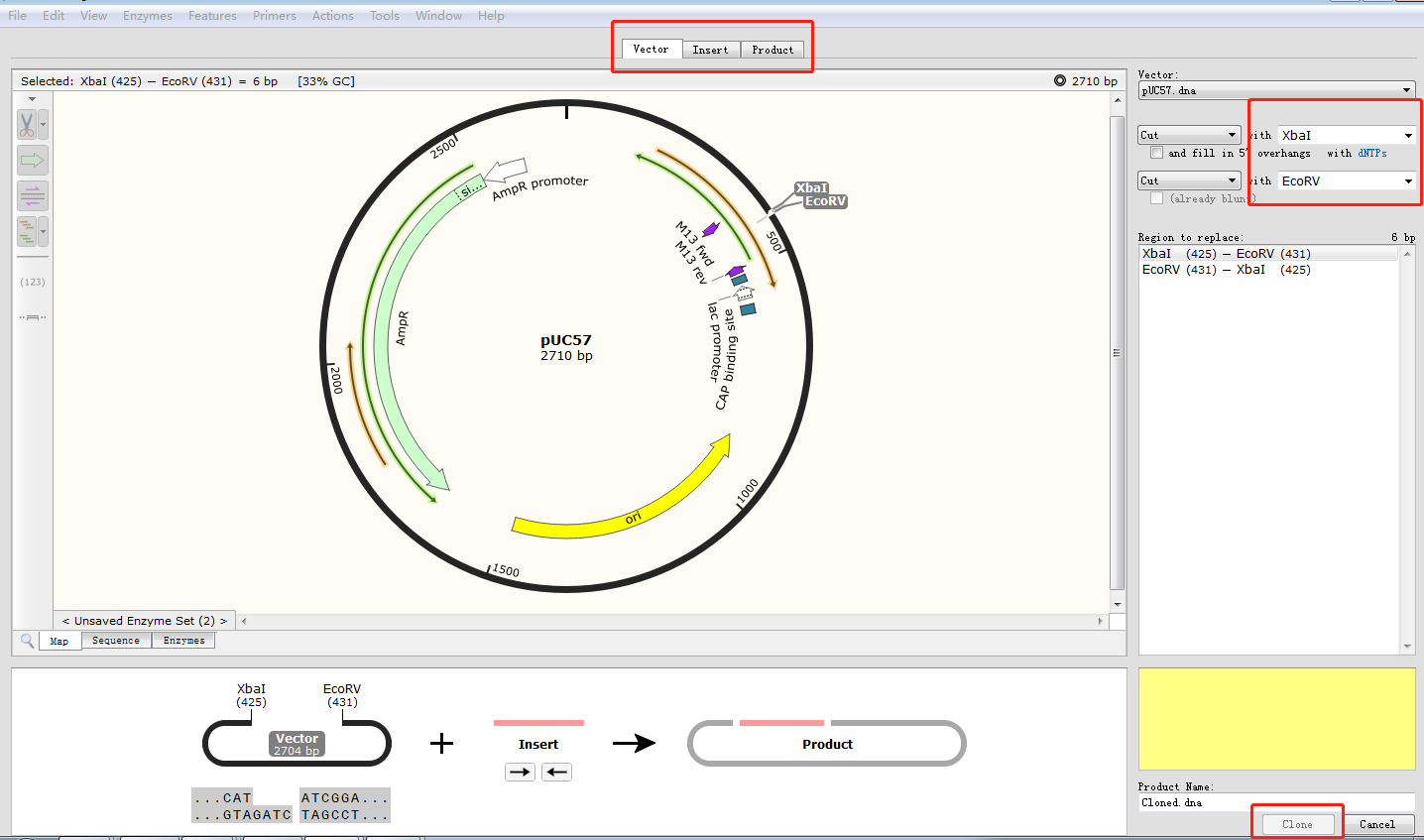 2.点击 Insert,在 sourceof fragment 处选择插入的目的片段来源。 2.点击 Insert,在 sourceof fragment 处选择插入的目的片段来源。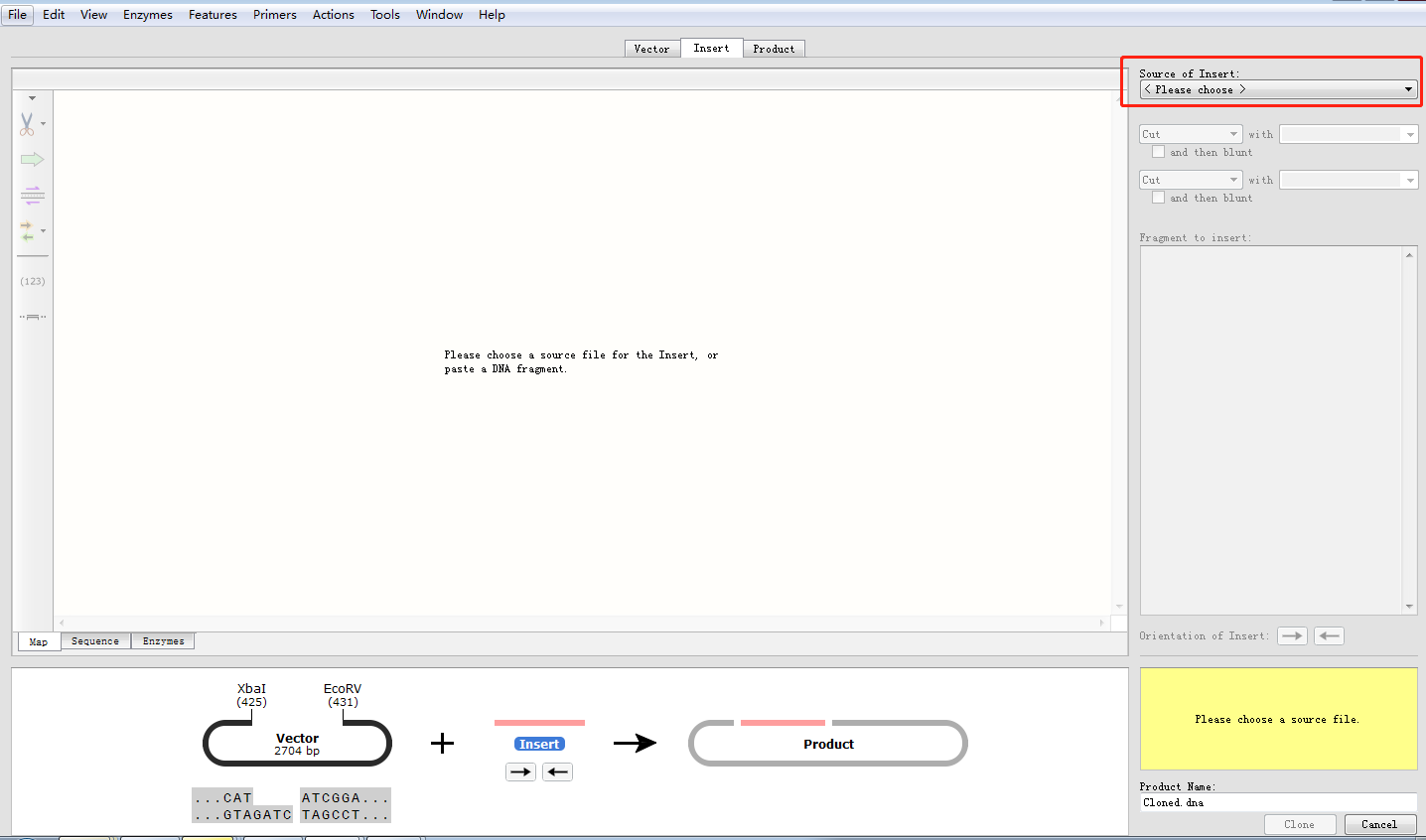 3.点击上述同样的酶,给该重组质粒命名,点击 clone。 3.点击上述同样的酶,给该重组质粒命名,点击 clone。五、模拟融合克隆1.打开一个需要插入片段质粒图谱,点击菜单栏 Actions→Insert One Fragments,弹出以下窗口: 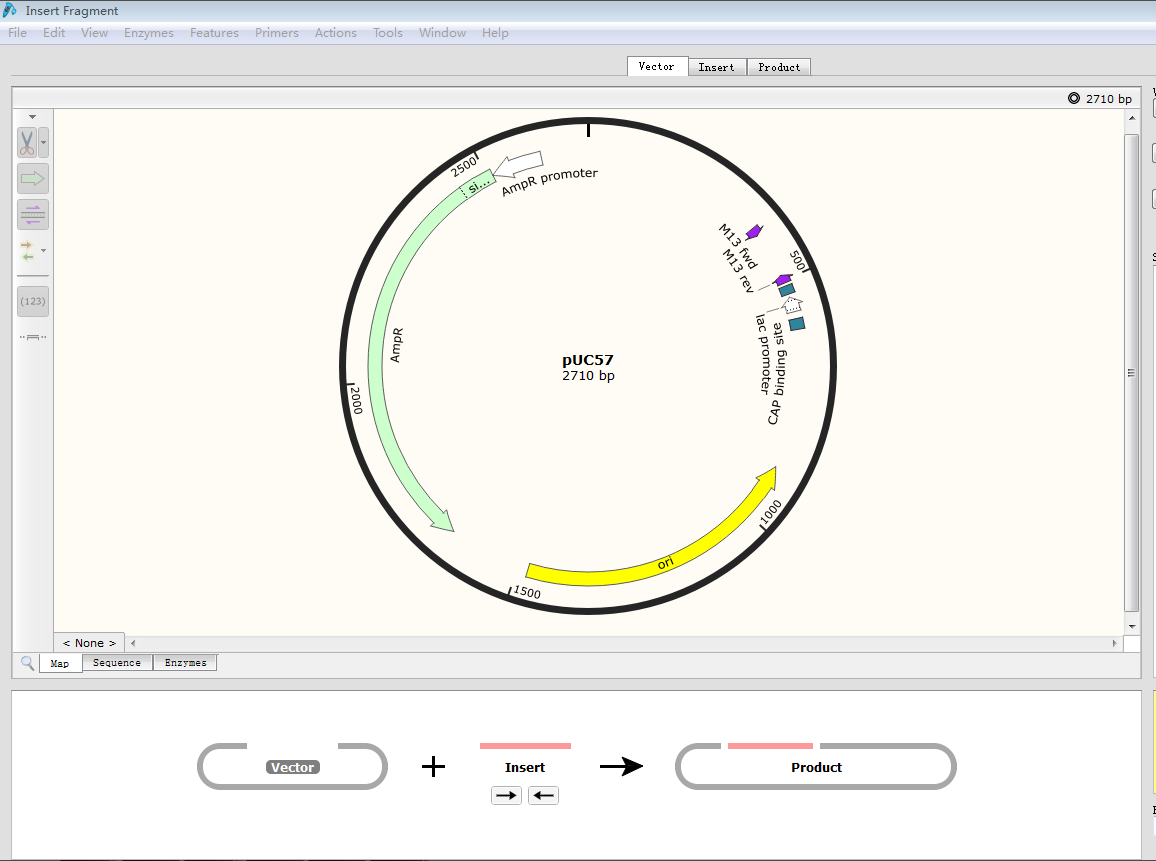 2.切换到 sequence 视图,找到想要发生替换的位点。3.点击 Insert,在 Source of Fragment 处选择拼接或替换片段的另外一个质粒图谱。此时弹出另外一个质粒图谱图样。4.点击用于替换的片段,观察该片段阅读方向与被替换质粒位点的方向是否一致,如不一致,点击 2.切换到 sequence 视图,找到想要发生替换的位点。3.点击 Insert,在 Source of Fragment 处选择拼接或替换片段的另外一个质粒图谱。此时弹出另外一个质粒图谱图样。4.点击用于替换的片段,观察该片段阅读方向与被替换质粒位点的方向是否一致,如不一致,点击六、模拟电泳功能1.点击菜单栏 Tools→SimulateAgarose Gel,显示如下界面: 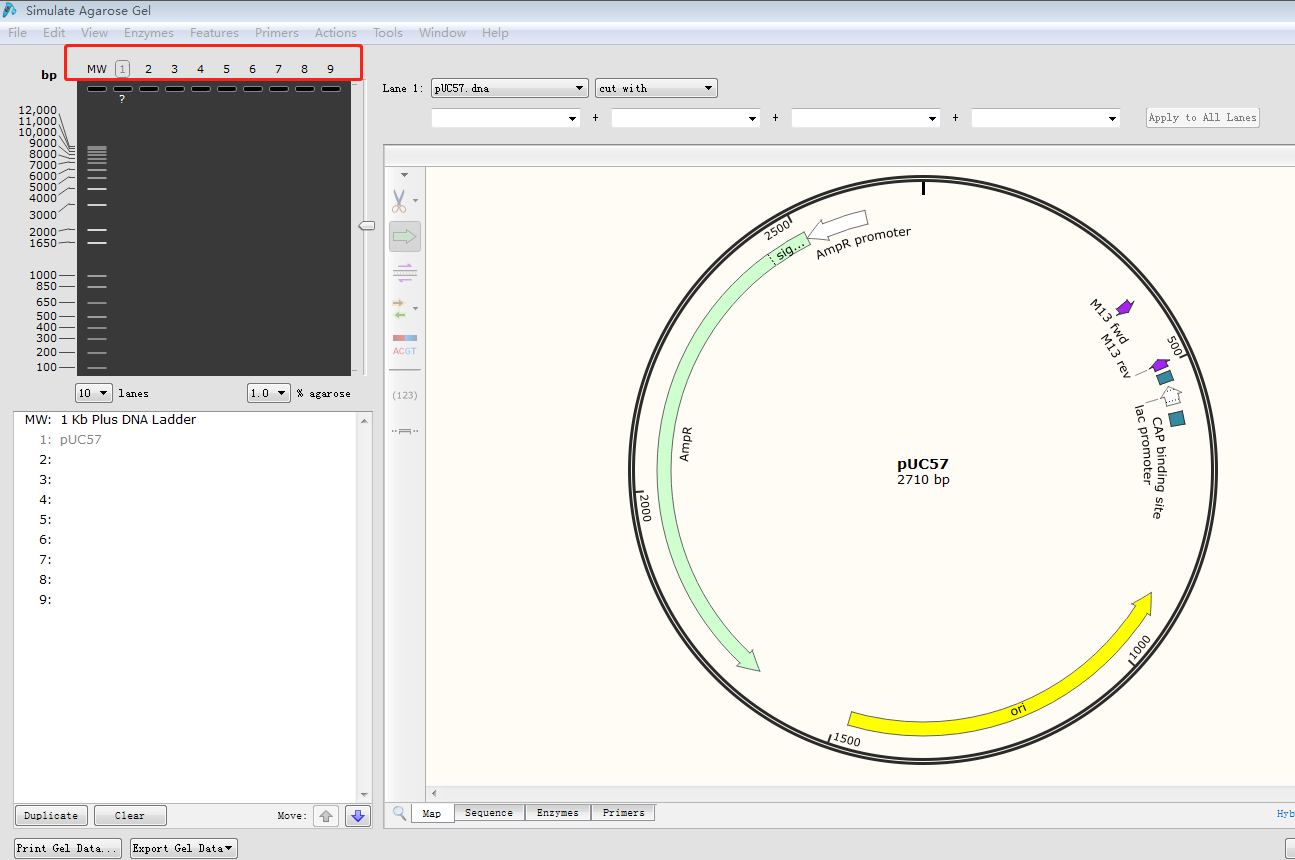 2.点击 MW 可以选择不同的 Marker,其中点击 1.2.3.4……分别代表不同的泳道,通过选择不同的酶切位点,模拟各个泳道的电泳结果。3.以第一泳道为例:点击选中 line1,选择 BanI、BfaI 两个酶切位点,得到模拟电泳结果,如下图: 2.点击 MW 可以选择不同的 Marker,其中点击 1.2.3.4……分别代表不同的泳道,通过选择不同的酶切位点,模拟各个泳道的电泳结果。3.以第一泳道为例:点击选中 line1,选择 BanI、BfaI 两个酶切位点,得到模拟电泳结果,如下图: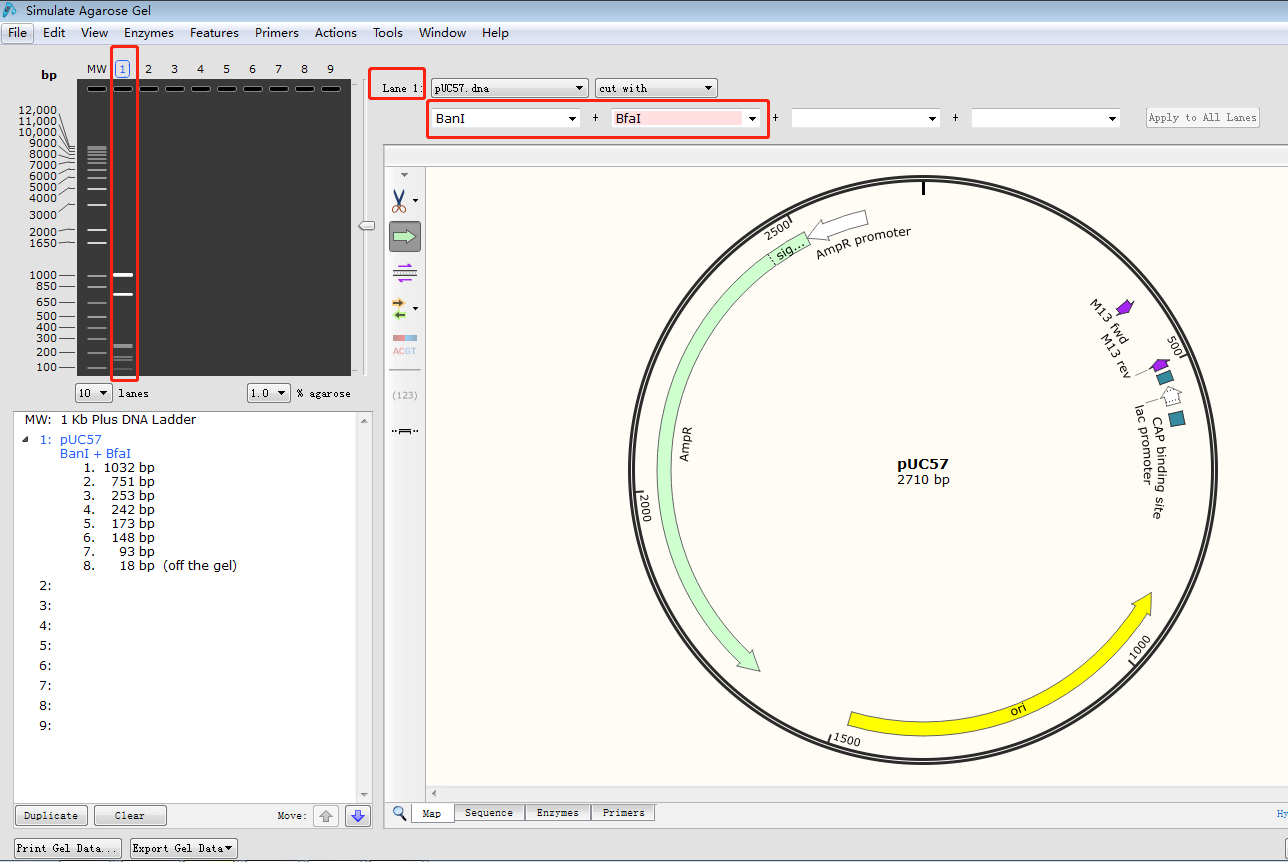 七、序列比对功能1.打开一个原始待比对的序列片段,点击菜单栏 Tool→Alignwith other Sequences 或 Alignwith Copied Sequences,如下图所示: 2.在本地文件中将需要比对的序列峰图全部选中,点击打开 3.得到如下比对结果,点击 sequence 可以看出每个样品的碱基缺失情况:
|
【本文地址】