Seurat 单细胞转录组测序数据分析教程(三) |
您所在的位置:网站首页 › 有序分类变量 › Seurat 单细胞转录组测序数据分析教程(三) |
Seurat 单细胞转录组测序数据分析教程(三)
|
Seurat 单细胞转录组测序数据分析教程(二)——python(scanpy)
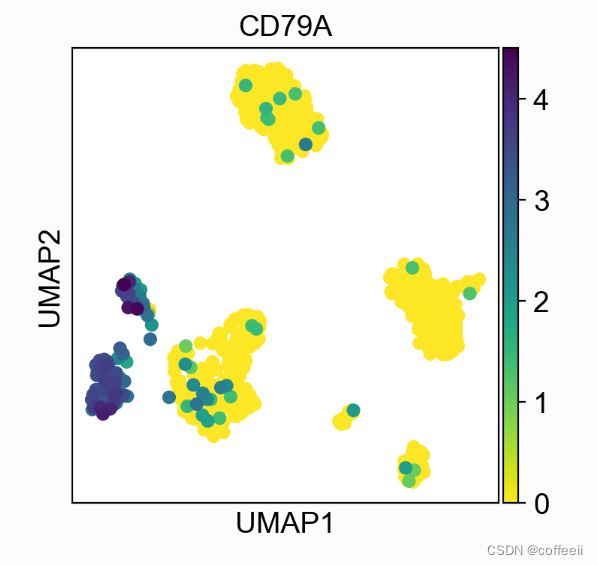
文章参考至scanpy官网,做了一个更详细的解读。 本教程探讨了 scanpy 的可视化可能性,分为三个部分: 嵌入的散点图(例如 UMAP、t-SNE) 使用已知标记基因鉴定簇 差异表达基因的可视化 在本教程中,我们将使用来自 10x 的数据集,其中包含来自PBMC的 68k 个细胞。Scanpy 在其分布中包括该数据集的简化样本,该数据集仅包含 700 个细胞和 765 个高度可变的基因。该数据集已经过预处理和 UMAP 计算。 在本教程中,我们还将使用以下文献标记: B细胞:CD79A、MS4A1 等离子: IGJ (JCHAIN) T细胞:CD3D NK:GNLY,NKG7 髓系:CST3、LYZ 单核细胞:FCGR3A 树突状:FCER1A 嵌入的散点图 import scanpy as sc import pandas as pd from matplotlib.pyplot import rc_context sc.set_figure_params(dpi=100, color_map = 'viridis_r') sc.settings.verbosity = 1 sc.logging.print_header() 加载 pbmc 数据集 pbmc = sc.datasets.pbmc68k_reduced() # inspect pbmc contents pbmc 基因表达和其他变量的可视化对于散点图,要绘制的值作为参数给出color。这可以是任何基因或 中的任何列.obs,其中.obs是包含每个观察/细胞注释的 DataFrame,请参阅AnnData了解更多信息。 # rc_context is used for the figure size, in this case 4x4 with rc_context({'figure.figsize': (4, 4)}): sc.pl.umap(pbmc, color='CD79A')
此外,我们将绘制另外两个值:n_counts这是每个单元格的 UMI 计数(存储在 中.obs),bulk_labels这是一个包含来自 10X 的单元格原始标记的分类值。 每行的图数由ncols参数控制。可以使用调整绘制的最大值vmax(同样vmin可以用于最小值)。在这种情况下,我们使用p99,这意味着使用 99 个百分位数作为最大值。如果 vmax 想要分别为多个地块设置,则最大值可以是数字或数字列表。 此外,我们正在使用frameon=False删除图周围的框并s=50设置点大小。 with rc_context({'figure.figsize': (3, 3)}): sc.pl.umap(pbmc, color=['CD79A', 'MS4A1', 'IGJ', 'CD3D', 'FCER1A', 'FCGR3A', 'n_counts', 'bulk_labels'], s=50, frameon=False, ncols=4, vmax='p99')
散点图的函数有很多选项,可以对图像进行微调。例如,我们可以按如下方式查看聚类: # compute clusters using the leiden method and store the results with the name `clusters` sc.tl.leiden(pbmc, key_added='clusters', resolution=0.5) with rc_context({'figure.figsize': (5, 5)}): sc.pl.umap(pbmc, color='clusters', add_outline=True, legend_loc='on data', legend_fontsize=12, legend_fontoutline=2,frameon=False, title='clustering of cells', palette='Set1')
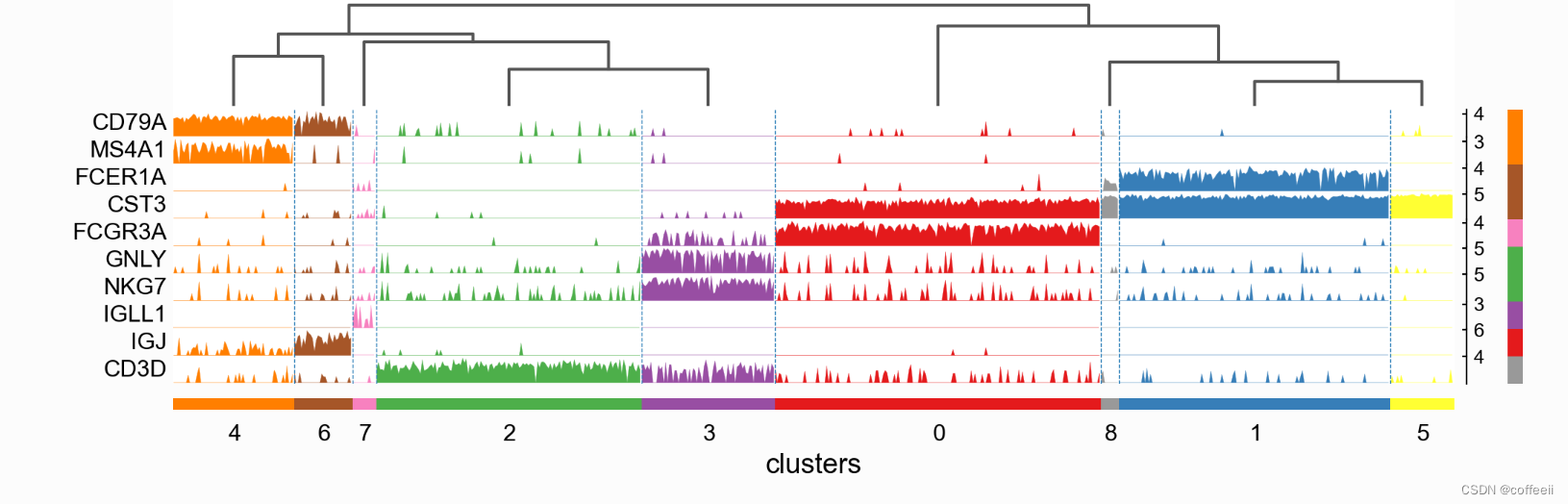
通常,需要使用众所周知的标记基因来标记簇。使用散点图,我们可以看到一个基因的表达,并可能将它与一个簇联系起来。在这里,我们将展示使用点图、小提琴图、热图和我们称之为“轨迹图”的东西将标记基因与聚类相关联的其他视觉方法。所有这些可视化都总结了相同的信息,按集群划分的表达,最佳结果的选择留给研究者决定。 首先,我们建立一个带有标记基因的字典,因为这将允许 scanpy 自动标记基因组: marker_genes_dict = { 'B-cell': ['CD79A', 'MS4A1'], 'Dendritic': ['FCER1A', 'CST3'], 'Monocytes': ['FCGR3A'], 'NK': ['GNLY', 'NKG7'], 'Other': ['IGLL1'], 'Plasma': ['IGJ'], 'T-cell': ['CD3D'], } 点图检查每个簇中这些基因表达的一种快速方法是使用点图。这种类型的图总结了两种类型的信息:颜色代表每个类别中的平均表达(在这种情况下是每个聚类),点大小表示类别中表达基因的细胞比例。 此外,向图中添加树状图以将相似的聚类聚集在一起也很有用。层次聚类是使用聚类之间 PCA 组件的相关性自动计算的。 sc.pl.dotplot(pbmc, marker_genes_dict, 'clusters', dendrogram=True)
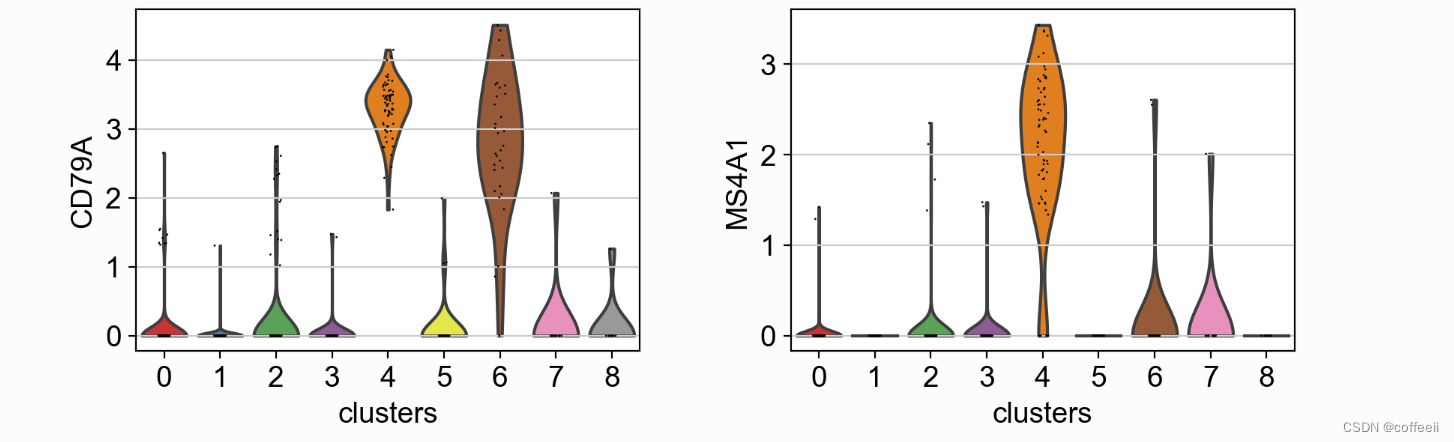
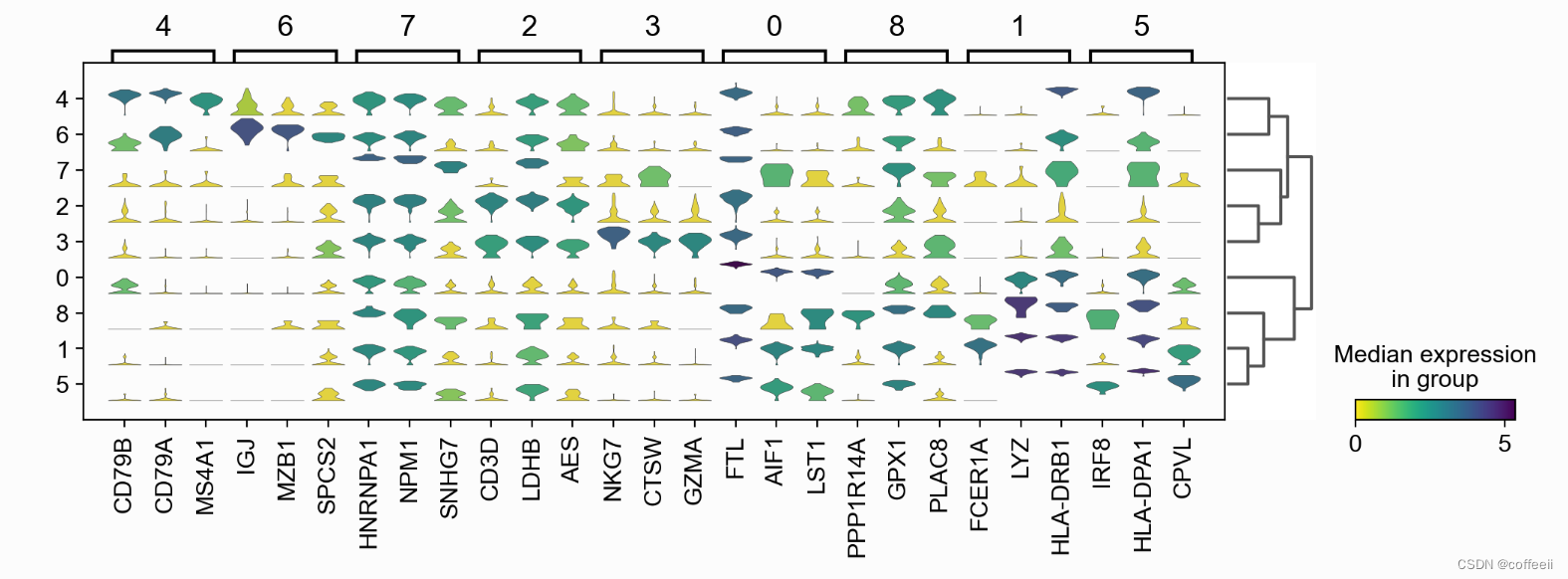
探索标记的另一种方法是使用小提琴图。在这里,我们可以在簇 5 和 8 中看到CD79A的表达,在簇 5 中看到MS4A1。与点图相比,小提琴图让我们了解了基因表达值在细胞之间的分布。 with rc_context({'figure.figsize': (4.5, 3)}): sc.pl.violin(pbmc, ['CD79A', 'MS4A1'], groupby='clusters' )
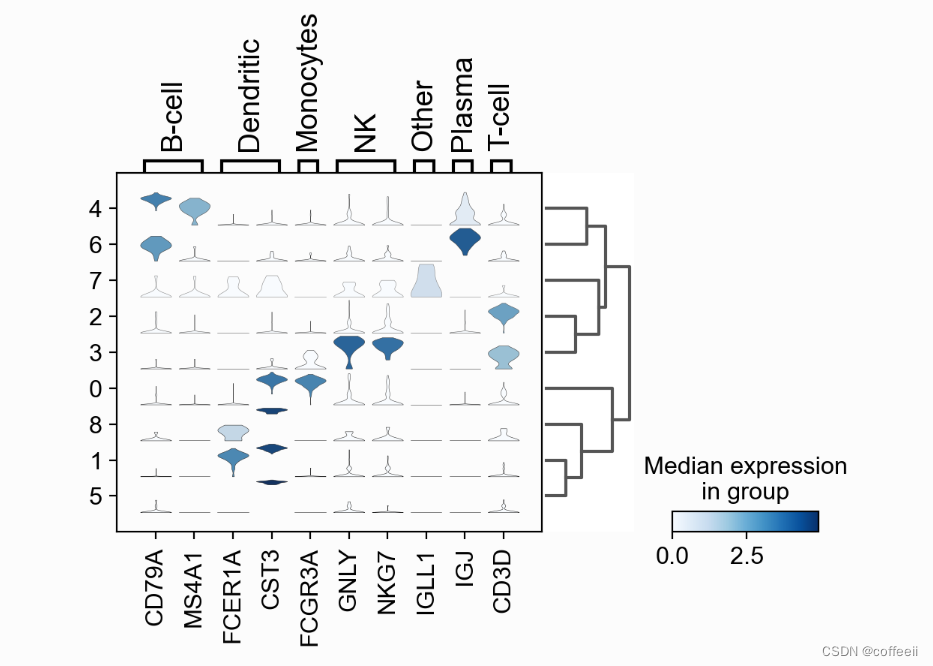
同时查看我们使用的所有标记基因的小提琴图sc.pl.stacked_violin。和以前一样,添加了一个树状图来对相似的集群进行分组 ax = sc.pl.stacked_violin(pbmc, marker_genes_dict, groupby='clusters', swap_axes=False, dendrogram=True)
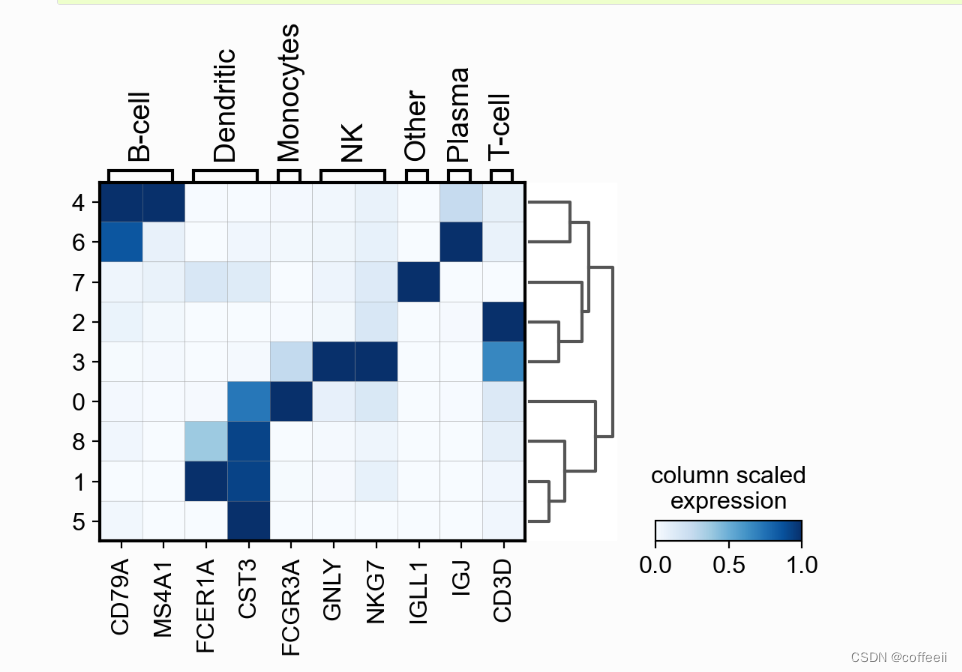
可视化基因表达的一种简单方法是使用. 这是按类别分组的每个基因的平均表达值的热图。这种类型的图基本上显示了与点图中颜色相同的信息。matrix plot 在这里,将基因的表达从 0 缩放到 1,即最大平均表达和 0 最小值。 sc.pl.matrixplot(pbmc, marker_genes_dict, 'clusters', dendrogram=True, cmap='Blues', standard_scale='var', colorbar_title='column scaled\nexpression')
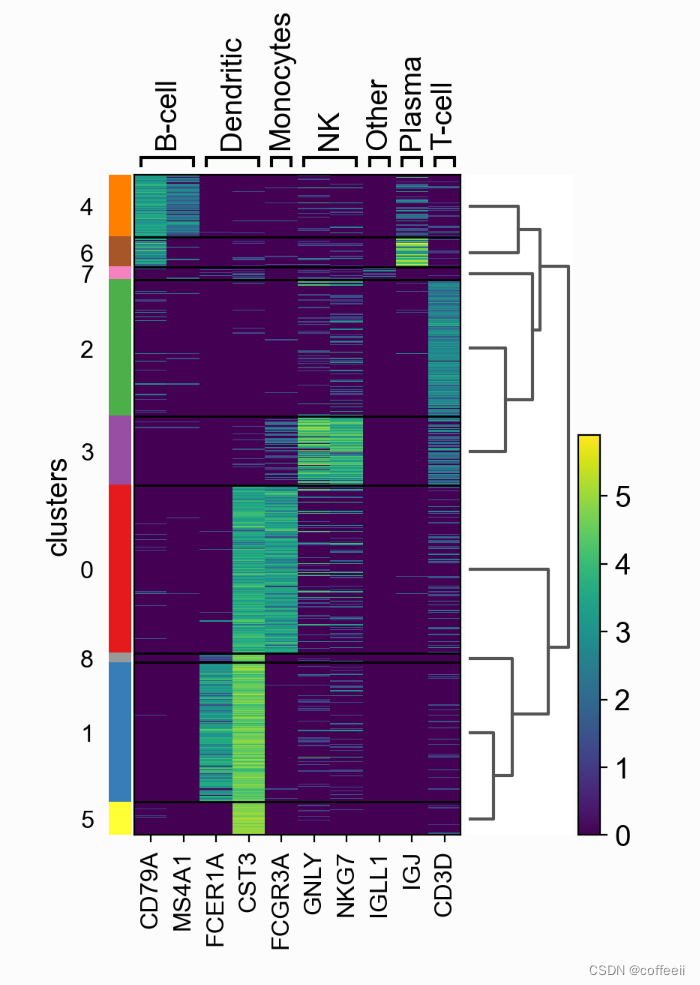
热图不会像以前的图中那样折叠单元格。相反,每个单元格都显示在一行中(如果是,则显示在列中swap_axes=True)。可以添加 groupby 信息,并使用为或任何其他嵌入找到的相同颜色代码显示sc.pl.umap。 ax = sc.pl.heatmap(pbmc, marker_genes_dict, groupby='clusters', cmap='viridis', dendrogram=True)
热图也可以绘制在缩放数据上。在下一张图片中,与之前的矩阵图类似,调整了最小值和最大值,并使用了发散色图。 ax = sc.pl.heatmap(pbmc, marker_genes_dict, groupby='clusters', layer='scaled', vmin=-2, vmax=2, cmap='RdBu_r', dendrogram=True, swap_axes=True, figsize=(11,4))
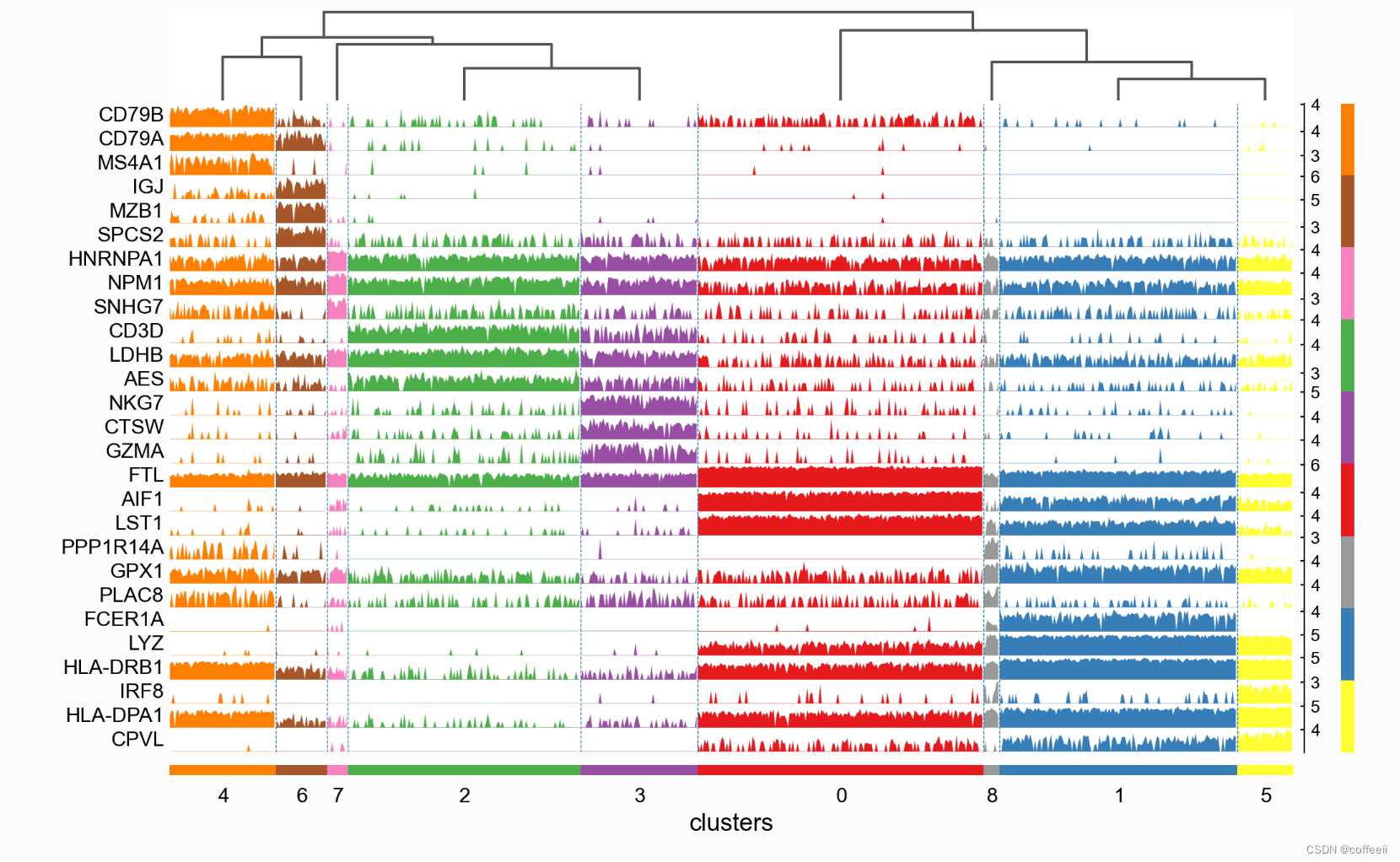
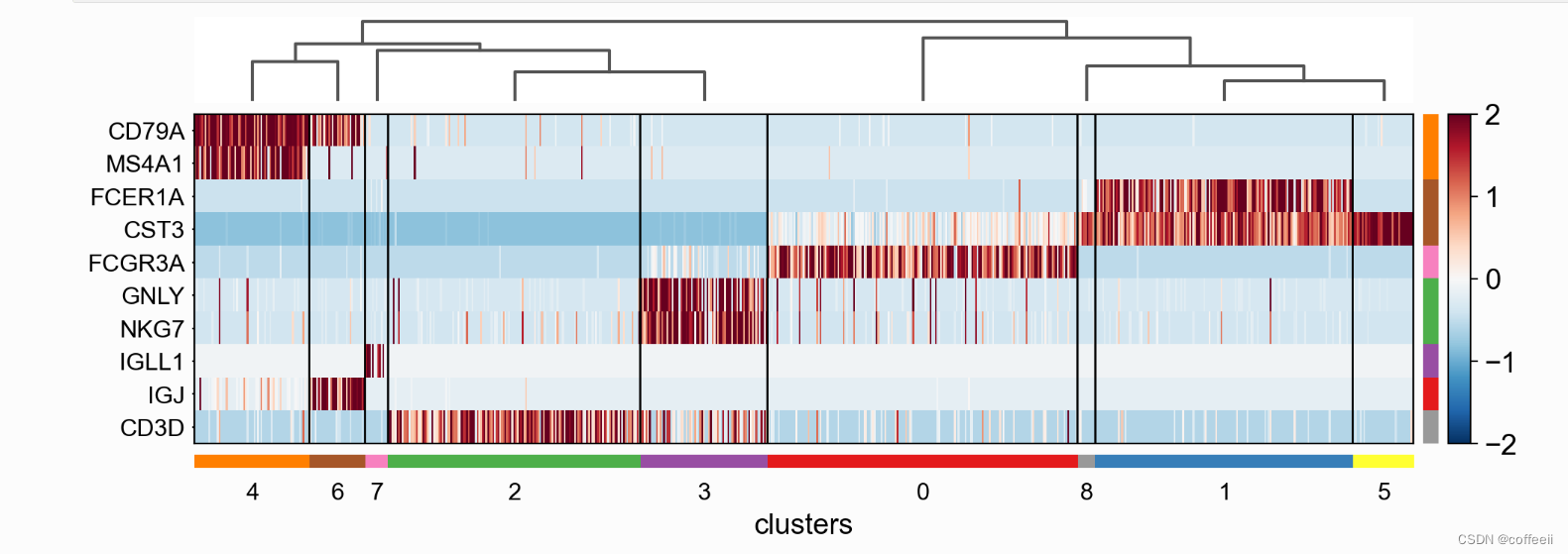
轨迹图显示与热图相同的信息,但基因表达由高度表示,而不是色标。 ax = sc.pl.tracksplot(pbmc, marker_genes_dict, groupby='clusters', dendrogram=True)
我们可以识别在簇或组中差异表达的基因,而不是像以前那样通过已知基因标记来表征簇。 为了识别我们运行的差异表达基因sc.tl.rank_genes_groups。该函数将获取每组细胞并将一组中每个基因的分布与不在该组中的所有其他细胞中的分布进行比较。在这里,我们将使用 10x 给出的原始细胞标签来识别这些细胞类型的标记基因。 sc.tl.rank_genes_groups(pbmc, groupby='clusters', method='wilcoxon') 使用点图可视化标记基因点图可视化有助于了解显示差异表达的基因的概况。为了使生成的图像更紧凑,我们将使用n_genes=4仅显示前 4 个得分基因。 sc.pl.rank_genes_groups_dotplot(pbmc, n_genes=4)
在这种情况下,我们设置values_to_plot='logfoldchanges’和min_logfoldchange=3。 因为对数倍数变化是发散尺度,我们还调整要绘制的最小值和最大值并使用发散色图。请注意,在下图中很难区分 T 细胞群。 sc.pl.rank_genes_groups_dotplot(pbmc, n_genes=4, values_to_plot='logfoldchanges', min_logfoldchange=3, vmax=7, vmin=-7, cmap='bwr') 专注于特定群体接下来,我们使用仅关注两个组的点图(组选项也可用于小提琴图、热图和矩阵图)。在这里,我们设置为在这种情况下它将显示所有具有最多 30 个的n_genes=30基因。min_logfoldchange=4 sc.pl.rank_genes_groups_dotplot(pbmc, n_genes=30, values_to_plot='logfoldchanges', min_logfoldchange=4, vmax=7, vmin=-7, cmap='bwr', groups=['1', '5'])
对于下图,我们使用先前计算的“缩放”值(存储在图层中scaled)并使用发散色图。\ sc.pl.rank_genes_groups_matrixplot(pbmc, n_genes=3, use_raw=False, vmin=-3, vmax=3, cmap='bwr', layer='scaled')
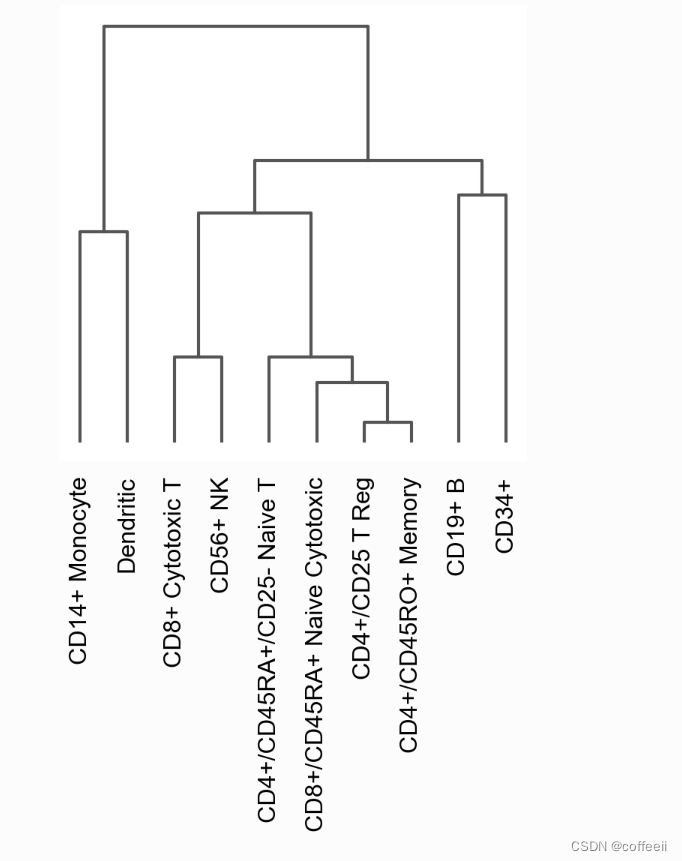
大多数可视化可以使用树状图来排列类别。然而,树状图也可以独立绘制如下: # compute hierarchical clustering using PCs (several distance metrics and linkage methods are available). sc.tl.dendrogram(pbmc, 'bulk_labels') ax = sc.pl.dendrogram(pbmc, 'bulk_labels')
|
【本文地址】
今日新闻 |
推荐新闻 |
 可以给 多个值color。在下面的示例中,我们将绘制 6 个基因:‘CD79A’、‘MS4A1’、‘IGJ’、CD3D’、‘FCER1A’ 和 ‘FCGR3A’ 以了解这些标记基因的表达位置。
可以给 多个值color。在下面的示例中,我们将绘制 6 个基因:‘CD79A’、‘MS4A1’、‘IGJ’、CD3D’、‘FCER1A’ 和 ‘FCGR3A’ 以了解这些标记基因的表达位置。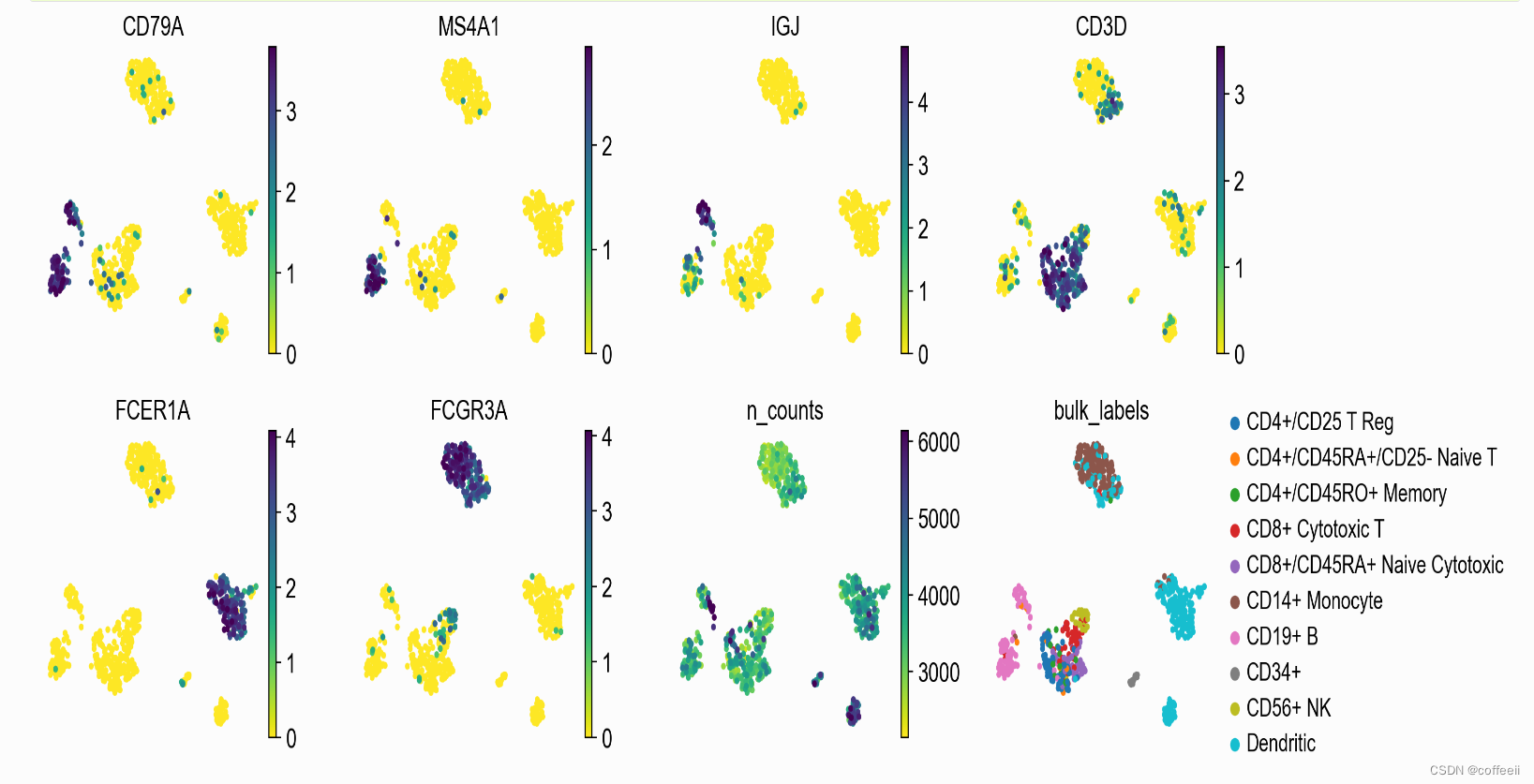 在此图中,我们可以看到表达标记基因的细胞组以及与原始细胞标签的一致性。
在此图中,我们可以看到表达标记基因的细胞组以及与原始细胞标签的一致性。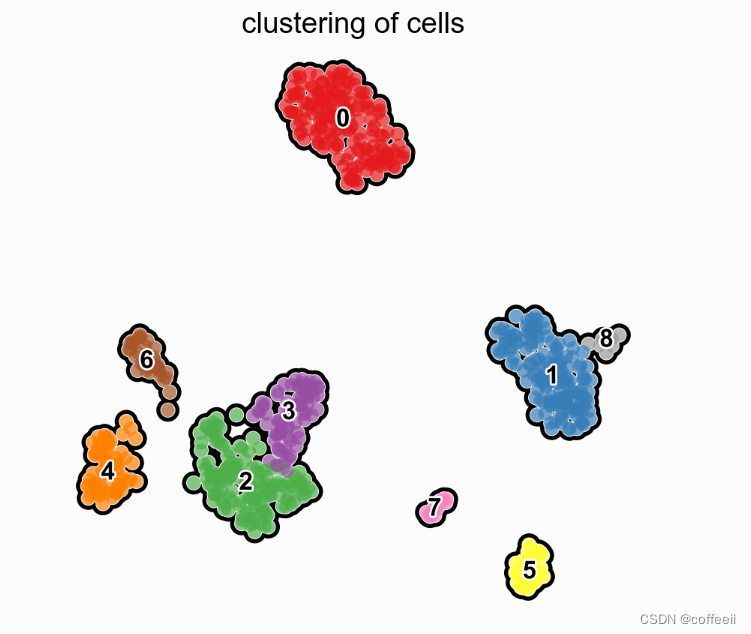
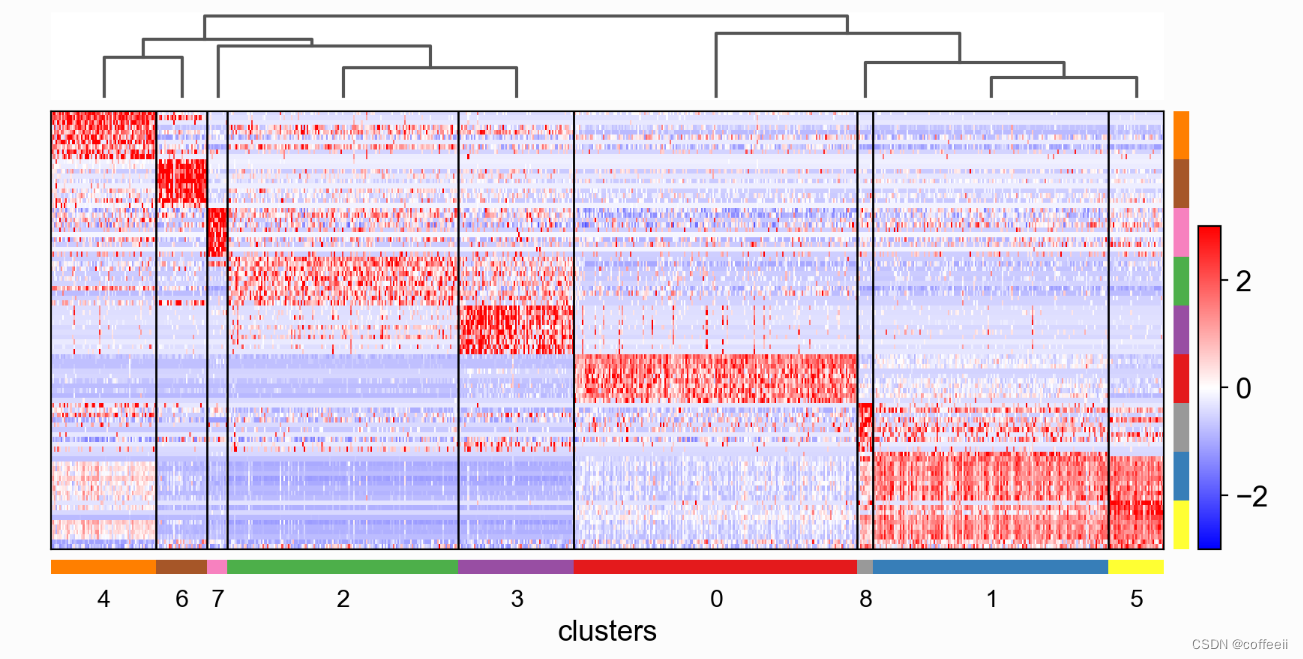
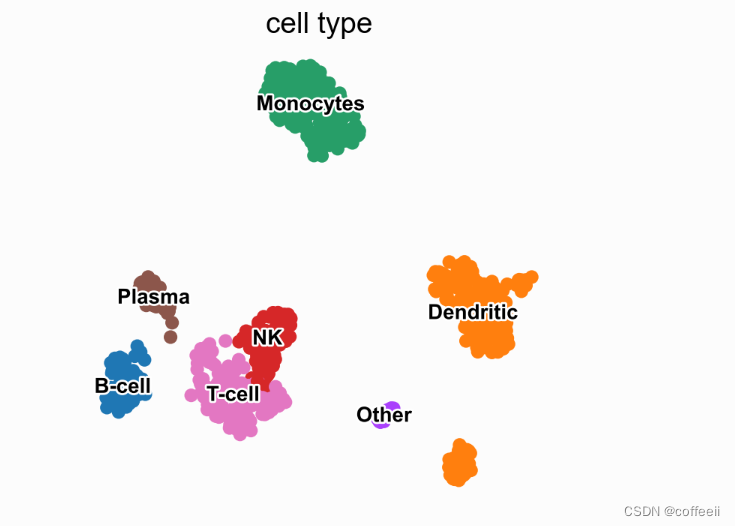
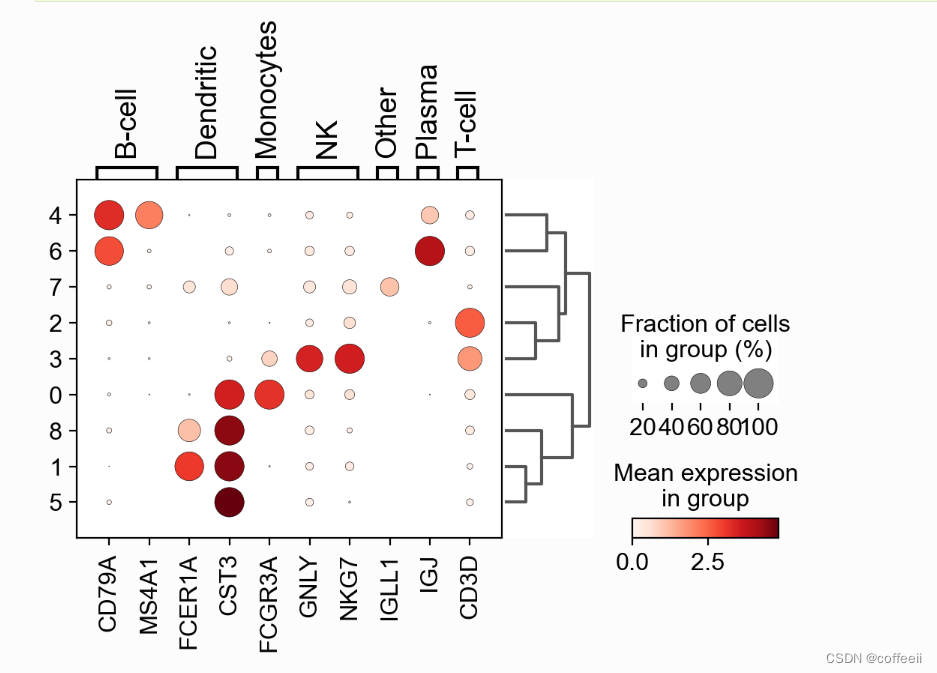 使用此图,我们可以看到簇 4 对应于 B 细胞,簇 2 对应于 T 细胞等。此信息可用于手动注释细胞,如下所示:
使用此图,我们可以看到簇 4 对应于 B 细胞,簇 2 对应于 T 细胞等。此信息可用于手动注释细胞,如下所示: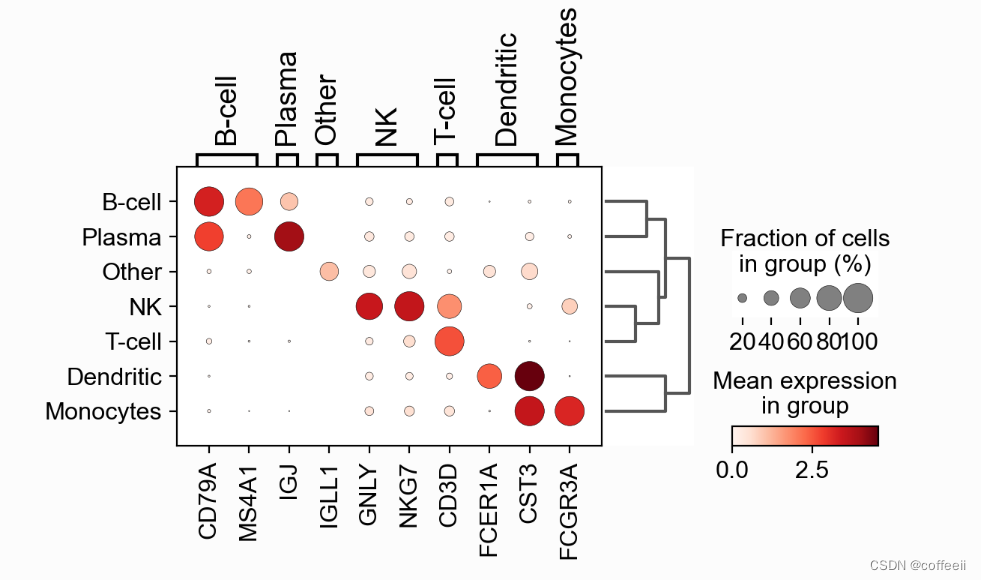
 其他有用的选项是使用sc.pp.scale. 在这里,我们将此信息存储在scale层下。之后我们调整绘图的最小值和最大值并使用发散的颜色图(在这种情况下,RdBu_r意思_r是相反的)。
其他有用的选项是使用sc.pp.scale. 在这里,我们将此信息存储在scale层下。之后我们调整绘图的最小值和最大值并使用发散的颜色图(在这种情况下,RdBu_r意思_r是相反的)。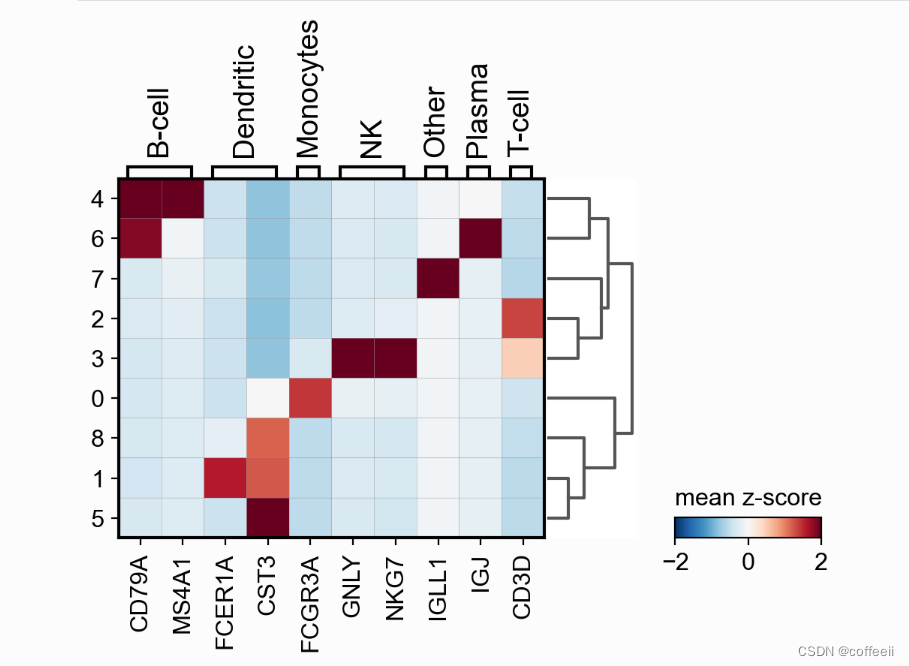
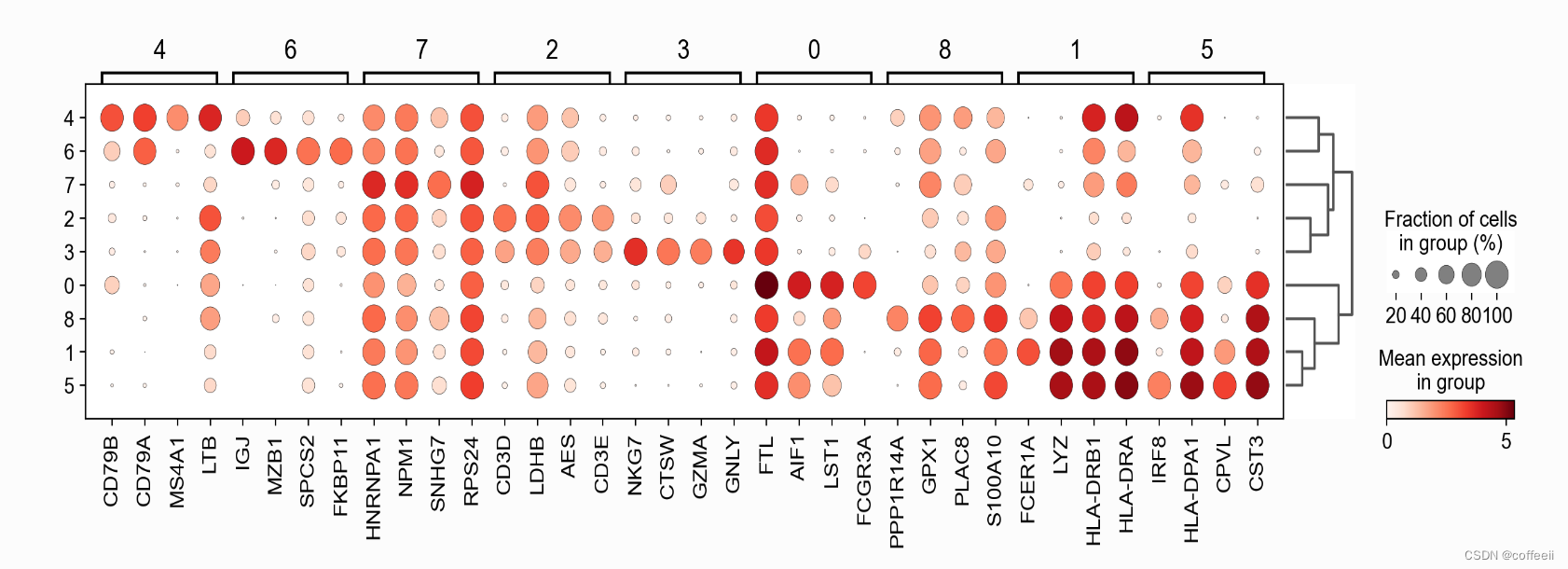 为了获得更好的表示,我们可以绘制对数倍数变化而不是基因表达。此外,我们希望关注在细胞类型表达和其余细胞之间具有对数倍数变化 >= 3 的基因。
为了获得更好的表示,我们可以绘制对数倍数变化而不是基因表达。此外,我们希望关注在细胞类型表达和其余细胞之间具有对数倍数变化 >= 3 的基因。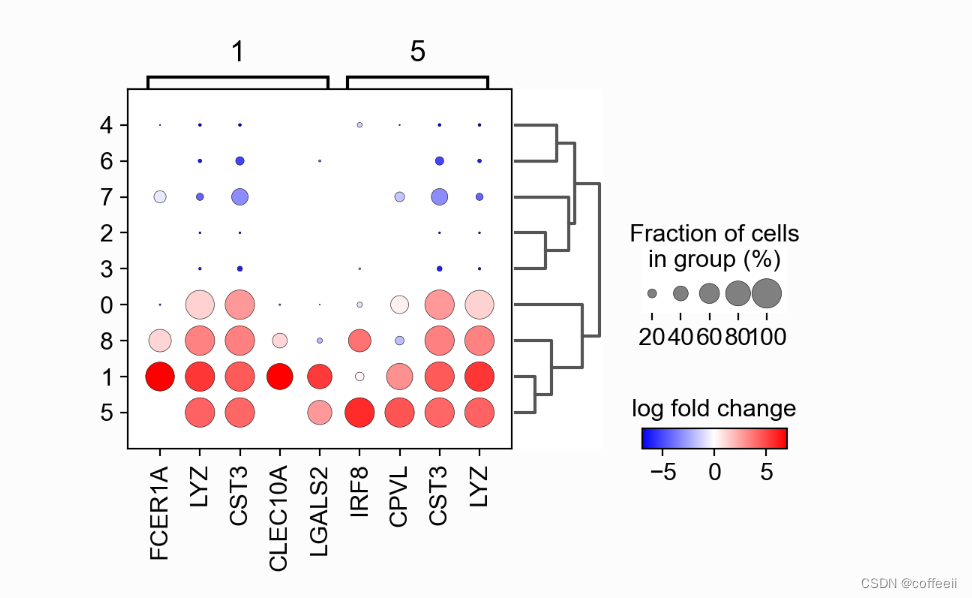
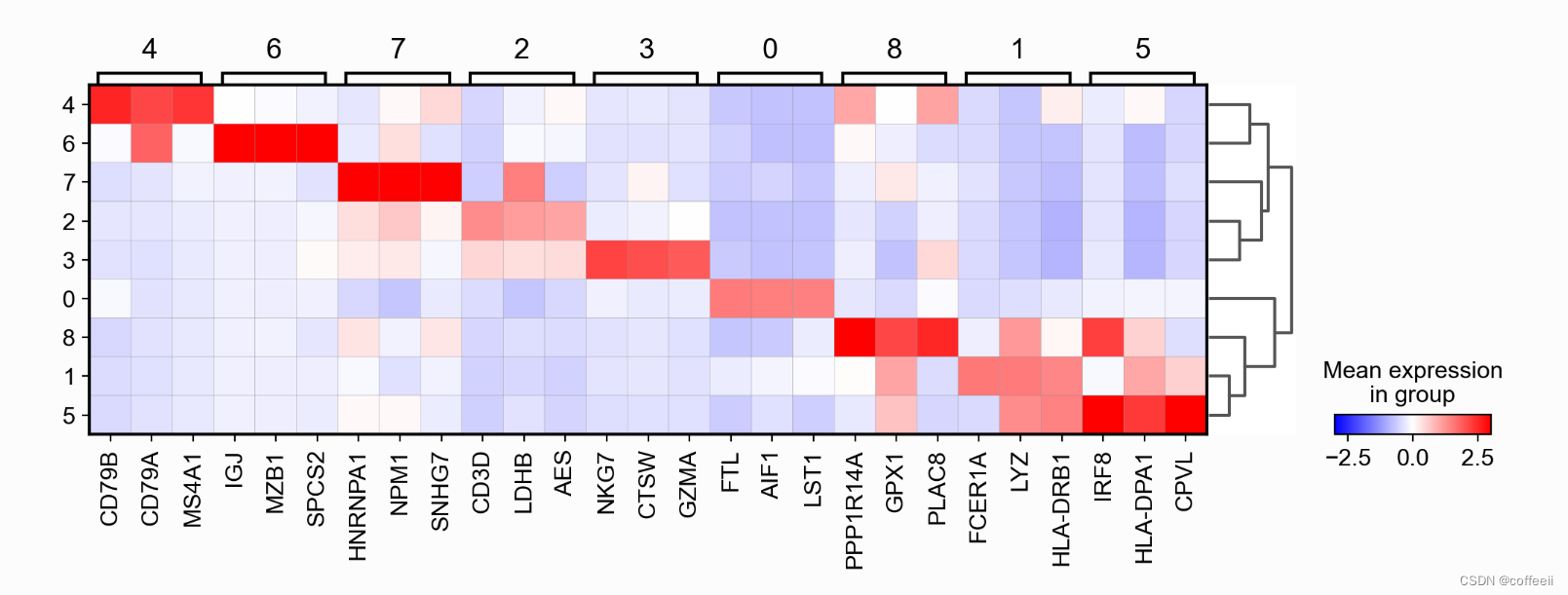
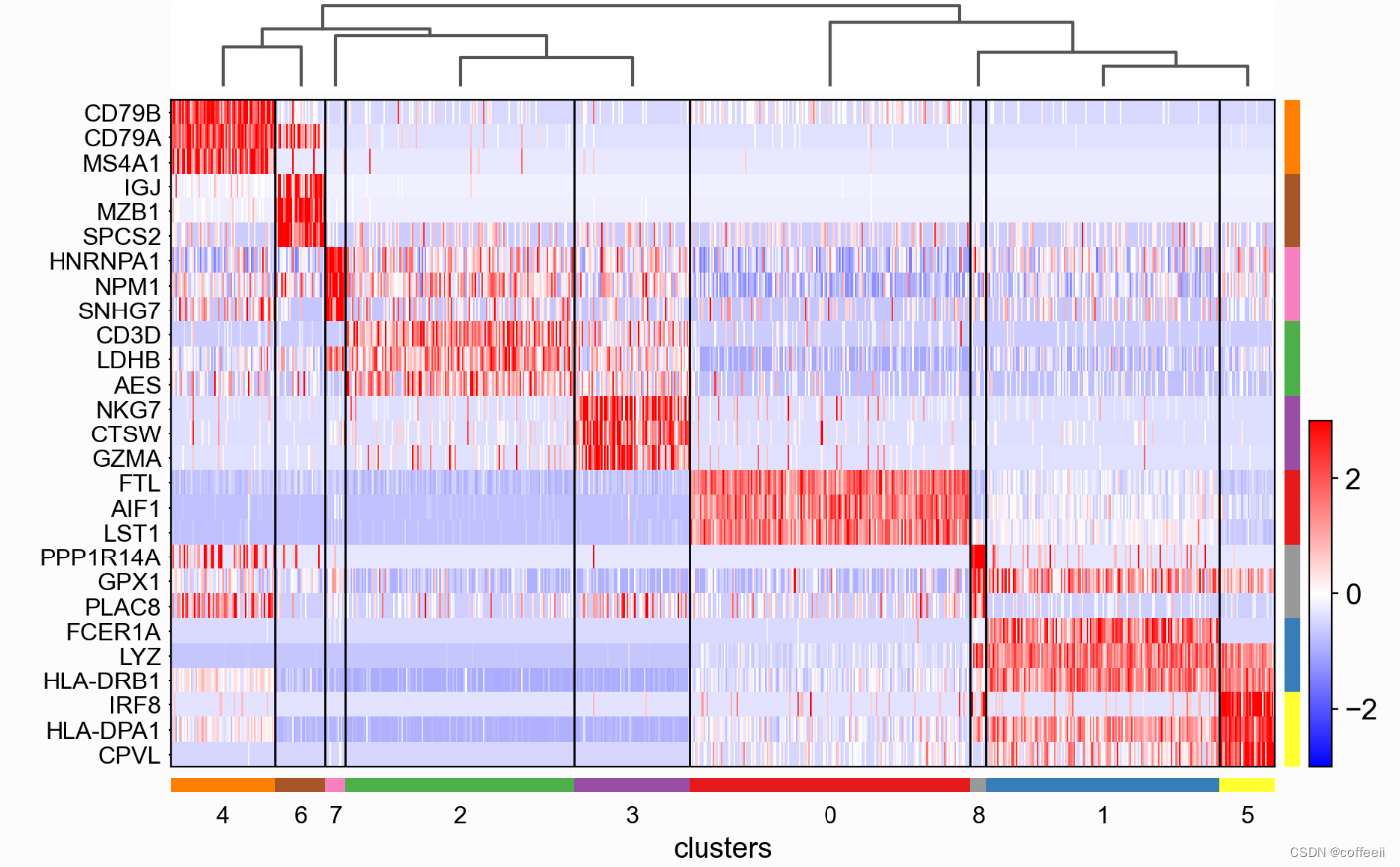 每个类别显示 10 个基因,关闭基因标签并交换轴。请注意,当交换图像时,会出现类别的颜色代码而不是“括号”。
每个类别显示 10 个基因,关闭基因标签并交换轴。请注意,当交换图像时,会出现类别的颜色代码而不是“括号”。