揭开虾蟹类基因组之谜,突破复杂组装难题! – GrandOmics |
您所在的位置:网站首页 › 日本的虾 › 揭开虾蟹类基因组之谜,突破复杂组装难题! – GrandOmics |
揭开虾蟹类基因组之谜,突破复杂组装难题! – GrandOmics
|
虾、蟹类作为重要养殖经济物种,养殖产量约占中国水产养殖总量的约60%以上,可谓是水产界的半壁江山。然而,由于缺乏高质量的基因组信息,虾、蟹类的种质资源创新利用一直受到限制,分子育种进展缓慢。通过深入研究虾蟹类基因组,我们可以更好地理解其生长发育、抗病性以及适应环境的机制,从而为育种和养殖管理提供科学依据。加强对虾蟹类基因组的研究和解析对于推动水产养殖行业的可持续发展具有重要意义。 虾蟹类是世界上公认的高复杂基因组,原因在于其基因组杂合度高、重复序列多、染色体数目多,对基因组组装造成了较大的困难。其次,虾蟹组织样本中蛋白含量较高,在前端实验提取DNA时容易堵孔、污染DNA造成提取困难,因此高质量的虾蟹类基因组DNA获取极为困难。 希望组深耕长读长测序领域十数年,通过结合多平台的测序数据,将SMRT、Nanopore、NGS、Hi-C/Pore-C、Bionano等多种形式的测序数据通过生物信息学技术有机结合,完成了多个虾蟹类基因组组装的合作项目,发表了多篇高分论文。希望组愿与您一起破解虾蟹基因组组装难题,推动水生生物基因组学的蓬勃发展,为水产领域科研增光添彩! 希望组在虾蟹类基因组研究具有极大优势:
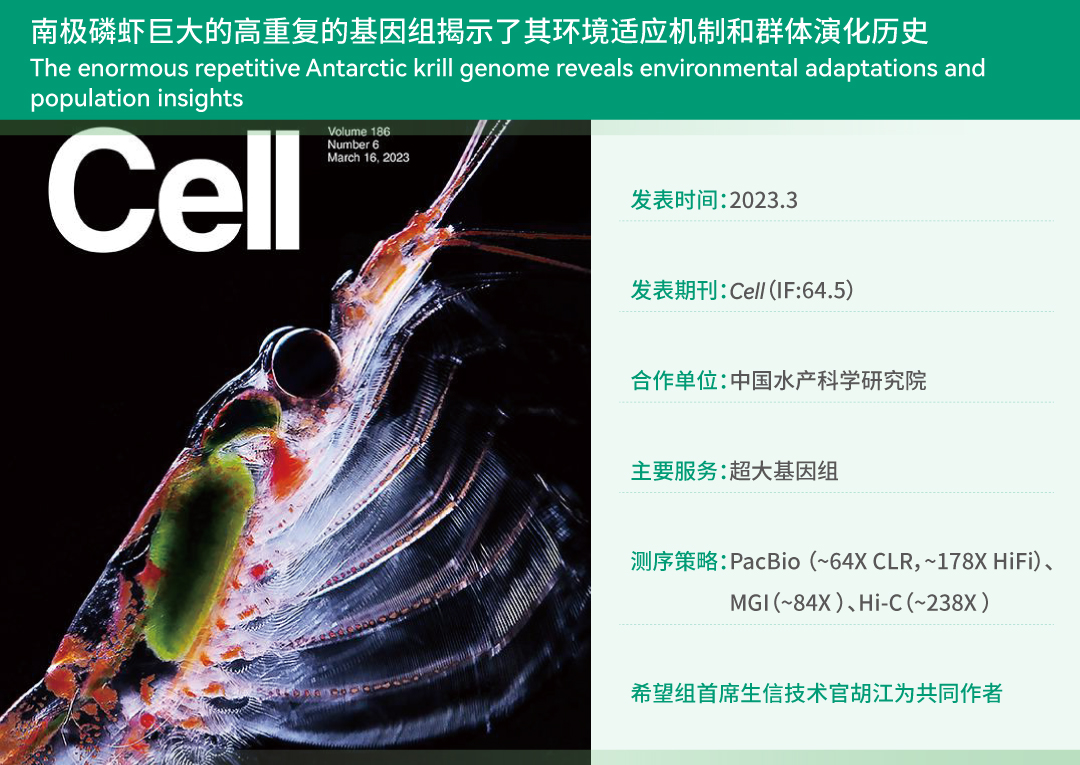
01 PacBio测序、NextDenovo组装助力破译迄今最大动物基因组—48Gb南极磷虾参考序列
摘要 该研究完成了迄今为止最大动物基因组参考序列——南极磷虾基因组组装,并揭示了南极磷虾适应极端环境和群体历史演化的分子基础。研究者利用PacBio、Hi-C结合短读长对南极磷虾进行测序,使用NextDenovo v2.30 组装了48.01Gb的基因组。研究发现,南极磷虾重复序列含量高达92.45%,这源于南极磷虾基因组重复序列的两次爆发式扩张。在该研究中,研究人员对极昼极夜环境的适应性进行了研究。结果发现,在磷虾基因组里鉴定得到的25个显著扩张的基因家族中,分别有6个基因家族与磷虾蜕壳及能量代谢相关。这表明,蜕壳和能量代谢相关基因的改变是南极磷虾对南大洋不稳定食物供应的适应。另外,研究团队还发现,虽然分布在不同的区域,但南极磷虾的遗传序列组成没有明显区别,这就意味着不同地域群体之间没有实质性差异,并且气候变化影响着南极磷虾种群的规模。武汉希望组为本研究提供基因组组装服务,武汉希望组首席生信技术官胡江为共同作者。 技术亮点 这项研究的主要技术亮点是组装有史以来最大的动物基因组测序。基因组中过度丰富的转座子重复序列加剧了这一技术挑战,这成为该研究的主要生物学发现之一。研究者仔细分析了导致巨大基因组大小的重复序列,提供了由重复元件活性引起的基因组大小扩张的一个最佳例子。组装后的基因组使得能够全面分析整个基因组中涉及光周期的基因。对南极高度变化的光照条件的生理反应是磷虾生物学的核心,研究者所组装的基因组资源对这种适应能力进行了极大的详细研究。 参考文献 Shao C, Sun S, Liu K, et al. The enormous repetitive Antarctic krill genome reveals environmental adaptations and population insights. Cell. 2023 Mar 16;186(6):1279-1294.e19. doi: 10.1016/j.cell.2023.02.005. Epub 2023 Mar 2. PMID: 36868220. 02 中华绒螯蟹“断臂再生”
摘要 中华绒螯蟹(Eriocheir sinensis),俗称大闸蟹,是我国高经济价值的水产甲壳动物,全国28个省市区养殖,产业规模大,文化底蕴深厚。由于中华绒螯蟹染色体数目多(2n=146),基因组重复序列高,组装困难,且不同水系群体表现出较为明显的环境适应性和生物学特征。同时,中华绒螯蟹断肢现象在生产上较为普遍,影响了其产业应用性和经济价值。本研究利用第三代测序技术,并结合BioNano光学图谱和Hi-C高通量染色体构象捕获技术,对长江水系中华绒螯蟹的全基因组测序和组装,获得染色体水平的精细基因组图谱。该研究还发现中华绒螯蟹断肢再生早期受表观遗传学的调控,其中SMYDA基因家族只存在于节肢动物,在中华绒螯蟹断肢早期下调表达,而在肢芽生长时期表达回复至未断肢时的水平。进一步分析发现,该基因家族还在中华绒螯蟹从大眼幼体到仔蟹的变态过程中整体差异表达,表明节肢动物特异的SMYDA基因家族在中华绒螯蟹涉及明显形态发生,如变态发育、再生的生物学过程中发挥重要的表观修饰作用。该研究为开展中华绒螯蟹的分子育种提供了重要的基因组资源和平台,为提升养殖生产与管理水平提供了有益指导。武汉希望组提供三代测序组装注释,Hi-C挂载和Bionano光学图谱服务。 参考文献 Wang J, Chen X, Hou X, et al. “Omics” data unveil early molecular response underlying limb regeneration in the Chinese mitten crab, Eriocheir sinensis. Sci Adv. 2022 Sep 16;8(37):eabl4642. doi: 10.1126/sciadv.abl4642. Epub 2022 Sep 16. PMID: 36112682; PMCID: PMC9481118. 03 重要商品蟹——三疣梭子蟹
摘要 三疣梭子蟹(Portunus trituberculatus)是中国重要的商业蟹种,广泛分布于亚太国家沿海水域。研究者结合MGI、Nanopore和Hi-C数据,组装了三疣梭子蟹的第一个染色体水平参考基因组。该基因组大小为1.00 Gb, Contig N50为4.12 Mb,BUSCO达到94.7%,重复序列为54.52%,共有16796个蛋白质编码基因被注释。研究者通过Hi-C数据成功将基因组挂载到50条染色体上,Scaffold N50长度为21.80 Mb。该染色体水平的基因组组装不仅可以促进三疣梭子蟹基本发育和进化的研究,还可以为三疣梭子蟹的繁殖提供重要的资源。武汉希望组提供Nanopore三代测序服务。 参考文献 Tang B, Zhang D, Li H, et al. Chromosome-level genome assembly reveals the unique genome evolution of the swimming crab (Portunus trituberculatus). Gigascience. 2020 Jan 1;9(1):giz161. doi: 10.1093/gigascience/giz161. PMID: 31904811; PMCID: PMC6944217.31904811; PMCID: PMC6944217. 04 红螯螯虾基因组
摘要 研究人员使用108X的PacBio CLR数据、58X的PE100 DNA二代测序数据组装了一个红螯螯虾染色体级基因组,大小为5.26 Gb,Contig N50为144.33 Kb。使用105X的Hi-C数据辅助挂载,将近90%的序列被锚定到100条染色体上,这是迄今报道的染色体数目最多的高质量甲壳动物基因组。该基因组含有78.69%的重复序列和20,460个蛋白编码基因,其中82.40%具有功能注释。这个染色体级基因组将成为其他复杂基因组的组装和甲壳动物进化研究的有价值的参考。 参考文献 Chen H, Zhang R, Liu F, et al. The chromosome-level genome of Cherax quadricarinatus. Sci Data. 2023 Apr 17;10(1):215. doi: 10.1038/s41597-023-02124-z. Erratum in: Sci Data. 2023 May 23;10(1):313. PMID: 37062798; PMCID: PMC10106460. 05 青虾全基因组图谱组装
摘要 日本沼虾,俗名青虾,是我国重要的经济虾类。雄性对虾比雌性对虾生长更快,体积更大,因此培养全雄性种群是实践中水育种繁育的重要目标,为此揭示日本沼虾的性别分化和生殖发育机制对实现遗传改良具有重要的支撑意义。该研究采用65.2X的Illumina数据、90X的PacBio数据和195X的Hi-C数据进行测序组装。通过survey分析,估计日本沼虾的基因组大小约为4.6 Gb。组装得到的日本沼虾染色体水平的基因组大小为4.5 Gb,Contig N50为231.2 Kb,基因组的完整度为92.6%。共构建了49条染色体,使用Hi-C测序数据辅助挂载至染色体水平,有94.7%的基因组数据被挂载到染色体上,scaffold N50长度达86.8 Mb。全基因组共预测到44,086个蛋白编码基因,其中39,317个基因被功能注释。此外,研究者还采集了生殖期和非生殖期的日本沼虾雄性个体样本,进行转录组测序和分析。 参考文献 Jin S, Bian C, Jiang S, et al. A chromosome-level genome assembly of the oriental river prawn, Macrobrachium nipponense. Gigascience. 2021 Jan 18;10(1):giaa160. doi: 10.1093/gigascience/giaa160. PMID: 33459341; PMCID: PMC7812440. 06 克氏原螯虾染色体水平参考基因组
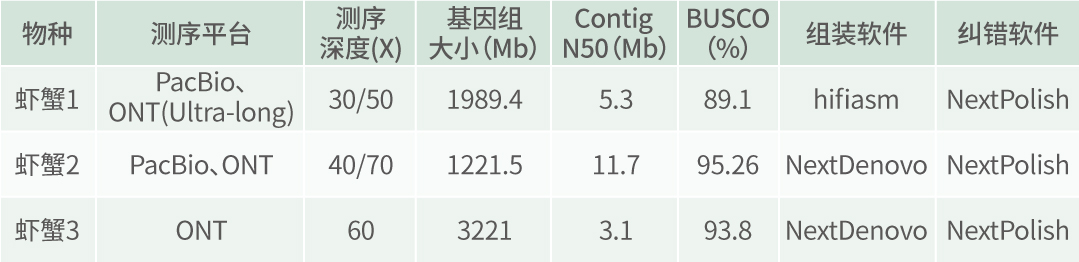
摘要 克氏原螯虾(Procambarus clarkii)(俗称小龙虾)是一种重要的生态和经济甲壳类动物。研究者基于146.93X PacBio数据、112.95X Illumina测序数据和142.23X Hi-C进行denovo组装。研究者得到的染色体水平小龙虾基因组,基因组大小为2.75 Gb,Contig N50为216.75 kb。共构建了94条单倍型染色体,使用Hi-C数据进行辅助挂载,91.22%的基因组数据被挂载到染色体上,scaffold N50长度为17.01Mb。在克氏原螯虾基因组中鉴定出超过2.26 Gb的重复序列,占总基因组序列的82.42%。在这些重复序列中,转座因子(TEs)占多数(约79.61%)。 参考文献 Xu Z, Gao T, Xu Y, et al. A chromosome-level reference genome of red swamp crayfish Procambarus clarkii provides insights into the gene families regarding growth or development in crustaceans. Genomics. 2021 Sep;113(5):3274-3284. doi: 10.1016/j.ygeno.2021.07.017. Epub 2021 Jul 22. PMID: 34303807. 虾蟹类基因组的复杂性无疑给科学家们带来了许多挑战,但是希望组正努力突破这些难题,致力于为大家提供高质量、可靠的虾蟹类基因组组装解决方案,为科学研究和海洋生物保护做出贡献。 最新虾蟹类物种组装结果展示
|
【本文地址】
今日新闻 |
推荐新闻 |