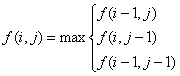生物信息学(2) |
您所在的位置:网站首页 › 序列算法是什么 › 生物信息学(2) |
生物信息学(2)
|
生物信息学系列博客索引
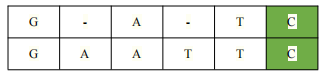
生物信息学(1)——双序列比对之Needleman-Wunsch(NW)算法详解及C++实现 生物信息学(2)——双序列比对之Smith-Waterman(SW)算法详解 生物信息学(3)——双序列比对之BLAST算法简介 生物信息学(4)——多序列比对之CLUSTAL算法详解及C++实现 生物信息学(5)——基于CUDA的多序列比对并行算法的设计与代码实现 项目gitee地址,内附源码、论文等文档信息 1. SW 算法简介Smith-Waterman 算法是由 Temple F. Smith 和 Michael S. Waterman 两人在 1981 年提出来的,是 Needleman-Wunsch 算法的改良版,通过算法的比对,能获 取到局部最优解。SW 算法罚分规则如下: SW 算法罚分规则如公式 给定序列 Seq1 = GGATCGA Seq2 = GAATTCAGTTA (1) 初始化打分矩阵首先将矩阵的第0行与第0列分别用Seq1与Seq2填充,填充时,注意预留出两个字符,将第二行与第二列的元素置0,如图所示:
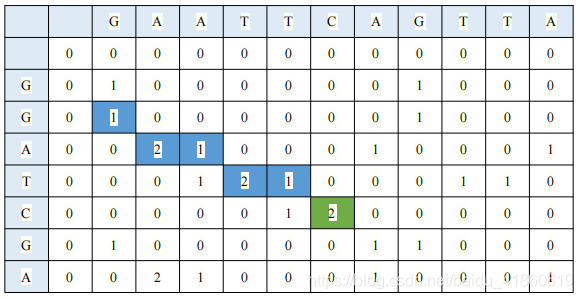
先找到全局最大的值,在此点往左上回溯,到 0 停止 通过回溯规则,构建最终的局部最优解,其中路径朝向左上,即 MATCH/DISMATCH,路径朝左为 seq1 出现 INDEL 情况,路径朝上为 seq2 出现 INDEL 情况,使用 ’-’ 代替。结果如下:最后得出如图 结果,为局部最优解。 问:为什么SW算法没有C++实现? 答:问得好!因为我太懒了,我的毕设没有用到SW算法具体实现,所以无代码,只有原理解释。 4. 总结SW算法相比较于NW算法,虽然S简化了计算过程,且是在NW算法的基础上进行改进,但是其不能获取到全局最优解,而是只能获取到局部最优解。 算法永远是时间、空间、准确性三者交错的结合,减少了时间复杂度也许会增加空间复杂度,减少了空间复杂度,也许会增加运行时间。缩短时空复杂度,算法的精度可能会丢失。有时候,我们能承受巨大的内存空间,无法容忍运行的速度低下,所以我们会选择时间复杂度低的算法,有时候我们想优化时空复杂度,对结果没有偏执的要求,那又是另一种选择。所以适合使用情况的算法才是好的算法。正如下一篇要介绍的BLAST,其并不确保能找到最优解,但尽力在更短时间内找到足够好的解。これは人生かもしれません。 |
【本文地址】
 以罚分规则为基础,得分矩阵的公式如下,可以看到,和上篇介绍的NW算法相比,最大的改变是,多了一个0。这样做就可以杜绝得分矩阵中的负数。
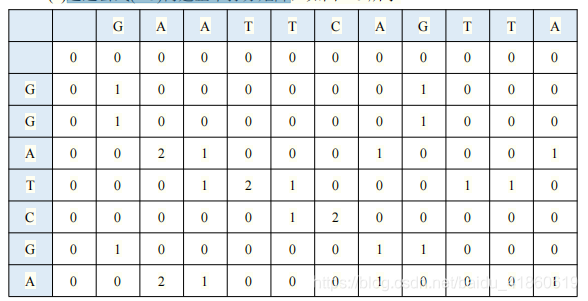
以罚分规则为基础,得分矩阵的公式如下,可以看到,和上篇介绍的NW算法相比,最大的改变是,多了一个0。这样做就可以杜绝得分矩阵中的负数。 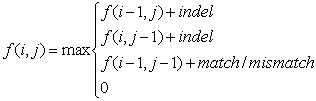 获取得分矩阵后,找全局最大的值,在此点往左上回溯,到0停止,然后根据回溯路径获取局部最优解。回溯规则:回溯规则如公式
获取得分矩阵后,找全局最大的值,在此点往左上回溯,到0停止,然后根据回溯路径获取局部最优解。回溯规则:回溯规则如公式