|
启动子的分析和预测
一、摘要
加深对基因启动子的理解和认知;学会如何获取已知基因的启动子序列数据;熟悉不同启动子分析软件的使用及其适用范围;学会设计启动子克隆引物。熟悉EPD和TransFac数据库的使用;学会使用已知的启动子和转录因子TransFac的HMM模型,并能够独立编程,利用该HMM模型来计算鉴别未知启动子
二、材料和方法
1、硬件平台
处理器:Intel(R) Core(TM)i7-4710MQ CPU @ 2.50GHz 安装内存(RAM):16.0GB
2、系统平台
Windows 8.1、Ubuntu
3、软件平台
【1】Primer-BLAST 【2】Softberry系列工具 【3】Promoter 2.0 【4】BDGP 【5】Cister 【6】NEBcutter
4、数据库资源
NCBI数据库:https://www.ncbi.nlm.nih.gov/ UCSC数据库:http://genome.ucsc.edu/
5、研究对象
人类谷胱甘肽硫转移酶M1的promoter区域
三、结果
基因启动子序列的获取
选择基因:谷胱甘肽硫转移酶M1(GSTM1) 概况:当携带风险基因型时,对环境毒素和致癌物质的敏感性提高,易发生DNA突变和染色体畸变,患白血病的风险因而显著增加。 首先进入UCSC genome browser 查看GSTM1上游5kb范围内有无其他基因。发现该基因的上游存在同一家族的GSTM2,所以promoter大概只有3kb。  图表 1UCSC genome browser 接下来进入Genbank,搜索GSTM1,查看该基因在基因组中的定位和基因结构。 图表 1UCSC genome browser 接下来进入Genbank,搜索GSTM1,查看该基因在基因组中的定位和基因结构。  图表 2查看基因定位和结构 打开该基因的序列信息,获取该基因的启动子序列(包含exon1) 图表 2查看基因定位和结构 打开该基因的序列信息,获取该基因的启动子序列(包含exon1)  
Neural Network Promoter Prediction
进入BDGP: Neural Network Promoter Prediction网站http://www.fruitfly.org/seq_tools/promoter.html,进行启动子预测  图表 3 BDGP: Neural Network Promoter Prediction网站 一共预测出来3个启动子(这个网站预测出来的promoter都是50bp) 图表 3 BDGP: Neural Network Promoter Prediction网站 一共预测出来3个启动子(这个网站预测出来的promoter都是50bp) 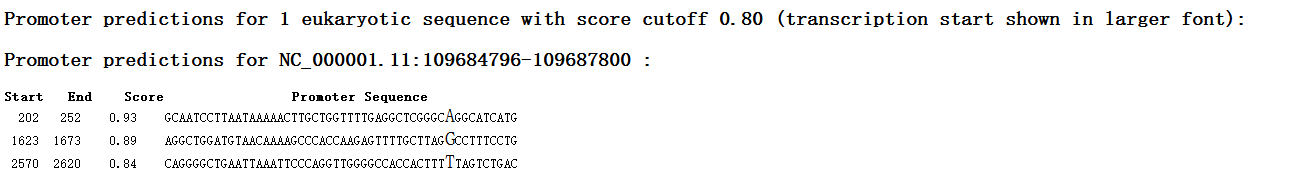
Promoter 2.0 Prediction
使用Promoter 2.0 Prediction Server http://www.cbs.dtu.dk/services/Promoter/ 进行启动子预测,也是一共预测出来3个启动子  图表 5Promoter 2.0预测结果 图表 5Promoter 2.0预测结果
Softberry预测
TSSW、TSSP、TSSG、FPROM都是softberry提供的启动子预测工具,进入 官网(http://www.softberry.com/),然后点击service即可,启动子预测工具网址: http://www.softberry.com/berry.phtml?topic=index&group=programs&subgroup=promoter
TSSW
TSSW具体网址如下(http://www.softberry.com/berry.phtml?topic=tssw&group=programs&subgroup=promoter),输入序列进行预测即可。TSSW并没有预测出来promoter区域。  图表 6TSSW预测结果 图表 6TSSW预测结果
TSSP
TSSP具体网址如下(http://www.softberry.com/berry.phtml?topic=tssp&group=programs&subgroup=promoter),输入序列进行预测即可。共计预测出来一个promoter区域。  图表 7 TSSP预测结果 图表 7 TSSP预测结果
TSSG
TSSG具体网址如下(http://www.softberry.com/berry.phtml?topic=tssg&group=programs&subgroup=promoter),输入序列进行预测即可。TSSG并没有预测出来promoter区域。  图表 8TSSG预测结果 图表 8TSSG预测结果
TSSW/ TSSP/ TSSB
根据一位网友"Janelight"的建议,TSSP、TSSG分别是预测植物和细菌的区域。我这里用的是谷胱甘肽硫转移酶M1(GSTM1) human,只需要使用TSSW预测就可以。 TSSW/ TSSP/ TSSB: Programs for predicting animal, plant and bacterial promoters and functional sites.
FPROM
FPROM具体网址如下(http://www.softberry.com/berry.phtml?topic=fprom&group=programs&subgroup=promoter),输入序列进行预测即可。FPROM并没有预测出来promoter区域。  图表 9FPROM预测结果 图表 9FPROM预测结果
Cister
Transcription Elements预测平台:Cis-element Cluster Finder https://zlab.bu.edu/~mfrith/cister.shtml 由于序列只有3kb,默认参数预测出来的转录元件太少,将average distance between clusters参数由默认的3w修改为3k,最有可能的结果还是NF-1  图表 10Cister预测结果 图表 10Cister预测结果
Match
转录因子预测集合网站http://gene-regulation.com/pub/programs.html (需要注册) 具体网址http://gene-regulation.com/cgi-bin/pub/programs/match/bin/match.cgi  图表 11Match预测结果 图表 11Match预测结果
AliBaba 2.1
转录因子预测集合网站http://gene-regulation.com/pub/programs.html (需要注册) 具体网址http://gene-regulation.com/pub/programs/alibaba2/index.html 预测出来一大堆,下面仅截取其中一部分。  图表 12AliBaba预测结果 图表 12AliBaba预测结果
基因结构绘图
虽然使用了6个promoter预测在线平台,但是只有3个平台预测出promoter。 利用在线平台processon绘制基因结构图  图表 13基因结构图 接下来大致将预测结果分为4个区域,将引物设计的范围同时绘制出来(箭头为引物) 图表 13基因结构图 接下来大致将预测结果分为4个区域,将引物设计的范围同时绘制出来(箭头为引物)
PrimerBlast引物设计
 图表 14引物结构 先使用默认参数进行尝试,设置好Forward primer和Reverse primer的Range,再将PCR product size最大值调成整段序列的长度3005,同时# of primers to return参数调整为1,以方便截图。 图表 14引物结构 先使用默认参数进行尝试,设置好Forward primer和Reverse primer的Range,再将PCR product size最大值调成整段序列的长度3005,同时# of primers to return参数调整为1,以方便截图。 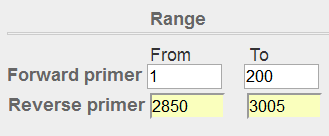 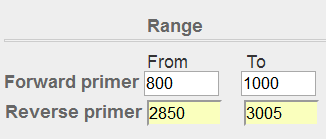 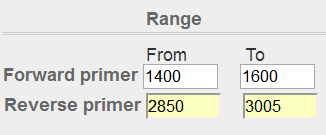 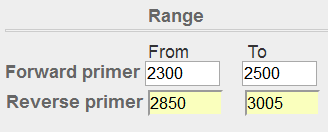 图表 15引物位置 图表 15引物位置  图表 16初步设置参数 然而由于有重复序列,经过repeat filtering,不会跑出来引物,只能将参数调宽松。 图表 16初步设置参数 然而由于有重复序列,经过repeat filtering,不会跑出来引物,只能将参数调宽松。  图表 17默认参数结果 在Advanced parameters的Primer Parameters栏目,将Repeat filter关掉,同样可以看出来GC含量和TM值也筛选掉很多引物,在Internal hybridization oligo parameters栏目,将引物内杂交的参数调整宽松。 图表 17默认参数结果 在Advanced parameters的Primer Parameters栏目,将Repeat filter关掉,同样可以看出来GC含量和TM值也筛选掉很多引物,在Internal hybridization oligo parameters栏目,将引物内杂交的参数调整宽松。  图表 18高级参数调整 图表 18高级参数调整
引物设计结果:
 图表 19第一段 图表 19第一段  图表 20第二段 图表 20第二段  图表 21第三段 图表 21第三段  图表 22第四段 第一段: 图表 22第四段 第一段:
·Sequence (5’->3’)Template strandLengthStartStopTmGC%Self complementaritySelf 3’ complementarityForward primerTCGTACCTACCCTCTGTTCGTPlus211641846052.3840Reverse primerGGGCTGCACTCAGTAAGACTMinus202918289959.395553
第二段:
·Sequence (5’->3’)Template strandLengthStartStopTmGC%Self complementaritySelf 3’ complementarityForward primerCCAAGTGCCCCAACTTAGCAPlus2084986860.545540Reverse primerGGGCTGCACTCAGTAAGACTMinus202918289959.395553
第三段:
·Sequence (5’->3’)Template strandLengthStartStopTmGC%Self complementaritySelf 3’ complementarityForward primerCCAGGCGTCACTAACACAGGPlus201522154160.676031Reverse primerGTTCCGGGAGCGAAGTCAGMinus192874285660.4563.1651
第四段:
·Sequence (5’->3’)Template strandLengthStartStopTmGC%Self complementaritySelf 3’ complementarityForward primerCGAGGGCCCCTAACAGAAAAPlus202405242459.675570Reverse primerCTGGGGCTGCACTCAGTAAGMinus202921290260.396053
NEBcutter酶切位点分析
使用NEBcutter分析该启动子序列,为了更加全面,查找全部的特异性位点Enzymes to use: All specificities http://nc2.neb.com/NEBcutter2/ 保存没有酶切位点“0 cutters”的核酸内切酶数据,见附录。  图表 23NEBcutter结果 图表 23NEBcutter结果
pGL4.17载体
查询pGL4.17的载体数据,获得酶切信息。查询关键词:promega pGL4.17。  图表 24pGL4.17载体信息 其中SfiI、Acc65I、KpnI、SacI、NheI、XhoI、EcoRV、BglII、HindIII这九个酶都属于在promoter内部没有酶切位点的,这些都可以选用。 图表 24pGL4.17载体信息 其中SfiI、Acc65I、KpnI、SacI、NheI、XhoI、EcoRV、BglII、HindIII这九个酶都属于在promoter内部没有酶切位点的,这些都可以选用。
引物设计
从上面九个酶中随便选两个(真实情况要考虑到切割率等问题) 选择KpnI和SacI,下面是酶切位点和保护碱基对应表,KpnI选两个保护碱基。  可以看出来酶切位点序列在反向互补以后和原序列相同,直接把这段序列加在引物前面就成。 最后按照“保护碱基+酶切序列+PCR引物”的顺序,设计用于可以转到pGL4.17载体的引物。 可以看出来酶切位点序列在反向互补以后和原序列相同,直接把这段序列加在引物前面就成。 最后按照“保护碱基+酶切序列+PCR引物”的顺序,设计用于可以转到pGL4.17载体的引物。
·164…184–2918…2899849…868–2918…28991522…1541–2874…28562405…2424–2921…2902Forward PrimerCGAGCTCTCGTACCTACCCTCTGTTCGTCGAGCTCCCAAGTGCCCCAACTTAGCACGAGCTCCCAGGCGTCACTAACACAGGCGAGCTCCGAGGGCCCCTAACAGAAAAReverse PrimerGGGGTACCGGGCTGCACTCAGTAAGACTGGGGTACCGGGCTGCACTCAGTAAGACTGGGGTACCGTTCCGGGAGCGAAGTCAGGGGGTACCCTGGGGCTGCACTCAGTAAG
后续实验流程
接下来,用这四组引物,把四个promoter区域PCR出来,顺带PCR出来的还有保护碱基和酶切序列,导入pGL4.17,用双荧光素酶报告系统看看哪儿个promoter活性最高,大概会出来下面这种图,后面那张图明显说明promoter3活性最高。  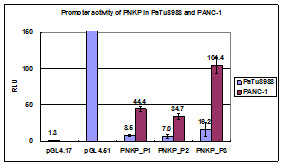 再接下来,还可以用TFSEARCH,TFBSs,TRED这样的转录因子预测软件(上面也做了几个预测),看看活性最高的那段区域和哪儿些转录因子相关,或者用pubmed查查看文献,ENCODE,TRANSFAC等数据库,查找这个基因启动子区域的转录因子信息。 再接下来,还可以用TFSEARCH,TFBSs,TRED这样的转录因子预测软件(上面也做了几个预测),看看活性最高的那段区域和哪儿些转录因子相关,或者用pubmed查查看文献,ENCODE,TRANSFAC等数据库,查找这个基因启动子区域的转录因子信息。
接下来是编程练习部分
HMM模型
TransFac是转录因子数据库,但是好像需要注册才能下载模型的矩阵。 从EPD真核生物启动子数据库下载脊椎动物TATA-box的矩阵(共计12位碱基)。 网址http://epd.vital-it.ch/promoter_elements.php 利用该矩阵建立打分模型,对上面谷胱甘肽硫转移酶M1(GSTM1)的启动子序列进行分析,具体代码见附录。 打分值:每次取出12bp序列计算,依次计算每位碱基所占比例,再累乘得到分值(由于数值太小,分值皆除以最大分值)  图表 25打分值统计图 P值计算:使用bootstrap方法,将12bp序列打乱1000次,再按照上述方法计算分值,如果1000次内有n次分值高于“打乱之前的分值”,则p值为n/1000 图表 25打分值统计图 P值计算:使用bootstrap方法,将12bp序列打乱1000次,再按照上述方法计算分值,如果1000次内有n次分值高于“打乱之前的分值”,则p值为n/1000  图表 26 p值统计图 看的出来,整段promoter区域大部分分值都为0,p值为1。之前在线预测软件中的200,400,1200,2570这四个位置,此处也能预测出来,效果还可以。 图表 26 p值统计图 看的出来,整段promoter区域大部分分值都为0,p值为1。之前在线预测软件中的200,400,1200,2570这四个位置,此处也能预测出来,效果还可以。
附录
”0 cutters”核酸内切酶
Col1Col2Col31AatIIGACGTC2AbaCIIICTATCAV3AbsICCTCGAGG4Acc65IGGTACC5Acc65VGACGCA6AclIAACGTT7AfeIAGCGCT8AflIICTTAAG9AflIIIACRYGT10AhyRBAHIGCYYGAC11AjuI(N)5(N)7GAA(N)7TTGG(N)6(N)512AleICACNNNNGTG13AloI(N)5(N)7GAAC(N)6TCC(N)7(N)514AlwFIGAAAY(N)5RTG15ApyPIATCGAC(N)18NN16AscIGGCGCGCC17AseIATTAAT18AsiSIGCGATCGC19AspDUT2VGNGCAAC20Asu14238IVCGTRAC21BaeI(N)5(N)10ACNNNNGTAYC(N)7(N)522BamHIGGATCC23BarI(N)5(N)7GAAG(N)6TAC(N)7(N)524Bce3081ITAGGAG25BceAIACGGC(N)12NN26BcgINN(N)10CGA(N)6TGC(N)10NN27BclITGATCA28BdaINN(N)10TGA(N)6TCA(N)10NN29BglIIAGATCT30BlpIGCTNAGC31BmgBICACGTC32BmtIGCTAGC33BpuJICCCGT34BsaAIYACGTR35BsbICAACAC(N)19NN36BsiEICGRYCG37BsiWICGTACG38Bsp24I(N)5(N)8GAC(N)6TGG(N)7(N)539Bsp3004IVCCGCAT40Bsp460IIICGCGCAG41BspDIATCGAT42BsrBICCGCTC43BsrGITGTACA44BssHIIGCGCGC45BstAPIGCANNNNNTGC46BstBITTCGAA47BstEIIGGTNACC48BstZ17IGTATAC49Bsu3610IGACGAG50BtgZIGCGATG(N)10NNNN51Cal14237IGGTTAG52CcrNAIIICGACCAG53Cdi11397IGCGCAG54Cdi81IIIGCMGAAG55CdiICATCG56Cgl13032IGGCGCA57Cgl13032IIACGABGG58ClaIATCGAT59Cma23826ICGGAAG60CstMIAAGGAG(N)18NN61DrdIGACNNNNNNGTC62EagICGGCCG63EciIGGCGGA(N)9NN64Eco53kIGAGCTC65EcoRVGATATC66Exi27195IGCCGAC67FseIGGCCGGCC68FspAIRTGCGCAY69FspITGCGCA70GauT27ICGCGCAGG71GdiIICGGCCR72HindIIIAAGCTT73HpaIGTTAAC74Hpy99ICGWCG75HpyAXIVGCGTA76Jma19592IGTATNAC77Jma19592IIGRGCRAC78Kor51IIRTCGAG79KpnIGGTACC80Lmo370IAGCGCCG81Lsp6406VICRAGCAC82Maf25IICACGCAG83MaqICRTTGAC(N)19NN84MauBICGCGCGCG85MkaDIIGAGAYGT86MluIACGCGT87MreICGCCGGCG88MslICAYNNNNRTG89MteIGCGCNGCGC90NaeIGCCGGC91Nbr128IIACCGAC92NgoMIVGCCGGC93NhaXICAAGRAG94NheIGCTAGC95NotIGCGGCCGC96NpeUS61IIGATCGAC97NruITCGCGA98PacITTAATTAA99PaeR7ICTCGAG100Pal408ICCRTGAG101PciIACATGT102PcsIWCGNNNNNNNCGW103Pfl1108ITCGTAG104PflFIGACNNNGTC105PflMICCANNNNNTGG106PlaDICATCAG(N)19NN107PliMICGCCGAC108PmeIGTTTAAAC109PmlICACGTG110PpiI(N)5(N)7GAAC(N)5CTC(N)8(N)5111PshAIGACNNNNGTC112PsiITTATAA113PspXIVCTCGAGB114PsrI(N)5(N)7GAAC(N)6TAC(N)7(N)5115Pst273IGATCGAG116PvuICGATCG117RceICATCGAC(N)18NN118RdeGBICCGCAG119RpaB5ICGRGGAC(N)18NN120RpaBICCCGCAG(N)18NN121RpaIGTYGGAG(N)9NN122RpaTIGRTGGAG123Rsp008IVACGCAG124RspPBTS2IIICTTCGAG125RsrIICGGWCCG126SacIGAGCTC127Saf8902IIICAATNAG128SalIGTCGAC129SbfICCTGCAGG130SexAIACCWGGT131SfiIGGCCNNNNNGGCC132SgrAICRCCGGYG133SgrDICGTCGACG134SnaBITACGTA135SpeIACTAGT136SphIGCATGC137SpnRIITCGAG138SrfIGCCCGGGC139SsmICTGATG140Ssp714IICGCAGCG141SstE37ICGAAGAC(N)18NN142Sth20745IIIGGACGAC143SwaIATTTAAAT144TaqIIICACCCA(N)9NN145TspARh3IGRACGAC146TssIGAGNNNCTC147Tth111IGACNNNGTC148UbaF12ICTACNNNGTC149UbaF13IGAG(N)6CTGG150UbaF14ICCA(N)5TCG151UbaF9ITAC(N)5RTGT152UbaPICGAACG153Xca85IVTACGAG154XhoICTCGAG155ZraIGACGTC
模型矩阵
从EPD下载,保存为data.txt http://epd.vital-it.ch/promoter_elements.php
Position123456789101112A17.719.36.683.409572.394.253.329.317.722.7C21.136.114.8000000932.533G2936.46.800005.820.151.237.733.2T32.28.271.816.6100527.7026.610.512.111.1
R代码
setwd("G:/AllShare/genomicsHomework/HMMmodel")
hmmmodel |  图表 1UCSC genome browser 接下来进入Genbank,搜索GSTM1,查看该基因在基因组中的定位和基因结构。
图表 1UCSC genome browser 接下来进入Genbank,搜索GSTM1,查看该基因在基因组中的定位和基因结构。  图表 2查看基因定位和结构 打开该基因的序列信息,获取该基因的启动子序列(包含exon1)
图表 2查看基因定位和结构 打开该基因的序列信息,获取该基因的启动子序列(包含exon1) 

 图表 3 BDGP: Neural Network Promoter Prediction网站 一共预测出来3个启动子(这个网站预测出来的promoter都是50bp)
图表 3 BDGP: Neural Network Promoter Prediction网站 一共预测出来3个启动子(这个网站预测出来的promoter都是50bp) 
 图表 5Promoter 2.0预测结果
图表 5Promoter 2.0预测结果 图表 6TSSW预测结果
图表 6TSSW预测结果 图表 7 TSSP预测结果
图表 7 TSSP预测结果 图表 8TSSG预测结果
图表 8TSSG预测结果 图表 9FPROM预测结果
图表 9FPROM预测结果 图表 10Cister预测结果
图表 10Cister预测结果 图表 11Match预测结果
图表 11Match预测结果 图表 12AliBaba预测结果
图表 12AliBaba预测结果 图表 13基因结构图 接下来大致将预测结果分为4个区域,将引物设计的范围同时绘制出来(箭头为引物)
图表 13基因结构图 接下来大致将预测结果分为4个区域,将引物设计的范围同时绘制出来(箭头为引物) 图表 14引物结构 先使用默认参数进行尝试,设置好Forward primer和Reverse primer的Range,再将PCR product size最大值调成整段序列的长度3005,同时# of primers to return参数调整为1,以方便截图。
图表 14引物结构 先使用默认参数进行尝试,设置好Forward primer和Reverse primer的Range,再将PCR product size最大值调成整段序列的长度3005,同时# of primers to return参数调整为1,以方便截图。 


 图表 15引物位置
图表 15引物位置  图表 16初步设置参数 然而由于有重复序列,经过repeat filtering,不会跑出来引物,只能将参数调宽松。
图表 16初步设置参数 然而由于有重复序列,经过repeat filtering,不会跑出来引物,只能将参数调宽松。  图表 17默认参数结果 在Advanced parameters的Primer Parameters栏目,将Repeat filter关掉,同样可以看出来GC含量和TM值也筛选掉很多引物,在Internal hybridization oligo parameters栏目,将引物内杂交的参数调整宽松。
图表 17默认参数结果 在Advanced parameters的Primer Parameters栏目,将Repeat filter关掉,同样可以看出来GC含量和TM值也筛选掉很多引物,在Internal hybridization oligo parameters栏目,将引物内杂交的参数调整宽松。  图表 18高级参数调整
图表 18高级参数调整 图表 19第一段
图表 19第一段  图表 20第二段
图表 20第二段  图表 21第三段
图表 21第三段  图表 22第四段 第一段:
图表 22第四段 第一段: 图表 23NEBcutter结果
图表 23NEBcutter结果 图表 24pGL4.17载体信息 其中SfiI、Acc65I、KpnI、SacI、NheI、XhoI、EcoRV、BglII、HindIII这九个酶都属于在promoter内部没有酶切位点的,这些都可以选用。
图表 24pGL4.17载体信息 其中SfiI、Acc65I、KpnI、SacI、NheI、XhoI、EcoRV、BglII、HindIII这九个酶都属于在promoter内部没有酶切位点的,这些都可以选用。 可以看出来酶切位点序列在反向互补以后和原序列相同,直接把这段序列加在引物前面就成。 最后按照“保护碱基+酶切序列+PCR引物”的顺序,设计用于可以转到pGL4.17载体的引物。
可以看出来酶切位点序列在反向互补以后和原序列相同,直接把这段序列加在引物前面就成。 最后按照“保护碱基+酶切序列+PCR引物”的顺序,设计用于可以转到pGL4.17载体的引物。
 再接下来,还可以用TFSEARCH,TFBSs,TRED这样的转录因子预测软件(上面也做了几个预测),看看活性最高的那段区域和哪儿些转录因子相关,或者用pubmed查查看文献,ENCODE,TRANSFAC等数据库,查找这个基因启动子区域的转录因子信息。
再接下来,还可以用TFSEARCH,TFBSs,TRED这样的转录因子预测软件(上面也做了几个预测),看看活性最高的那段区域和哪儿些转录因子相关,或者用pubmed查查看文献,ENCODE,TRANSFAC等数据库,查找这个基因启动子区域的转录因子信息。 图表 25打分值统计图 P值计算:使用bootstrap方法,将12bp序列打乱1000次,再按照上述方法计算分值,如果1000次内有n次分值高于“打乱之前的分值”,则p值为n/1000
图表 25打分值统计图 P值计算:使用bootstrap方法,将12bp序列打乱1000次,再按照上述方法计算分值,如果1000次内有n次分值高于“打乱之前的分值”,则p值为n/1000  图表 26 p值统计图 看的出来,整段promoter区域大部分分值都为0,p值为1。之前在线预测软件中的200,400,1200,2570这四个位置,此处也能预测出来,效果还可以。
图表 26 p值统计图 看的出来,整段promoter区域大部分分值都为0,p值为1。之前在线预测软件中的200,400,1200,2570这四个位置,此处也能预测出来,效果还可以。