单细胞测序数据量和捕获细胞数对数据结果影响大吗? |
您所在的位置:网站首页 › 单细胞测序样品是什么意思 › 单细胞测序数据量和捕获细胞数对数据结果影响大吗? |
单细胞测序数据量和捕获细胞数对数据结果影响大吗?
|
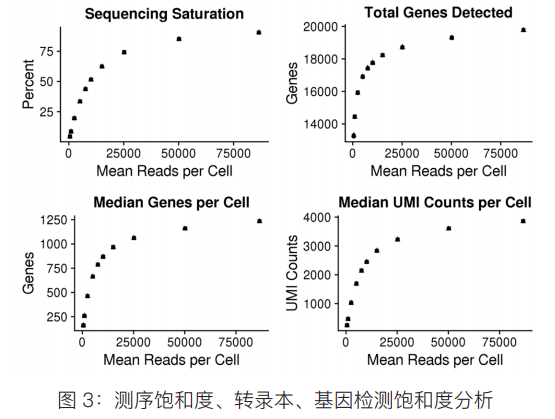
Heimberg et al 的结果表明,低测序深度情况下可以实现样本分群 / 细胞聚类。而为了进一步分析测序数据量和细胞数对细胞聚类的影响,10X 官方设置了不同测序数据量和细胞数对 PBMC 主要细胞类型检测的实验: 1、对 4000 个 PBMC 细胞测序,平均测序数据量为 50K reads/cell; 2、随机抽样设置不同测序深度:500、1K、2.5K、 5K、 7.5K、10K、 15K,、25K和 50K reads/cell; 3、随机抽样设置不同细胞数:100、200、400、600、800、1K、2K、 3K 和 4K细胞 测序深度的影响 此次评估过程采用的是 10X Genomics 单细胞转录组 V2 试剂,V2 试剂建议的最低测序数据量是 50K reads/cell,通过图 3 可知,当平均测序数据达到 50K reads/cell 时,测序饱和度、检测到的转录本数量 / 细胞、基因数量 / 细胞以及总基因数基本饱和,表明官方推荐数据量足够数据分析。
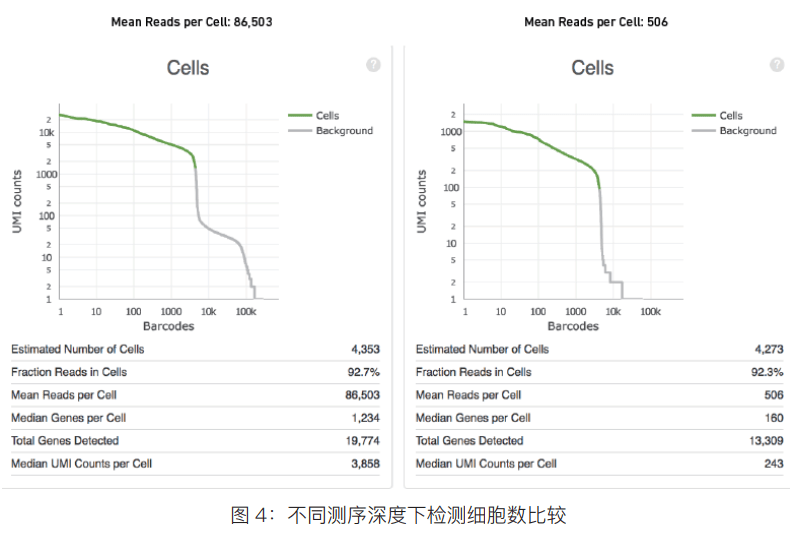
对于检测细胞数,低测序数据量(平均 506 reads/cell)和高测序数据量(平均86503 reads/cell)相差不大,检测到细胞数分别是 4273、4353,但是两种测序深度下,检测到的基因中位数差别很大,分别是 160、1234(图 4)。
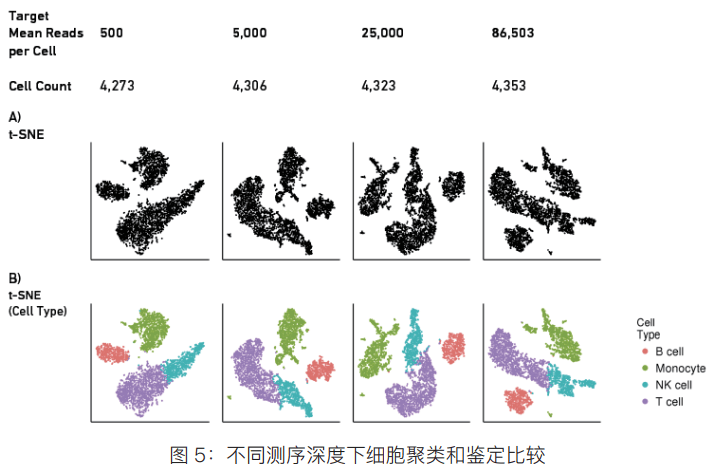
尽管低测序深度时部分指标灵敏度较低,但是通过 graph-based 聚类依然推定了不同细胞亚群,t-SNE 聚类图见图 5A。基于 CD3D、CD3E 鉴定 T 细胞 ; GNLY、NKG7 鉴定NK 细胞 ; CD79A、CD79B 鉴定 B 细胞;CD14、FCGR3A 鉴定单核细胞(图 5B)。
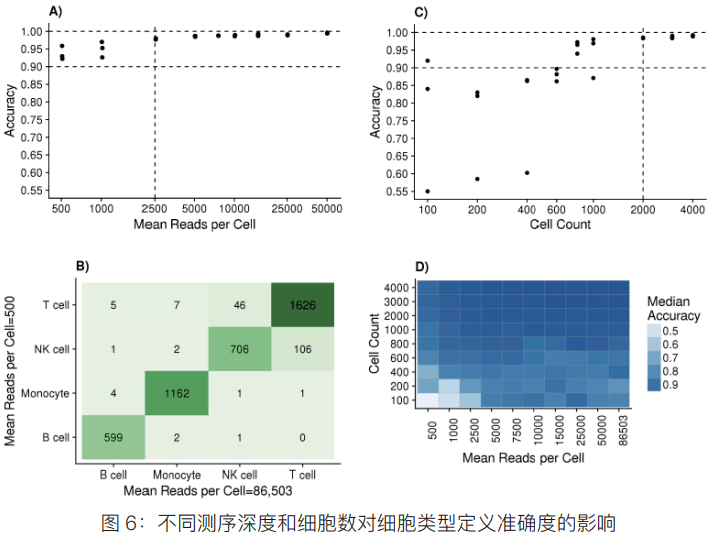
在细胞聚类和鉴定基础上进一步分析,不同测序深度范围内,四种细胞类型(T 细胞、NK 细胞、B 细胞、单核细胞)分类准确度范围为 93-99%(图 6A、B),当测序深度达到 2.5K reads/cell 时,细胞类型分类准确度达到 98% 并且相对一致。当测序深度降低到500 reads/cell 时,相比饱和测序数据量,细胞类型分类准确度降低幅度(7%)远远低于转录本中位数 /cell(94%)、基因中位数 /cell(87%)和总基因鉴定数(33%)的降低幅度。
另 外,2019 年 发 表 在 Circulation 上 的 文 章“Single-Cell Analysis of the Normal Mouse Aorta Reveals Functionally Distinct Endothelial Cell Populations”在设计实验室 比较了不同测序深度对细胞聚类的影响,4 个主动脉样本中,2 条主动脉样本测序深度低 (17,000 reads/cell),2 条主动脉样本测序深度高 (145,000 reads/cell),分析得到的细胞数量二者之间无差异。
以上数据说明,单细胞转录组测序在官方推荐的数据量下(V2 试剂,官方推荐数据量 50K reads/cell;V3 试剂,官方推荐数据量 20K reads/cell),分析获得的各项指标:细胞检测数、测序饱和度、转录本、基因检测饱和度以及检测基因总数均可以达到饱和,当然考虑到单细胞悬液制备时可能会存在细胞碎片等背景干扰,占用一部分数据,可以在选择测序数据量时,对测序数据量进行加倍(例如 5000 个细胞官方推荐最低测序数据量为30G,实际测序可以测60G),但是也不需要加测太多数据,因为加测太多数据得到的收益(例如提高测序饱和度等)增幅远低于付出的测序成本。 细胞数量的影响 为了评估捕获得到的细胞数对细胞类型分类准确性的影响,在每个读取深度基础上按照 100-4000 个细胞进行二次取样。在测序深度为 50K reads/cell 时,不同细胞数量下细胞类型分类准确性范围为 82-99%(图 6C)。当细胞数是 1000 时,精确度变化幅度为10%(87-98%),而在 100 个细胞时,准确度估计值为 55-92%,表明随着细胞计数的减少,细胞分类准确度变异性增加。综合测序数据量和细胞数,可知在低细胞数情况下,随着测序深度增加,细胞类型分类准确度依然维持在较低水平,但在低测序深度情况下,细胞类型分类准确度可以随细胞数量增加而增加,表明细胞数对细胞类型分类准确度的影响大于测序深度。 参考文献 Heimberg, G., Bhatnagar, R., El-Samad, H., & Thomson, M. (2016). Low Dimensionality in Gene Expression Data Enables the Accurate Extraction of Tranional Programs from Shallow Sequencing. Cell Systems, 2(4), 239–250. doi:10.1016/j.cels.2016.04.001. Kalluri, A. S., Vellarikkal, S. K., Edelman, E. R., Nguyen, L., Subramanian, A., Ellinor, P. T., … Gupta, R. M. (2019). Single Cell Analysis of the Normal Mouse Aorta Reveals Functionally Distinct Endothelial Cell Populations. Circulation.doi:10.1161/circulationaha.118.038362.
|
【本文地址】
今日新闻 |
推荐新闻 |

 返回搜狐,查看更多
返回搜狐,查看更多