g |
您所在的位置:网站首页 › 人工转录因子是将某种已知转录因子的 › g |
g
|
TF结合与人类基因组中的内源性G4结构紧密相关gydF4y2Ba
由于DNA结构是DNA-蛋白相互作用的基础,我们探索了内源性TF结合与G4二级结构的关系。为此,我们使用了人类K562慢性髓性白血病细胞和HepG2肝细胞癌细胞,因为这些细胞已经被ENCODE广泛地定位为蛋白质结合位点[gydF4y2Ba22gydF4y2Ba]。我们首先生成了G4结构的全基因组图谱(附加文件)gydF4y2Ba1gydF4y2Ba:图S1b,以下简称内源性G4s)通过G4 ChIP-seq从K562和HepG2细胞的染色质中提取[gydF4y2Ba23gydF4y2Ba]使用G4结构特异性抗体BG4 [gydF4y2Ba24gydF4y2Ba]。为了消除可能的抗体与染色质相关RNA或DNA/RNA杂交G4s的相互作用,在免疫沉淀之前用RNase A处理染色质[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba25gydF4y2Ba]。我们在K562(9205位点)和HepG2(8805位点)中观察到数千个内源性G4位点,细胞系之间共有4825个位点(附加文件)gydF4y2Ba1gydF4y2Ba图S1c-e)。大多数内源G4s (8688/9205, K562 94%;6894/8805,在HepG2中占78%)包含先前通过体外全基因组DNA聚合酶停止测定显示物理形成G4结构的序列[gydF4y2Ba26gydF4y2Ba](以下称为潜在的G4s)(附加文件gydF4y2Ba1gydF4y2Ba:图S1f和图g)。大多数内源性G4s(9043/9205, 98%在K562;8430/8805 (HepG2中96%)位于开放染色质上,与dna酶超敏位点重叠。在这两种细胞系中,许多G4s(约40%)位于转录起始位点(TSS)上游约80 bp的启动子中gydF4y2Ba1gydF4y2Ba:图s1和图i)。然后,我们将内源性G4s与各种染色质相关蛋白和来自ENCODE的组蛋白标记的结合位点进行了比较(完整列表见附加文件)gydF4y2Ba2gydF4y2Ba和gydF4y2Ba3.gydF4y2Ba:补充数据表S1和S2)。该分析表明,许多tf在内源性G4位点富集,并立即提示TF-G4直接相互作用(图4)。gydF4y2Ba1gydF4y2Baa和附加文件gydF4y2Ba1gydF4y2Ba(图S2),特别是因为一些最富集的蛋白质,如FUS和SP1,先前已被认为在体外与DNA G4s相互作用[gydF4y2Ba20.gydF4y2Ba]。尽管每个细胞系具有不同的G4景观,但tf在内源性G4s上大多表现出相似的富集(Spearman相关性)gydF4y2BargydF4y2Ba年代gydF4y2Ba= 0.54,见图。gydF4y2Ba1gydF4y2Bab和附加文件gydF4y2Ba4gydF4y2Ba(补充数据表S3),表明G4结合是某些tf的普遍特性。内源性G4s基本上缺乏转录抑制因子(如CBX8、ZNF318、EZH2和PHB2)和抑制性组蛋白标记(如H3K27me3、H3K9me3)。gydF4y2Ba1gydF4y2Baa和附加文件gydF4y2Ba1gydF4y2Ba:图S2),这与之前的观察结果一致,即内源性启动子G4s与高转录水平相关[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba]。gydF4y2Ba 图1gydF4y2Ba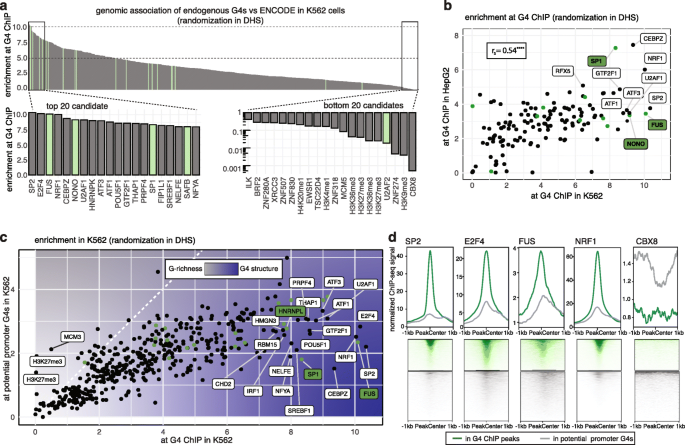
TF结合与人类基因组中的内源性G4结构紧密相关。gydF4y2Ba一个gydF4y2BaK562中G4 ChIP-seq位点的524个ENCODE蛋白富集用于开放染色质(DHS)的随机化。突出显示前20名和后20名候选人。绿色阴影表示与g4相关的蛋白。gydF4y2BabgydF4y2Ba与内源性G4s的基因组关联在K562和HepG2细胞中是一致的。斯皮尔曼相关检验(gydF4y2BargydF4y2Ba年代gydF4y2Ba, * * * *gydF4y2BaPgydF4y2BaxgydF4y2Ba-轴)和HepG2 (gydF4y2BaygydF4y2Ba设在)细胞。绿色阴影表示与g4相关的蛋白。gydF4y2BacgydF4y2BaENCODE获得的TFs与内源性G4s的基因组关联(gydF4y2BaxgydF4y2Ba-轴)和启动子上潜在的G4控制位点(gydF4y2BaygydF4y2Ba设在)。结合独立于二级结构形成的蛋白质在两个数据集中应该显示出相似的富集(白色虚线)。绿色阴影表示与G4相关的蛋白。gydF4y2BadgydF4y2Ba内源性G4位点(绿色)和对照序列(灰色)周围富集候选SP2、E2F4、FUS和NRF1以及非富集TF CBX8的占用谱。内源性G4s的链结性来源于具有G4形成电位的序列的链结数据[gydF4y2Ba26gydF4y2Ba(参见“gydF4y2Ba材料与方法gydF4y2Ba”)gydF4y2Ba 全尺寸图像gydF4y2Ba为了证实在G4s处观察到的TF富集不是由于一级序列的g丰富度,而是由二级结构决定的(附加文件)gydF4y2Ba1gydF4y2Ba:图S3a),我们评估了具有g4形成潜力的控制位点[gydF4y2Ba26gydF4y2Ba]在开放染色质的启动子(上游1 kb TSS和5'UTR),但没有检测到内源性G4结构(附加文件)gydF4y2Ba1gydF4y2Ba:图S3b和c)。许多tf在内源性G4s上的富集程度高于富g对照位点(图S3b和c)。gydF4y2Ba1gydF4y2Bac和附加文件gydF4y2Ba5gydF4y2Ba:补充数据表S4)。例如,在内源性G4s, SP2富集10.3倍,而在富含g的对照位点,SP2富集2.2倍,这表明G4二级结构对特定的tf很重要。与对照位点相比,内源性G4s中最强富集TF的平均TF ChIP-seq结合信号也要高得多(图2)。gydF4y2Ba1gydF4y2Bad)。内源性G4s中最强富集TF的平均TF ChIP-seq信号也比对照位点高得多(图4)。gydF4y2Ba1gydF4y2Bad).与TFs直接招募到G4结构一致,大量TFs的占位谱通常以内源性G4位点为中心(例如,100个TFs在±20 bp内,177个在±40 bp内)(附加文件)gydF4y2Ba1gydF4y2Ba:图S2e)。gydF4y2Ba r环(三链DNA - RNA杂交体)在新生RNA退火回模板DNA时形成。r -环与富含gc的启动子区域有关[gydF4y2Ba27gydF4y2Ba],而G4s和r环的相互作用被认为会影响转录[gydF4y2Ba28gydF4y2Ba]。利用R-ChIP和DRIP-seq数据分析K562细胞[gydF4y2Ba29gydF4y2Ba],内源性G4s和R-loop部分共出现(1431个重叠峰)。r环主要位于G4s的相反链和下游(~ 140 bp)(附加文件)gydF4y2Ba1gydF4y2Ba:图S4a)。虽然有几个tf似乎富集在内源性G4s的下游,表明与r -环相互作用,但大多数tf集中在G4s上(附加文件)gydF4y2Ba1gydF4y2Ba:图S2e)。重要的是,在G4s,如SP2、E2F4、NRF1或FUS高度富集的TF的ChIP信号谱被发现集中在G4s而不是r环上,支持直接募集到G4s而不是r环(图S4b)。gydF4y2Ba 接下来,我们通过比较内源性G4s和JASPAR获得的一致结合位点上的TF富集,研究了G4s和双链DNA对TF募集的相对贡献[gydF4y2Ba30.gydF4y2Ba]。大多数tf(165/193, ~ 85%)在K562细胞内源性G4s上的富集程度与一致的启动子结合位点相同或更高(附加文件)gydF4y2Ba1gydF4y2Ba:图5a和附加文件gydF4y2Ba6gydF4y2Ba:补充数据表S5)和32个tf(包括SP2, SP1和E2F4)在开放染色质中内源性G4s的富集程度比预测的一致启动子结合位点强2倍以上(附加文件)gydF4y2Ba1gydF4y2Ba:图S5b和附加文件gydF4y2Ba6gydF4y2Ba:补充数据表S5)。这些数据表明G4二级结构比双链DNA更有效地招募几个tf。gydF4y2Ba K562细胞中G4s最富集的20个蛋白(FUS、NONO、U2AF1、HNRNPK和HNRNPL)中有5个被归类为识别RNA或单链DNA(附加文件)gydF4y2Ba1gydF4y2Ba:图S5c),通常不被认为是传统的tf,因为它们缺乏特异性的双链DNA结合序列[gydF4y2Ba1gydF4y2Ba]。这些蛋白在转录调控中显然很重要[gydF4y2Ba31gydF4y2Ba],但不知道它们是否直接结合DNA。我们的研究结果支持这些因子与G4密切相关,并且其中一些蛋白质可以通过DNA G4结构被招募到染色质上。gydF4y2Ba tf选择性结合G4结构gydF4y2Ba为了证实上述鉴定的内源性g4富集tf直接与DNA G4s结合,我们进行了生物物理相互作用试验。单链,3 ' -生物素化寡核苷酸折叠成良好表征(附加文件)gydF4y2Ba1gydF4y2Ba:表S1) G4结构(G4 Myc和G4 Kit1)与双链DNA控制寡核苷酸和突变或8-氮杂-7-去氮杂鸟苷取代[gydF4y2Ba32gydF4y2Ba] (ssMyc*)控件不能折叠成G4s,其中ssMyc*控件保持父序列相同的g丰富度。通过圆二色光谱法确认了G4形成的存在与否(附加文件)gydF4y2Ba1gydF4y2Ba:图S6)。利用固定化寡核苷酸从K562核提取物中亲和捕获g4结合tf,然后用特异性抗体进行western blotting分析。基于它们在K562和HepG2细胞内源性G4s上的富集,我们选择了33个高富集的tf来研究它们与G4的结合特性。引人注目的是,很大一部分tf(22/ 33,66%)显示出与G4结构结合的能力(图2)。gydF4y2Ba2gydF4y2Baa、附加文件gydF4y2Ba1gydF4y2Ba:图S7和表S2)。大多数候选基因结合Myc G4和Kit1 G4,而少数tf(如SRSF1, RBM15)偏爱一种G4结构。至关重要的是,大多数g4结合tf很少或没有与突变体单链和双链对照结合(17/22)。此外,最富集的候选蛋白(SP2、FUS和NRF1)与单链7-脱氮鸟嘌呤控制序列(ssMyc*)很少或没有结合;无花果。gydF4y2Ba2gydF4y2Bab),这进一步证实了G4结构形成本身而不需要g丰富度来结合。相比之下,四个候选序列(如NONO)更混杂,与至少一个对照序列的G4s结合程度相似,而TARDBP对单链DNA表现出非常强的偏好。对于一些高富集的tf,如E2F4和CEBPZ,没有检测到G4结合,因此这些蛋白质可能通过其他间接相互作用被招募到G4上。或者,与内源性G4s相关的结构特征,如i-motif [gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba]或R-loops [gydF4y2Ba35gydF4y2Ba,可能有助于他们的招募。SP2、NRF1、FUS、MYC、YY1和ZHX1的富集水平与其与共识序列对照的结合水平相当(图2)。gydF4y2Ba2gydF4y2Bab和附加文件gydF4y2Ba1gydF4y2Ba: S7b),大致与以往的报告一致[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba]。重要的是,两个阴性对照,FOXA1和CTCF(图2)。gydF4y2Ba2gydF4y2Baa),在内源性G4s处显示低富集(附加文件)gydF4y2Ba2gydF4y2Ba:补充数据表S1),不与G4寡核苷酸结合,由于其富含g的共识结合基序,CTCF也可以作为对照。值得注意的是,核裂解物的亲和富集实验不能区分G4直接结合和共结合事件;然而,我们的发现与染色质中大量tf向G4结构的募集是一致的(附加文件)gydF4y2Ba1gydF4y2Ba:图S8)。gydF4y2Ba 图2gydF4y2Ba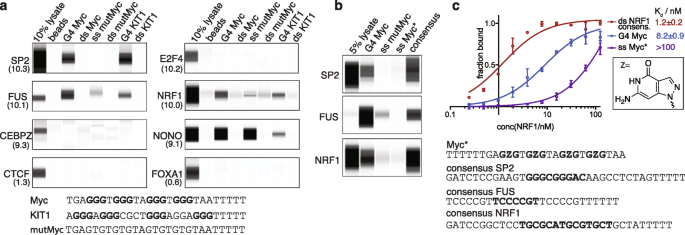
tf选择性地与G4结构结合。gydF4y2Ba一个gydF4y2Ba不同G4寡核苷酸及对照序列的亲和下拉western blot分析。在开放染色质中随机化的K562内源性G4s的基因组富集显示在括号中。gydF4y2BabgydF4y2BaSP2、FUS和NRF1使用G4寡聚物、无法形成G4结构的单链寡聚物(ss mutMyc和ss Myc*)和各自的一致序列进行亲和下拉。gydF4y2BacgydF4y2BaELISA测定的结合曲线显示重组FLAG-NRF1与NRF1双链DNA一致序列和G4结构具有高亲和力结合,但与单链7-deaza对照的结合明显较弱(误差条显示标准差)。gydF4y2BaNgydF4y2Ba= 3)gydF4y2Ba 全尺寸图像gydF4y2Ba为了测量TF-G4相互作用的明显结合亲和力,我们采用了以NRF1为例的酶联免疫吸附试验(ELISA),因为NRF1在K562和HepG2染色质的内源性G4s上都高度富集,但值得注意的是,它不具有富含g的双链DNA共识基序(附加文件)gydF4y2Ba1gydF4y2Ba:图S3)。重组NRF1与双链DNA一致序列(gydF4y2BaKgydF4y2BadgydF4y2Ba= 1.2±0.2 nM)和折叠的G4 Myc结构(gydF4y2BaKgydF4y2BadgydF4y2Ba= 8.2±0.9 nM),但与单链8-氮杂-7-去氮杂鸟苷取代的ssMyc* (gydF4y2BaKgydF4y2BadgydF4y2Ba> 100 nM)。gydF4y2Ba2gydF4y2Bac).我们还观察到对其他四种确定的G4结构的纳摩尔亲和力(gydF4y2BaKgydF4y2BadgydF4y2Ba范围从1.9-7.5 nM)和4-14倍于相应的双链对照序列的选择性,突出了G4二级结构形成对这些位点结合的重要性(附加文件)gydF4y2Ba1gydF4y2Ba:图S9)。gydF4y2Ba 小分子配体对天然染色质中TF与G4s结合的竞争gydF4y2Ba染色质结构影响TF募集和G4格局[gydF4y2Ba16gydF4y2Ba];因此,在天然染色质环境下验证和研究TF-G4相互作用是至关重要的。启动子中G4形成序列的基因组编辑将不可避免地改变双链DNA中TF结合位点的序列,因此我们使用G4特异性小分子选择性地与内源性G4位点的TF竞争。我们评估了小分子吡哆抑制素(PDS) [gydF4y2Ba39gydF4y2Ba]使用elisa进行选择性竞争。PDS与人NRF1与Myc G4 DNA的结合形成竞争gydF4y2Ba集成电路gydF4y2Ba50gydF4y2Ba值为0.18±0.03 μM,这与之前测定的G4对PDS的结合亲和力一致[gydF4y2Ba40gydF4y2Ba(图。gydF4y2Ba3.gydF4y2Baa).相比之下,PDS并未损害NRF1与其双链DNA一致序列的结合(图2)。gydF4y2Ba3.gydF4y2Baa).同样,K562核裂解物对SP2、NRF1和FUS的亲和富集实验表明,PDS可以抑制所有三种tf与折叠G4低聚物的结合,并与IC呈剂量依赖性gydF4y2Ba50gydF4y2Ba值范围从60 nM到> 5 μM,当使用双一致性序列时没有竞争(图2)。gydF4y2Ba3.gydF4y2Bab和附加文件gydF4y2Ba1gydF4y2Ba: S10)。然后,我们研究了PDS与TFs在K562染色质G4位点的竞争。我们使用了保持转录活性的分离细胞核[gydF4y2Ba41gydF4y2Ba],染色质组织[gydF4y2Ba42gydF4y2Ba]和TF结合概况[gydF4y2Ba43gydF4y2Ba]以改善对小分子剂量的控制,并采用ChIP方法对天然染色质而不是交联染色质进行分析,以分析TF结合[gydF4y2Ba43gydF4y2Ba(图。gydF4y2Ba3.gydF4y2Bac).通过ChIP-qPCR测量已知内源性G4结构(来自TF ChIP-seq和G4 ChIP-seq)的TF占用率(附加文件)gydF4y2Ba1gydF4y2Ba表5)。PDS处理导致G4测试位点SP2、NRF1和FUS占用显著减少(47-71%)。gydF4y2Ba3.gydF4y2Bad)。对照(非g4结合)tf FOXA1和CTCF的占用率未见变化(图4)。gydF4y2Ba3.gydF4y2Bae).与染色质中G4位点结合的SP2、NRF1和FUS因此被竞争的G4配体所减少,正如TF招募到G4结构所预期的那样。对于SP2,一个ICgydF4y2Ba50gydF4y2Ba根据剂量响应实验估计~ 60 μM的值(图2)。gydF4y2Ba3.gydF4y2Baf),与TF亲和度为~ 10 nM、核TF蛋白浓度为~ 1.5 μM的单位点直接竞争模型一致(见附加文件)gydF4y2Ba7gydF4y2Ba:补充讨论)。gydF4y2Ba 图3gydF4y2Ba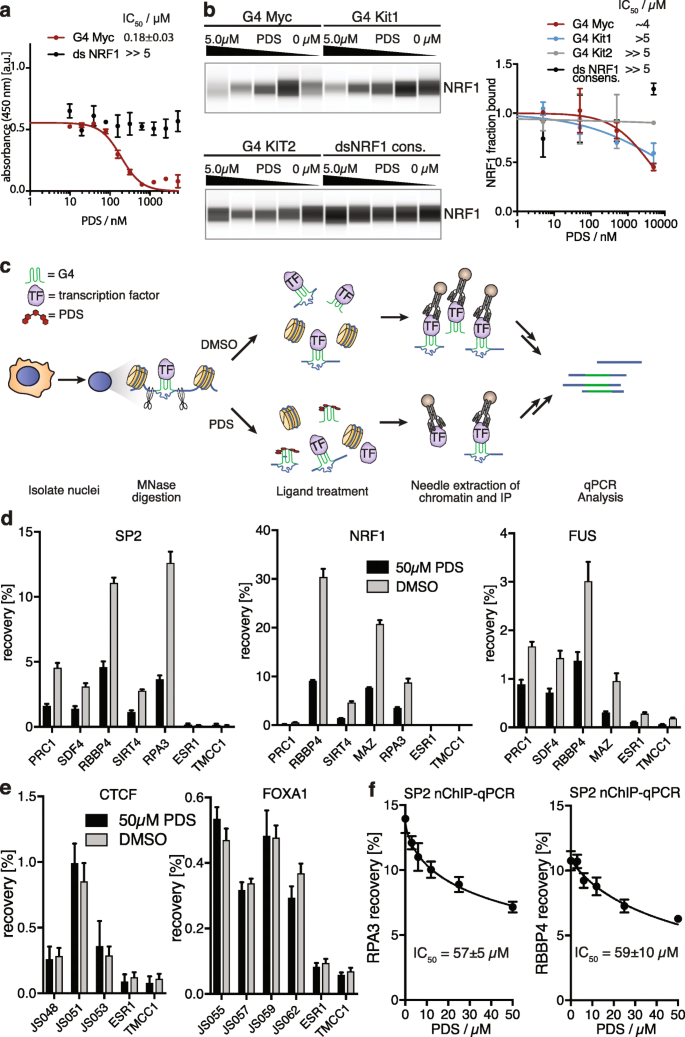
小分子配体对天然染色质中TF与G4s结合的竞争。gydF4y2Ba一个gydF4y2Ba竞争ELISA。固定化的G4 Myc和双链DNA共识寡聚物与G4配体PDS浓度增加预孵育,然后用重组FLAG-NRF1 (20 nM)预孵育(误差条显示标准偏差,gydF4y2BaNgydF4y2Ba= 3)。gydF4y2BabgydF4y2BaK562细胞核裂解物中NRF1的PDS剂量依赖性竞争。PDS取代了来自不同G4低聚物的tf,但不干扰与双链DNA一致低聚物的结合(误差条显示标准偏差,gydF4y2BaNgydF4y2Ba= 2)。gydF4y2BacgydF4y2BaG4配体处理和芯片检测后TF置换方案。gydF4y2BadgydF4y2Bag4相关SP2、NRF1和FUS结合的原生ChIP-qPCR显示pds依赖性信号减少。gydF4y2BaxgydF4y2Ba-轴,选择G4 ChIP-seq和TF ENCODE ChIP信号的阳性区域和两个没有G4和TF ChIP-seq信号的阴性控制区域(ESR1, TMCC1)(误差条显示平均值的标准误差,gydF4y2BaNgydF4y2Ba= 4)。gydF4y2BaegydF4y2Ba对照CTCF和FOXA1的原生ChIP-qPCR未被PDS取代(误差条显示平均值的标准误差,gydF4y2BaNgydF4y2Ba= 4)。gydF4y2BafgydF4y2Ba原生SP2 ChIP-qPCR在两个阳性区域的pds依赖性信号减少(误差条显示平均值的标准误差,gydF4y2BaNgydF4y2Ba= 3)gydF4y2Ba 全尺寸图像gydF4y2Ba G4s是TFs募集的枢纽,以增强转录gydF4y2Ba我们注意到,在体外,相当数量的tf与相同的G4结构结合(图2)。gydF4y2Ba2gydF4y2Baa和附加文件gydF4y2Ba1gydF4y2Ba: S7)和染色质(图7)。gydF4y2Ba3.gydF4y2Bad和附加文件gydF4y2Ba1gydF4y2Ba: S8)。在K562和HepG2染色质中,大多数内源性G4s(位于开放染色质中可接近的启动子中)与缺乏内源性G4s的启动子相比,与更多的TF结合位点重叠(图5)。gydF4y2Ba4gydF4y2Baa).在先前的研究中,在哺乳动物基因组中突出显示了数千个不同tf结合的高占用靶点[gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba]。虽然这一观察结果部分归因于高表达基因和富含gc基因座的ChIP技术产物[gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba],最近的研究表明,这种结合现象不是人为的,而是基于TF-DNA的直接相互作用[gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba]。一个主要的争论点是发现许多TF结合位点与已知的共识基序不匹配[gydF4y2Ba47gydF4y2Ba]。我们现在假设,像G4s这样的DNA二级结构是一种识别特征,解释了多个tf如何与相同的基因组位点结合。此外,我们发现随着内源性G4s上结合的tf数量的增加,RNA聚合酶2的占用和转录活性也随之增加(图2)。gydF4y2Ba4gydF4y2Bab和附加文件gydF4y2Ba1gydF4y2Ba:图S11)。在缺乏G4s的启动子中也观察到类似的相关性,但应该注意的是,内源性G4s被更多数量的tf占据得更多(参见图2中的不同类别)。gydF4y2Ba4gydF4y2Bab)。这就从机制上解释了为什么内源性启动子G4s标记的基因显示出更高的总体转录水平(gydF4y2BaPgydF4y2Ba−16gydF4y2Ba,未配对Wilcoxon检验)(图。gydF4y2Ba4gydF4y2BaC),如先前在人表皮角质形成细胞中观察到的[gydF4y2Ba16gydF4y2Ba]。综上所述,我们认为内源性G4s为许多不同的TF复合物提供了非规范的对接位点,通过增加RNA聚合酶2的招募,从而实现更频繁和有效的相互作用,从而导致更大的转录输出(图2)。gydF4y2Ba4gydF4y2Bad)。在同一细胞中,数十或数百个tf同时共同占据单个大分子复合体的基因组位点是不太可能的[gydF4y2Ba48gydF4y2Ba]。虽然某些tf可能会合作结合它们的目标[gydF4y2Ba4gydF4y2Ba],更有可能的是,许多tf可以动态竞争结合到同一个G4位点。在一个大的细胞群中,由于整个细胞群的信号平均,这将导致在同一位点明显的共定位。gydF4y2Ba 图4gydF4y2Ba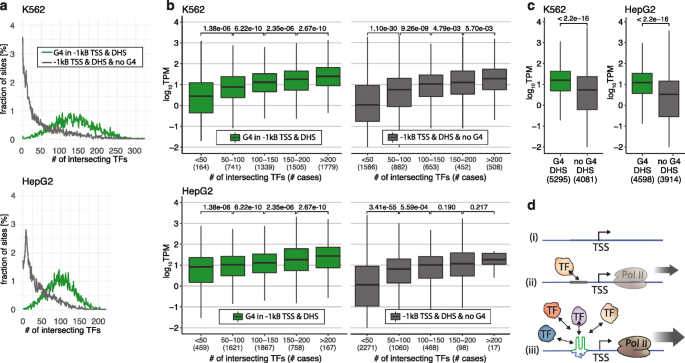
G4s是招募tf增强转录的枢纽。在面板gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba开放染色质上可达的启动子(上游TSS - 1 kb, DHS阳性)中的基因内源性G4用绿色表示,而缺乏内源性G4的启动子用灰色表示。gydF4y2Ba一个gydF4y2Ba内源性G4s标记了被tf高度占据的基因组区域。K562细胞(上)和HepG2细胞(下)中G4s与多种不同tf重叠的比例。gydF4y2BabgydF4y2Ba转录本水平的分布由K562细胞(上)和HepG2细胞(下)启动子中结合在G4s处或缺乏G4s的启动子处的tf数量划分(未配对Wilcoxon检验)。高TF占用率的案例数(括号内所示)对于G4s来说要高得多。gydF4y2BacgydF4y2Ba平均转录输出(以每百万转录数(TPM)显示)、日志gydF4y2Ba10gydF4y2BaK562细胞(左)和HepG2细胞(右)启动子中含有内源性G4s和不含有内源性G4s的基因的比较(未配对Wilcoxon试验)。gydF4y2BadgydF4y2Ba内源性G4s如何促进多个tf占据启动子的模型:(i)被抑制的启动子不被tf占据。(ii)双链DNA一致结合位点招募特定的tf到启动子,导致主动转录。(iii) G4s可以招募许多不同的tf,导致更活跃的转录基因gydF4y2Ba 全尺寸图像gydF4y2Ba |
【本文地址】
今日新闻 |
推荐新闻 |