|
目录
分类模型的评估模型优化与选择1.交叉验证2.网格搜索
【分类】K近邻算法【分类】朴素贝叶斯——文本分类实例:新闻数据分类
【分类】决策树和随机森林1.决策树2.决策树的算法3.代码实现实例:泰坦尼克号预测生死
【集成学习】随机森林1.集成学习2.随机森林3.学习算法4.代码实现5.优点
【分类】逻辑回归——二分类实例:良/恶性乳腺癌肿数据
【分类】SVM模型
分类模型的评估
模型优化与选择
1.交叉验证
交叉验证:为了让被评估的模型更加准确可信 交叉验证:将拿到的数据,分为训练和验证集。以下图为例:将数据分成5份,其中一份作为验证集。然后经过5次(组)的测试,每次都更换不同的验证集。即得到5组模型的结果,取平均值作为最终结果。又称5折交叉验证。

2.网格搜索
通常情况下,有很多参数是需要手动指定的(如k-近邻算法中的K值),这种叫超参数。但是手动过程繁杂,所以需要对模型预设几种超参数组合。每组超参数都采用交叉验证来进行评估。最后选出最优参数组合建立模型。

knn = KNeighborsClassifier()
# # fit, predict 预测, score 得出准确率
# knn.fit(x_train, y_train)
# # 得出预测结果
# y_predict = knn.predict(x_test)
# print("预测的目标签到位置为:", y_predict)
# # 得出准确率
# print("预测的准确率:", knn.score(x_test, y_test))
# 构造一些参数的值进行搜索
param = {"n_neighbors": [3, 5, 10]}
# 进行网格搜索
gc = GridSearchCV(knn, param_grid=param, cv=2)#二折交叉验证
gc.fit(x_train, y_train)
# 预测准确率
print("在测试集上准确率:", gc.score(x_test, y_test))
print("在交叉验证当中最好的结果:", gc.best_score_)
print("选择最好的模型是:", gc.best_estimator_)
print("每个超参数每次交叉验证的结果:", gc.cv_results_)
【分类】K近邻算法
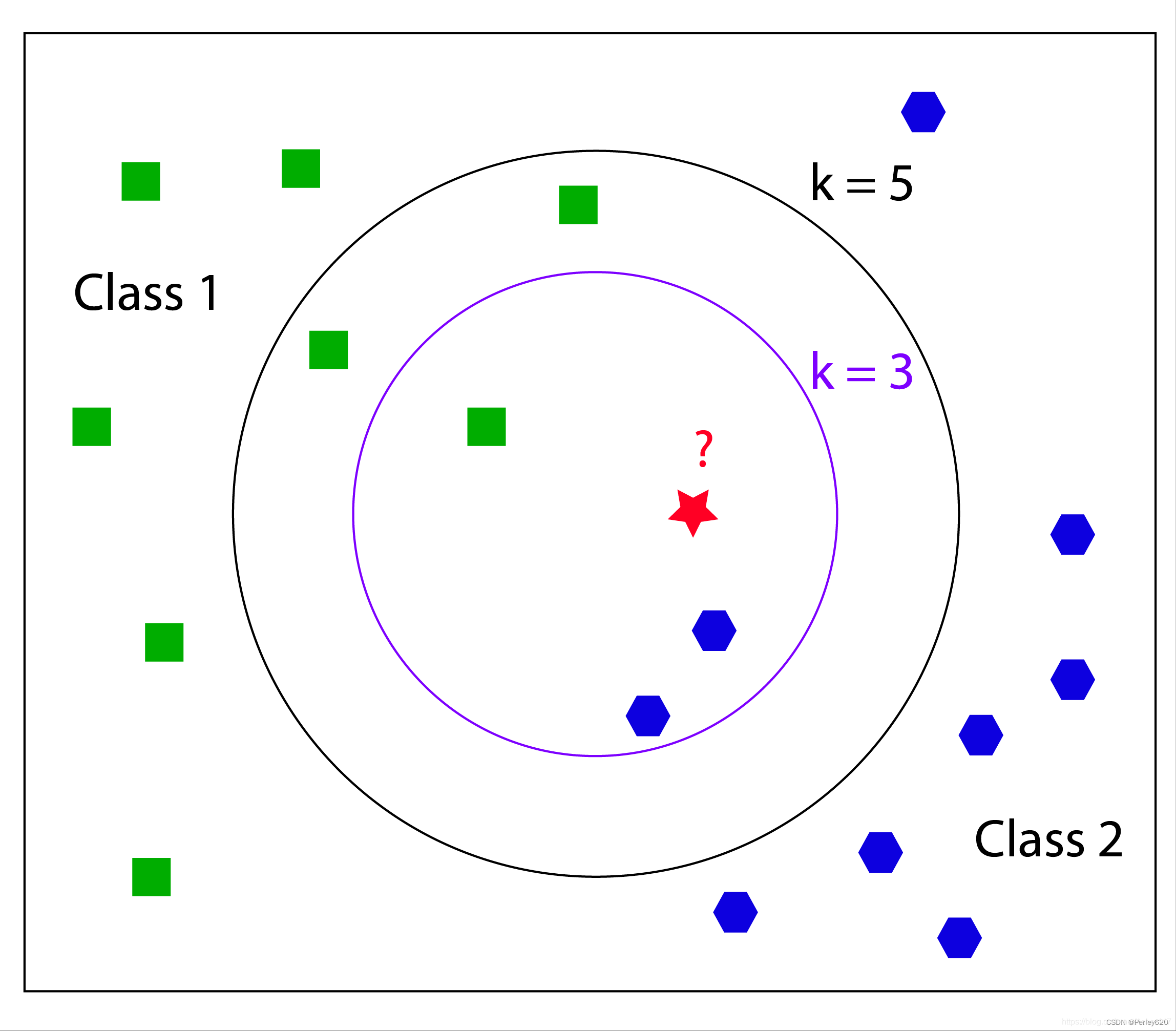 
sklearn.neighbors.KNeighborsClassifier(n_neighbors=5,algorithm='auto')

k值取多大?有什么影响?
k值取很小:容易受异常点影响k值取很大:容易受最近数据太多导致比例变化

from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.neighbors import KNeighborsClassifier
from sklearn.preprocessing import StandardScaler
if __name__ == "__main__":
decision()
def knncls():
"""
K-近邻预测用户签到位置
:return:None
"""
# 读取数据
data = pd.read_csv("./data/FBlocation/train.csv")
# print(data.head(10))
# 处理数据
# 1、缩小数据,查询数据晒讯
data = data.query("x > 1.0 & x < 1.25 & y > 2.5 & y < 2.75")
# 处理时间的数据
time_value = pd.to_datetime(data['time'], unit='s')
print(time_value)
# 把日期格式转换成 字典格式
time_value = pd.DatetimeIndex(time_value)
# 构造一些特征
data['day'] = time_value.day
data['hour'] = time_value.hour
data['weekday'] = time_value.weekday
# 把时间戳特征删除
data = data.drop(['time'], axis=1)#1表示列,0表示行
print(data)#没有时间戳特征的数据
# 把签到数量少于n个目标位置删除
place_count = data.groupby('place_id').count()
tf = place_count[place_count.row_id > 3].reset_index()#分组后逆操作,重新设置索引
data = data[data['place_id'].isin(tf.place_id)]
# 取出数据当中的特征值和目标值
y = data['place_id']#取目标值
x = data.drop(['place_id'], axis=1)#删除特征值就得到目标值
# 进行数据的分割训练集合测试集
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.25)
# 特征工程(标准化)
std = StandardScaler()
# 对测试集和训练集的特征值进行标准化
x_train = std.fit_transform(x_train)
x_test = std.transform(x_test)
# 进行算法流程 # 超参数
knn = KNeighborsClassifier()
# # fit, predict 预测, score 得出准确率
# knn.fit(x_train, y_train)
#
# # 得出预测结果
# y_predict = knn.predict(x_test)
#
# print("预测的目标签到位置为:", y_predict)
#
# # 得出准确率
# print("预测的准确率:", knn.score(x_test, y_test))
# 构造一些参数的值进行搜索
param = {"n_neighbors": [3, 5, 10]}
# 进行网格搜索
gc = GridSearchCV(knn, param_grid=param, cv=2)
gc.fit(x_train, y_train)
# 预测准确率
print("在测试集上准确率:", gc.score(x_test, y_test))
print("在交叉验证当中最好的结果:", gc.best_score_)
print("选择最好的模型是:", gc.best_estimator_)
print("每个超参数每次交叉验证的结果:", gc.cv_results_)
return None
【分类】朴素贝叶斯——文本分类
     优点: 优点:
朴素贝叶斯模型发源于古典数学理论,有稳定的分类效率。对缺失数据不太敏感,算法也比较简单,常用于文本分类。分类准确度高,速度快
缺点:
需要知道先验概率P(F1,F2,…|C),因此在某些时候会由于假设的先验模型的原因导致预测效果不佳。
实例:新闻数据分类
from sklearn.datasets import load_iris, fetch_20newsgroups, load_boston
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.feature_extraction.text import TfidfVectorizer
from sklearn.naive_bayes import MultinomialNB
from sklearn.metrics import classification_report
import numpy as pd
def naviebayes():
"""
朴素贝叶斯进行文本分类
:return: None
"""
news = fetch_20newsgroups(subset='all')
# 进行数据分割
x_train, x_test, y_train, y_test = train_test_split(news.data, news.target, test_size=0.25)
# 对数据集进行特征抽取
tf = TfidfVectorizer()
# 以训练集当中的词的列表进行每篇文章重要性统计['a','b','c','d']
x_train = tf.fit_transform(x_train)
print(tf.get_feature_names())
x_test = tf.transform(x_test)
# 进行朴素贝叶斯算法的预测
mlt = MultinomialNB(alpha=1.0)
print(x_train)
mlt.fit(x_train, y_train)
y_predict = mlt.predict(x_test)
print("预测的文章类别为:", y_predict)
# 得出准确率
print("准确率为:", mlt.score(x_test, y_test))
print("每个类别的精确率和召回率:", classification_report(y_test, y_predict, target_names=news.target_names))
return None
if __name__ == "__main__":
naviebayes()
【分类】决策树和随机森林
1.决策树
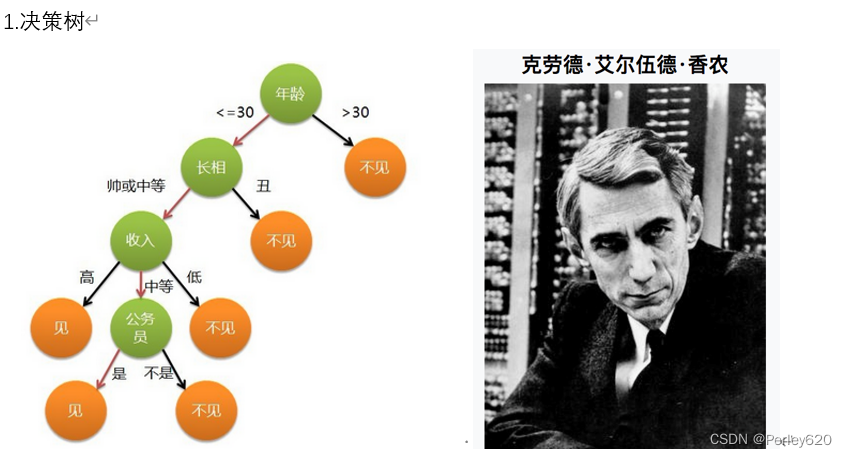 1.信息论 信息论的创始人,香农是密歇根大学学士,麻省理工学院博士。 1948年,香农发表了划时代的论文——通信的数学原理,奠定了现代信息论的基础 信息的单位:比特 32支球队,log32=5比特 64支球队,log64=6比特 1.信息论 信息论的创始人,香农是密歇根大学学士,麻省理工学院博士。 1948年,香农发表了划时代的论文——通信的数学原理,奠定了现代信息论的基础 信息的单位:比特 32支球队,log32=5比特 64支球队,log64=6比特
 
2.决策树的算法
ID3 信息增益 最大的准则 C4.5 信息增益比 最大的准则 CART 回归树: 平方误差 最小 分类树: 基尼系数 最小的准则 在sklearn中可以选择划分的原则 基尼系数:划分更仔细
3.代码实现
 优点: 优点:
简单的理解和解释,树木可视化。需要很少的数据准备,其他技术通常需要数据归一化,
缺点:
决策树学习者可以创建不能很好地推广数据的过于复杂的树,这被称为过拟合。决策树可能不稳定,因为数据的小变化可能会导致完全不同的树生成
改进:
减枝cart算法(决策树API中已经实现)随机森林
实例:泰坦尼克号预测生死
泰坦尼克号数据 在泰坦尼克号和titanic2数据帧描述泰坦尼克号上的个别乘客的生存状态。在泰坦尼克号的数据帧不包含从剧组信息,但它确实包含了乘客的一半的实际年龄。关于泰坦尼克号旅客的数据的主要来源是百科全书Titanica。这里使用的数据集是由各种研究人员开始的。其中包括许多研究人员创建的旅客名单,由Michael A. Findlay编辑。
我们提取的数据集中的特征是票的类别,存活,乘坐班,年龄,登陆,home.dest,房间,票,船和性别。乘坐班是指乘客班(1,2,3),是社会经济阶层的代表。
其中age数据存在缺失。
  
from sklearn.model_selection import train_test_split, GridSearchCV
import pandas as pd
from sklearn.tree import DecisionTreeClassifier, export_graphviz
def dieornot():
"""
泰坦尼克生与死
:return: None
"""
data=pd.read_csv("http://biostat.mc.vanderbilt.edu/wiki/pub/Main/DataSets/titanic.txt")
# print(data.info())
x=data[['pclass', 'age', 'sex']]
y=data['survived']
#缺失年龄数据处理
x["age"].fillna(x["age"].mean(),inplace=True)
# print(x)
#划分训练和测试集
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.25)
# 进行处理(特征工程)特征-》类别-》one_hot编码
from sklearn.feature_extraction import DictVectorizer
dict = DictVectorizer(sparse=False)
x_train = dict.fit_transform(x_train.to_dict(orient="records"))
# print(x_train)
print(dict.get_feature_names())#输出编码后的特征
x_test = dict.fit_transform(x_test.to_dict(orient="records"))
##########################################
# # 用决策树进行预测
# dec = DecisionTreeClassifier()
# dec.fit(x_train, y_train)
# # 预测准确率
# print("预测的准确率:", dec.score(x_test, y_test))
# # 导出决策树的结构
# export_graphviz(dec, out_file="./tree.dot", feature_names=['年龄', 'pclass=1st', 'pclass=2nd', 'pclass=3rd', '女性', '男性'])
###########################################
from sklearn.ensemble import RandomForestClassifier
rf = RandomForestClassifier()
param = {"n_estimators": [120, 200, 300, 500, 800, 1200], "max_depth": [5, 8, 15, 25, 30]}
# 网格搜索与交叉验证
gc = GridSearchCV(rf, param_grid=param, cv=2)
gc.fit(x_train, y_train)
print("准确率:", gc.score(x_test, y_test))
print("查看选择的参数模型:", gc.best_params_)
return None
if __name__ == "__main__":
dieornot()
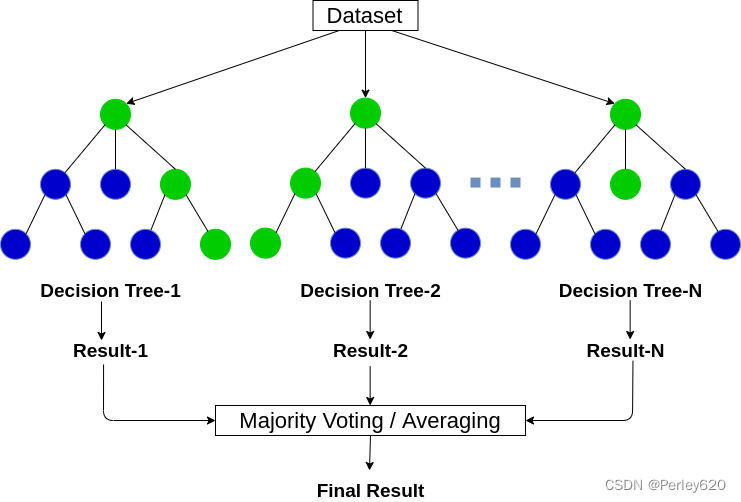
【集成学习】随机森林

1.集成学习
集成学习通过建立几个模型组合的来解决单一预测问题。它的工作原理是生成多个分类器/模型,各自独立地学习和作出预测。这些预测最后结合成单预测,因此优于任何一个单分类的做出预测。
2.随机森林
定义:在机器学习中,随机森林是一个包含多个决策树的分类器,并且其输出的类别是由个别树输出的类别的众数而定
3.学习算法

4.代码实现

from sklearn.ensemble import RandomForestClassifier
rf = RandomForestClassifier()
param = {"n_estimators": [120, 200, 300, 500, 800, 1200], "max_depth": [5, 8, 15, 25, 30]}
# 网格搜索与交叉验证
gc = GridSearchCV(rf, param_grid=param, cv=2)
gc.fit(x_train, y_train)
print("准确率:", gc.score(x_test, y_test))
print("查看选择的参数模型:", gc.best_params_)
return None
5.优点
决策树改进:
减枝cart算法(决策树API中已经实现)随机森林
优点:
在当前所有算法中,具有极好的准确率能够有效地运行在大数据集上能够处理具有高维特征的输入样本,而且不需要降维能够评估各个特征在分类问题上的重要性对于缺省值问题也能够获得很好得结果
【分类】逻辑回归——二分类
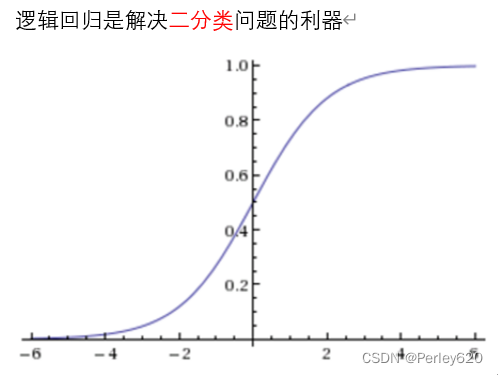 
哪个类别少,判定概率值是值得这个类别 (恶性:正例;良性:反例)
 应用:广告点击率预测、电商购物搭配推荐 优点:适合需要得到一个分类概率的场景 缺点:当特征空间很大时,逻辑回归的性能不是很好(看硬件能力) 应用:广告点击率预测、电商购物搭配推荐 优点:适合需要得到一个分类概率的场景 缺点:当特征空间很大时,逻辑回归的性能不是很好(看硬件能力)
实例:良/恶性乳腺癌肿数据
原始数据的下载地址: https://archive.ics.uci.edu/ml/machine-learning-databases/
数据描述 (1)699条样本,共11列数据,第一列用语检索的id,后9列分别是与肿瘤相关的医学特征,最后一列表示肿瘤类型的数值。 (2)包含16个缺失值,用”?”标出。
 
from sklearn.linear_model import LinearRegression, SGDRegressor, Ridge, LogisticRegression
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import mean_squared_error, classification_report
from sklearn.externals import joblib
import pandas as pd
import numpy as np
def ljhg():
"""
逻辑回归预测肿瘤的分类问题
:return: None
"""
# 获取数据
column = ['Sample code number', 'Clump Thickness', 'Uniformity of Cell Size','Uniformity of Cell Shape', 'Marginal Adhesion', 'Single Epithelial Cell Size','Bare Nuclei', 'Bland Chromatin', 'Normal Nucleoli', 'Mitoses', 'Class']
lb = pd.read_csv(r"F:\python\data\breast-cancer-wisconsin.data",names=column)
print(lb.shape[1])
#缺失数据处理
lb=lb.replace(to_replace="?", value =np.nan)
lb.dropna(inplace=True)
# 分割数据集到训练集和测试集 # 按照列名取数据
x_train, x_test, y_train, y_test = train_test_split(lb[column[1:10]], lb[column[10]], test_size=0.25)
# 进行标准化处理 ## 不需要对目标值进行标准化
std = StandardScaler()
x_train = std.fit_transform(x_train)
x_test = std.transform(x_test)
lj=LogisticRegression(C = 1.0)
lj.fit(x_train, y_train)
print(lj.coef_)
y_predict = lj.predict(x_test)
print("准确率为:", lj.score(x_test, y_test))
print("每个类别的精确率和召回率:", classification_report(y_test, y_predict,labels=[2, 4], target_names=["良性", "恶性"]))
return None
if __name__ == '__main__':
ljhg()
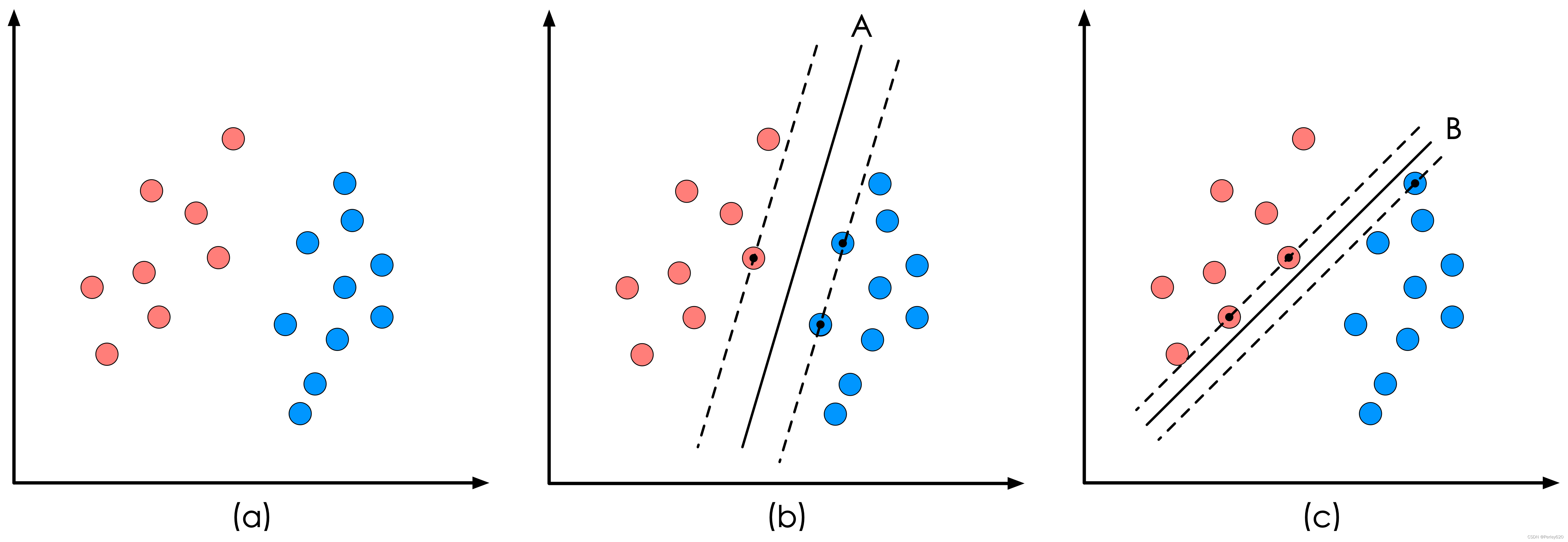
【分类】SVM模型
SVC(C=1.0, cache_size=200, class_weight=None, coef0=0.0,
decision_function_shape='ovr', degree=3, gamma='auto_deprecated',
kernel='rbf', max_iter=-1, probability=False, random_state=None,
shrinking=True, tol=0.001, verbose=False)

## 加载所需的函数,
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.svm import SVC
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
cancer = load_breast_cancer()
cancer_data = cancer['data']
cancer_target = cancer['target']
cancer_names = cancer['feature_names']
## 将数据划分为训练集测试集
cancer_data_train,cancer_data_test, \
cancer_target_train,cancer_target_test = \
train_test_split(cancer_data,cancer_target,
test_size = 0.2,random_state = 22)
## 数据标准化
stdScaler = StandardScaler().fit(cancer_data_train)
cancer_trainStd = stdScaler.transform(cancer_data_train)
cancer_testStd = stdScaler.transform(cancer_data_test)
## 建立SVM模型
svm = SVC().fit(cancer_trainStd,cancer_target_train)
print('建立的SVM模型为:\n',svm)
## 预测训练集结果
cancer_target_pred = svm.predict(cancer_testStd)
print('预测前20个结果为:\n',cancer_target_pred[:20])
## 求出预测和真实一样的数目
true = np.sum(cancer_target_pred == cancer_target_test )
print('预测对的结果数目为:', true)
print('预测错的的结果数目为:', cancer_target_test.shape[0]-true)
print('预测结果准确率为:', true/cancer_target_test.shape[0])
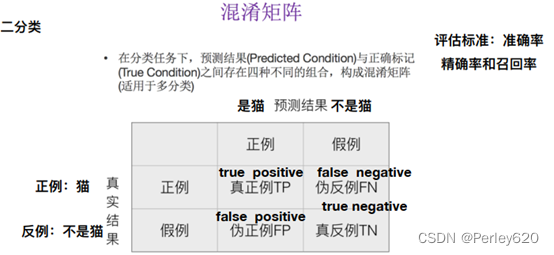
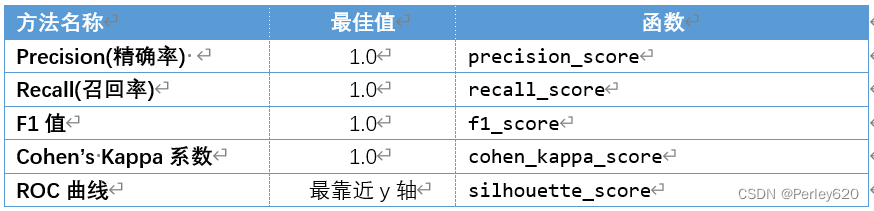
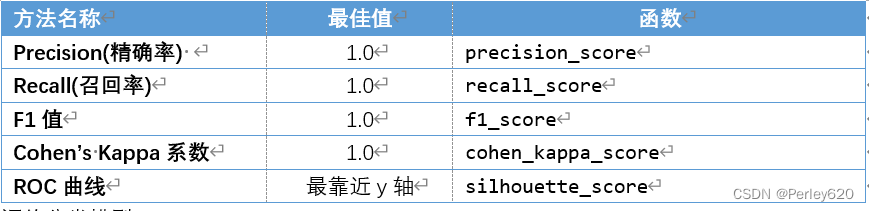 评价分类模型 评价分类模型
#接上页,评价分类模型
from sklearn.metrics import accuracy_score,precision_score, \
recall_score,f1_score,cohen_kappa_score
print('使用SVM预测breast_cancer数据的准确率为:',
accuracy_score(cancer_target_test,cancer_target_pred))
print('使用SVM预测breast_cancer数据的精确率为:',
precision_score(cancer_target_test,cancer_target_pred))
print('使用SVM预测breast_cancer数据的召回率为:',
recall_score(cancer_target_test,cancer_target_pred))
print('使用SVM预测breast_cancer数据的F1值为:',
f1_score(cancer_target_test,cancer_target_pred))
print('使用SVM预测breast_cancer数据的Cohen’s Kappa系数为:',
cohen_kappa_score(cancer_target_test,cancer_target_pred))
#输出分类模型评价报告函数
from sklearn.metrics import classification_report
print('使用SVM预测iris数据的分类报告为:','\n',
classification_report(cancer_target_test,
cancer_target_pred))
from sklearn.metrics import roc_curve
import matplotlib.pyplot as plt
## 求出ROC曲线的x轴和y轴
fpr, tpr, thresholds = \
roc_curve(cancer_target_test,cancer_target_pred)
plt.figure(figsize=(10,6))
plt.xlim(0,1) ##设定x轴的范围
plt.ylim(0.0,1.1) ## 设定y轴的范围
plt.xlabel('False Postive Rate')
plt.ylabel('True Postive Rate')
plt.plot(fpr,tpr,linewidth=2, linestyle="-",color='red')
plt.show()
import pandas as pd
from sklearn.svm import SVC
from sklearn.metrics import classification_report
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
abalone = pd.read_csv('F:\\python\\abalone.data',sep=',')
## 将数据和标签拆开
abalone_data = abalone.iloc[:,:8]
abalone_target = abalone.iloc[:,8]
## 连续型特征离散化
sex = pd.get_dummies(abalone_data['sex'])
abalone_data = pd.concat([abalone_data,sex],axis = 1 )
abalone_data.drop('sex',axis = 1,inplace = True)
## 划分训练集,测试集
abalone_train,abalone_test, \
abalone_target_train,abalone_target_test = \
train_test_split(abalone_data,abalone_target,
train_size = 0.8,random_state = 42)
## 标准化
stdScaler = StandardScaler().fit(abalone_train)
abalone_std_train = stdScaler.transform(abalone_train)
abalone_std_test = stdScaler.transform(abalone_test)
## 建模
svm_abalone = SVC(kernel = 'linear',C=1).fit(abalone_std_train,abalone_target_train)
#改变核函数的方法
print('建立的SVM模型为:','\n',svm_abalone )
# 代码 6-23
abalone_target_pred = svm_abalone.predict(abalone_std_test)
print('abalone数据集的SVM分类报告为:\n',
classification_report(abalone_target_test,abalone_target_pred))
|















 优点:
优点:
 1.信息论 信息论的创始人,香农是密歇根大学学士,麻省理工学院博士。 1948年,香农发表了划时代的论文——通信的数学原理,奠定了现代信息论的基础 信息的单位:比特 32支球队,log32=5比特 64支球队,log64=6比特
1.信息论 信息论的创始人,香农是密歇根大学学士,麻省理工学院博士。 1948年,香农发表了划时代的论文——通信的数学原理,奠定了现代信息论的基础 信息的单位:比特 32支球队,log32=5比特 64支球队,log64=6比特

 优点:
优点:






 应用:广告点击率预测、电商购物搭配推荐 优点:适合需要得到一个分类概率的场景 缺点:当特征空间很大时,逻辑回归的性能不是很好(看硬件能力)
应用:广告点击率预测、电商购物搭配推荐 优点:适合需要得到一个分类概率的场景 缺点:当特征空间很大时,逻辑回归的性能不是很好(看硬件能力)



 评价分类模型
评价分类模型