R语言数据可视化 |
您所在的位置:网站首页 › r语言数据框架 › R语言数据可视化 |
R语言数据可视化
|
在基本统计中,需要比较两组或多组数值之间的差异。在可视化图表中添加P值或显著性标志能更直观的显示比较数值之间的差异。本文基于ggpubr包进行演示。 1、加载包 library(ggplot2) library(ggpubr)在ggpubr包中有两个函数可以进行计算两组或多组数据间的比较, compare_means() stat_compare_means( ) 2、两个独立样本t 检验 compare_means(len ~ supp, data = ToothGrowth) ## # A tibble: 1 × 8 ## .y. group1 group2 p p.adj p.format p.signif method ## ## 1 len OJ VC 0.0645 0.064 0.064 ns Wilcoxon ggboxplot(ToothGrowth, x = "supp", y = "len", fill = "supp", palette = "npg", add = "jitter")+ stat_compare_means() #添加p值,默认为 wilcoxon 检验 #更改为 t 检验 ggboxplot(ToothGrowth, x = "supp", y = "len", fill = "supp", palette = "npg", add = "jitter")+ stat_compare_means(method=“t.test”) 3、配对样本t检验 ggpaired(ToothGrowth, x = "supp", y = "len", fill = "supp", line.color = "gray", line.size = 0.8, palette = "jco")+ stat_compare_means(paired = TRUE)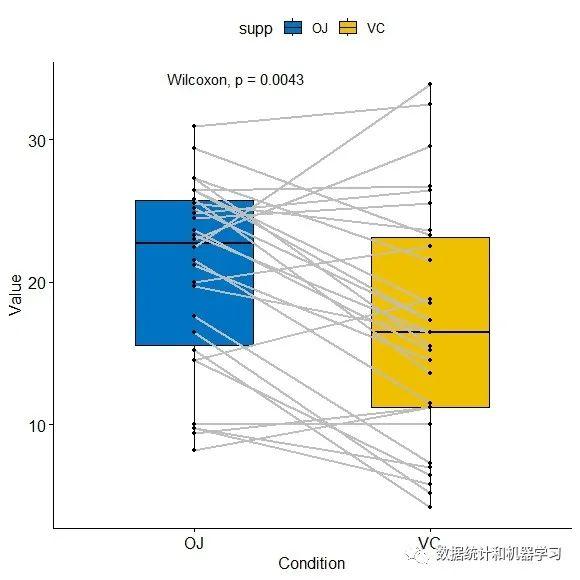 4、多组之间的比较 #全局检验 compare_means(Sepal.Length ~ Species, data = iris, method = "anova") ## # A tibble: 1 × 6 ## .y. p p.adj p.format p.signif method ## ## 1 Sepal.Length 1.67e-31 1.7e-31如果分组变量包含两个以上的水平,两两比较的检验(pairwise test)将自动执行。默认方法为“wilcox.test”,也可设置为“t.test”。 #两两比较 compare_means(Sepal.Length ~ Species,data = iris) ## # A tibble: 3 × 8 ## .y. group1 group2 p p.adj p.format p.signif method ## ## 1 Sepal.Length setosa versicolor 8.35e-14 1.7e-13 8.3e-14 **** Wilcoxon ## 2 Sepal.Length setosa virginica 6.40e-17 1.9e-16 < 2e-16 **** Wilcoxon ## 3 Sepal.Length versicolor virginica 5.87e- 7 5.9e- 7 5.9e-07 **** Wilcoxon #设置需要比较的组 comparisons #设置显著性水平 ggboxplot(iris, x = "Species", y = "Sepal.Length", fill= "Species", palette = "jco")+ stat_compare_means(comparisons = comparisons, label = "p.signif", #设置星标 label.y = c(7,7.5,8))+ # 分组比较的p值 stat_compare_means(label.y = 8.5)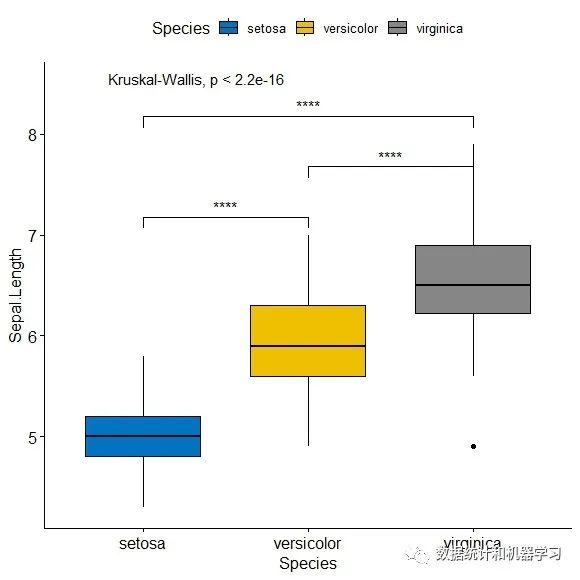 分面比较 ggboxplot(ToothGrowth, x = "supp", y = "len", fill = "supp", palette = "jco", add = "jitter", facet.by = "dose", short.panel.labs = FALSE)+ stat_compare_means(label = "p.format")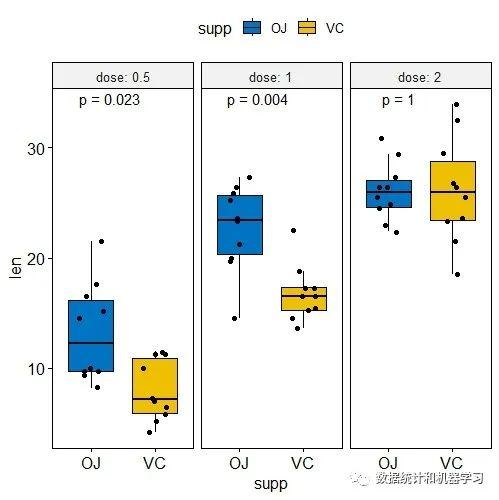 分组后配对样本比较 compare_means(len ~ supp, data = ToothGrowth, group.by = "dose", paired = TRUE) ggpaired(ToothGrowth, x="supp", y="len", fill="supp", palette = "jco", line.color = "grey", ine.size =0.4, facet.by = "dose", short.panel.labs = FALSE) + stat_compare_means(label = "p.format", paired = TRUE) 参考资料:http://www.sthda.com/english/rpkgs/ggpubr 更多R语言的知识请关注公众号【PRLearning】数据统计和机器学习 进行交流学习。公众号后台回复“显著性P值”索取代码。如果对您有帮助请关注、点赞、转发。 |
【本文地址】
今日新闻 |
推荐新闻 |