生存分析 |
您所在的位置:网站首页 › rossi是什么意思中文 › 生存分析 |
生存分析
|
笔者看到lifelines的文档里面涵盖的生存分析的模块以及讲解,比能查到的任何地方都要完整,所以 把这个库作为学习素材。 当然,另外也可以参考一下SPSS软件里面的模块,也是非常经典的一些学习对象。 文章目录 0 lifelines介绍0.1 lifelines几个重要方法0.2 完整的生存分析流程 1 生存概率估计1.1 非参数:KM数据样式——durations1.2 绘制KM曲线1.3 分组KM图1.4 分组KM曲线差异指标1.4.1 中位数50%存活率1.4.2 Logrank检验 1.5 参数估计:生存函数 2 Cox 比例风险回归模型2.1 数据集加载2.2 比例风险Cox回归2.3 比例cox回归中协变量值如何影响生存曲线 3 非比例风险模型——时变风险:Time varying survival regression3.1 Aalen’s Additive model 模型3.2 COX风险时变模型:Cox’s time varying proportional hazard model3.2.1 数据样式3.2.2 CoxTimeVaryingFitter 建模 3.3 完整 比例cox -> CoxTimeVarying 探索建模过程3.3.1 数据加载3.3.2 先构建比例COX模型3.3.3 检验PH假定:proportional_hazard_test3.3.4 分类变量未沟通PH检验处理方式:strata分层变量3.3.5 连续变量未沟通PH检验处理方式一:strata分层变量3.3.6 连续变量未沟通PH检验处理方式二:修改cox公式3.3.7 连续变量未沟通PH检验处理方式三:分段数据集 3.4 PH检验(proportional hazard assumption)是否一定需要? 4 非比例风险模型—— 参数估计模型 Weibull AFT model5 lifelines相关工具函数5.1 寿命表制作函数——survival_table_from_events5.2 KM曲线数据样式制作函数5.3 COX 时变回归模型中的 数据样式制作函数5.3.1 第一种:add_covariate_to_timeline5.3.2 第二种:to_episodic_format 6 累计风险函数:Cumulative hazard function6.1 非参数估计:Nelson-Aalen 累计风险函数图6.2 参数 估计:其他累计风险参数估计模型 7 如何选择最佳的参数估计模型7.1 QQ图7.2 AIC图 8 模型一致性、准确率以及校准8.1 模型一致性8.2 模型校准性8.3 精准校验值:brier_score_loss 8 其他本系列的续作: 生存分析——快手的基于深度学习框架的集成⽣存分析软件KwaiSurvival(一)生存分析——KM生存曲线、hazard比例、PH假定检验、非比例风险模型(分层/时变/参数模型)(二)生存分析——跟着lifelines学生存分析建模(三) 0 lifelines介绍github地址:CamDavidsonPilon/lifelines 文档地址:lifelines 生存分析最初是由精算师和医学界大量开发和应用的。它的目的是回答为什么事件发生在现在,而不是在不确定的情况下(事件可能涉及死亡,疾病缓解等)。 这对那些对测量寿命感兴趣的研究人员来说是件好事:他们可以回答诸如什么因素可能影响死亡之类的问题。但在医学和精算科学之外,还有许多其他有趣和令人兴奋的生存分析应用。 例如: SaaS提供商感兴趣的是度量订阅者的生命周期,即首次操作所需的时间库存缺货是对商品真正“需求”的审查事件。社会学家对衡量政党的寿命、关系或婚姻感兴趣A/B测试是为了确定不同的团队执行一个动作需要多长时间。还有用来判断用户流失以及快手有一篇来判定用户活跃度(本质也是从判定流失开始) 0.1 lifelines几个重要方法在生存分析——KM生存曲线、hazard比例、PH假定检验、非比例风险模型(分层/时变/参数模型)(二)提及到几个,这里笔者自己总结一下lifelines中几个比较核心的: 模块描述类型方法survival function研究对象从试验开始直到某个特定时间点仍然存活的概率参数估计Exponential, Log-Logistic, Log-Normal and Splines非参数估计Kaplan-Meier估计cumulative hazard风险函数的估计值参数估计Exponential, Log-Logistic, Log-Normal and Splines非参数估计Nelson-Aalen估计Survival regression会加入额外的协变量(如年龄、国家等)与另一个变量进行回归比例回归Cox 比例风险回归模型,指数回归模型 ,Weibull回归模型,Poisson回归模型非比例回归含参数与半参模型:Aalen’s Additive model 模型 、 CoxTimeVarying时变模型、AFT(accelerated failure time model)加速失效模型 0.2 完整的生存分析流程从Time-Dependent 生存模型分析用户流失来看一个完整的生存分析可归纳为: 原始数据格式处理:把数据处理为用户、生存时长、是否删失的数据格式。KM估计及生存曲线的绘制。判断协变量是否存在时变变量,如果有,进行数据格式的二次处理,将数据打断为用户、起始时间、结束时间、是否删失的格式。判断协变量系数是否存在时变效果,即著名的PH假设检验。如果检验不通过,对时变效果进行绘制,并基于绘制结果进行数据分层(stratify)。建立Extended Cox PH Model,对风险因子进行影响估计。一个比较好的案例可以参考:【3.3 完整 比例cox -> CoxTimeVarying 探索建模过程】 1 生存概率估计从这篇 数据分析系列:生存分析(生存曲线分析、Cox回归分析)——附生存分析python代码。开始来看一下lifelines实现KM方法,结合lifelines文档,来完整看看KM流程。 生存函数估计有非常多方法: 非参估计:Kaplan-Meier参数估计:WeibullFitter等 1.1 非参数:KM数据样式——durations数据集样式: from lifelines.datasets import load_waltons from lifelines import KaplanMeierFitter from lifelines.utils import median_survival_times import matplotlib.pyplot as plt # 数据载入 df = load_waltons() print(df.head(),'\n') print(df['T'].min(), df['T'].max(),'\n') print(df['E'].value_counts(),'\n') print(df['group'].value_counts(),'\n')
需要注意,该格式并非严格的寿命表,具体的转化为寿命表可以看[5.1 小节] 1.2 绘制KM曲线利用此数据取拟合拟生存分析中的Kaplan Meier模型(专用于估计生存函数的模型) # KM初始化 kmf = KaplanMeierFitter() kmf.fit(df['T'], event_observed=df['E']) # 全体: K-M 存活曲线 kmf.survival_function_ # km 生存概率 kmf.plot_survival_function()
这里来看几种画法: kmf.survival_function_ # km 生存概率 kmf.plot_survival_function()kmf.survival_function_给出了随着时间增加,KM生存估计概率的趋势。
这并不是我们关注的重点,我们真正要关注的实验组(存在病毒)和对照组(未存在病毒)的生存曲线差异。 因此我们要按照group等于“miR-137”和“control”分组,分别观察对应的生存曲线: 可以看到,带有miR-137病毒的生存曲线在control组下方。说明其平均存活时间明显小于control组。同时带有miR-137病毒存活50%对应的存活时间95%置信区间为[19,29],对应的control组为[56,60]。 差异较大,这个方法可以应用在分析用户流失等场景,比如我们对一组人群实行了一些防止流行活动,我们可以通过此种方式分析我们活动是否有效。 1.4 分组KM曲线差异指标除了直接看图,还可以用什么方式来区分不同组的KM曲线: 1.4.1 中位数50%存活率另外还有中位数50%存活率下存活时间的置信区间: median_ = kmf.median_survival_time_ median_confidence_interval_ = median_survival_times(kmf.confidence_interval_) print(median_confidence_interval_) >>> KM_estimate_lower_0.95 KM_estimate_upper_0.95 >>>0.5 53.0 58.0代表着,53-58天左右,存活着整体的一半人。 1.4.2 Logrank检验Logrank 检验的零假设是指两组的生存时间分布完全一致,当我们通过计算拒绝零假设时,就可以认为两组的生存时间分布存在统计学差异。 from lifelines.statistics import logrank_test T = df['T'] E = df['E'] dem = (df['group'] == 'miR-137') results = logrank_test(T[dem], T[~dem], E[dem], E[~dem], alpha=.99) results.print_summary() print(results.p_value) print(results.test_statistic)输出结果: t_0 = -1 null_distribution = chi squared degrees_of_freedom = 1 alpha = 0.99 test_name = logrank_test --- test_statistic p -log2(p) 122.25 |
【本文地址】
今日新闻 |
推荐新闻 |
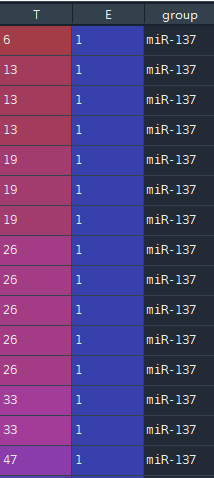 可以看到数据有三列,其中T代表min(T, C),其中T为死亡时间,C为观测截止时间。 E代表是否观察到“死亡”,1代表观测到了,0代表未观测到,即生存分析中的删失数据,共7个。 group代表是否存在病毒, miR-137代表存在病毒,control代表为不存在即对照组,根据统计,存在miR-137病毒人数34人,不存在129人。
可以看到数据有三列,其中T代表min(T, C),其中T为死亡时间,C为观测截止时间。 E代表是否观察到“死亡”,1代表观测到了,0代表未观测到,即生存分析中的删失数据,共7个。 group代表是否存在病毒, miR-137代表存在病毒,control代表为不存在即对照组,根据统计,存在miR-137病毒人数34人,不存在129人。 图中蓝色实线为生存曲线,浅蓝色带代表了95%置信区间。 随着时间增加,存活概率S(t)越来越小,这是一定的,同时S(t)=0.5时,t的95%置信区间为[53, 58]。
图中蓝色实线为生存曲线,浅蓝色带代表了95%置信区间。 随着时间增加,存活概率S(t)越来越小,这是一定的,同时S(t)=0.5时,t的95%置信区间为[53, 58]。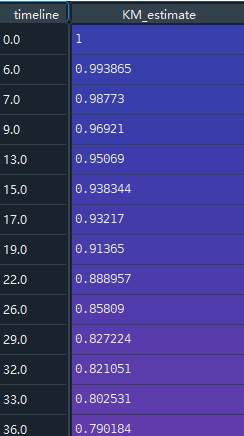 同时还可以在图中画出KM值随着时间上升,原始的删失值以及发生概率:
同时还可以在图中画出KM值随着时间上升,原始的删失值以及发生概率: 这里还可以画出一条,关于生存概率的,累计概率密度图:
这里还可以画出一条,关于生存概率的,累计概率密度图:
