我应该使用哪个最大似然树构建器? Geneious |
您所在的位置:网站首页 › phyML在线构建系统发育树教程 › 我应该使用哪个最大似然树构建器? Geneious |
我应该使用哪个最大似然树构建器? Geneious
|
Geneious 包含用于最大似然树构建器 PHYML、Garli、RAxML、PAUP* 和 FastTree 的插件。 在这篇文章中,我们将简要回顾每种数据集最适合哪种类型的数据集,哪种数据集最快,以及每种数据集都有哪些选择。 有关每个程序使用的算法的详细信息,请参阅程序的网站。 如果您要发布这些插件的结果,请记住引用您使用的程序的原始作者。 引文信息可以在相应的插件页面上找到。 每个程序的背景 1物理层 PHYML 由Stephane Guindon和他在法国蒙彼利埃大学 LIRMM 的同事编写。它于 2003 年首次发布,Geneious 插件使用 3.2 版本,本文对此进行了描述。 PHYML 因其简单、准确和速度而成为最著名的最大似然程序之一。 2RAxML RaxML 来自德国海德堡理论研究所的 Alexandros Stamatakis的Exelixis 实验室。它是为处理大型数据集而开发的,具有相对较低的内存消耗、高级搜索算法和加速可能性的使用。 Geneious 插件当前使用 RAxML 版本 8.2.7,因此下表中列出的功能适用于该版本。 3加利 Garli 由目前在堪萨斯大学的Derrick Zwickl编写和维护。它松散地基于程序 GAML ( Lewis 1998 )。 可以在此处找到该程序的文档。 *Note:Garli 在 Geneious Prime 2020 中不可用,因为不再支持 32 位应用程序 4PAUP* PAUP* 是由 Dave Swofford 编写的流行系统发育程序,可用于构建最大简约树、距离树和最大似然树。 本文中有关 PAUP* 的信息仅与最大似然树有关。PAUP*4.0b10 以前可从 Sinnauer Associates 购买,但目前正在进行重大更新。目前可以从这里获得免费的“测试”版本。 请注意,Geneious PAUP* 插件不包含程序本身,它仅提供用于运行您自己的 PAUP* 副本的界面。 首次在 Geneious 中运行插件时,您必须下载自己的 PAUP* 副本并设置可执行文件的路径。 该插件目前兼容旧的 4.0b10 版本,以及新的测试 alpha 版本(4.0a149 及以上) 4快树 FastTree 由劳伦斯伯克利国家实验室的Adam Arkin 小组的 Morgan N. Price 开发。 它针对多达 100 万个序列的超大比对进行了优化,并结合使用相邻连接、最小进化和最大似然来推断近似最大似然树。 这里给出了它是如何工作的详细描述,但总而言之,FastTree 使用邻居连接来获得一个近似的起始树,然后使用最小进化方法来减少树的长度,然后最大似然进一步改进树。Geneious 实现了 FastTree 2.1.5。 你可以用这些程序做什么? 所有程序都将根据 DNA 和蛋白质比对构建树,但是您从每个程序中获得的选项存在一些差异,总结在下表中。 请注意,PAUP* 将为蛋白质比对构建最大简约树和距离树,但不会构建最大似然树。
PHYML 和 PAUP* 为您提供最广泛的模型选择,能够输入 Modeltest 比较 DNA 数据的大多数模型。但是,请记住,这些模型中的大多数都嵌套在其他程序中实现的通用时间可逆 (GTR) 模型中。 PAUP* 包括 Modeltest,因此您可以选择将其作为树构建过程的一部分运行。 对于 PHYML 和其他程序,您需要在 Geneious 之外运行 jModeltest,然后在 Geneious 中手动配置适当的模型选项。 PHYML 还为您提供了多种计算支持度值的方法,但它对分类群的数量有一个内置的限制。 我不知道 Garli、PAUP* 和 RAxML 有类似的数据集大小限制(尽管正如您将在下面看到的那样,FastTree 对于真正大的数据集的性能都优于 FastTree)。 RAxML 和 PAUP* 允许您对数据进行分区,例如,如果您希望估计不同密码子位置或基因的不同比率。 在 PAUP* 中,这是通过编辑自定义命令块来完成的 - 请参阅PAUP* 命令行指南以获取您可以通过这种方式实现的选项的完整列表。 关于这些程序如何在 Geneious 中运行的简要说明 这些插件不在 Geneious Java 运行时环境中运行,因此它们不使用分配给 Geneious 的 RAM。相反,它们作为独立程序运行,Geneious 提供了一个界面。 Geneious 将您的文件导出到插件,插件程序运行,然后将结果导入回 Geneious。 尽管树构建过程本身不使用分配给 Geneious 的 RAM,但您确实需要为 Geneious 分配足够的 RAM 才能处理文件的导出/导入 - 对于大文件,这可能需要大量内存。 哪个最快? 这个问题的答案很大程度上取决于您拥有的数据集类型。 作为一个非常普遍的规则,速度是这样的: FastTree >> RAxML > PHYML > Garli >> PAUP*。 FastTree 是迄今为止对于具有大量分类群的大树的最快算法。 FastTree 可以在几分钟内生成具有支持值的 10,000 个分类单元树,而由 RAxML 或 Garli 构建的同一棵树可能需要几天才能运行。 PHYML 甚至不会在这么大的对齐方式上运行,因为它内置了 4000 个分类群的截止值。 然而,FastTree 生成的树是“近似最大似然”树,对于分类群之间关系不那么明确的数据集,它们可能不如其他方法生成的树那么准确,这些方法对树执行更密集的搜索拓扑(有关 FastTree 与 PHYML 与 RAxML 的速度和准确性的更深入讨论,请参阅 FastTree 网站)。 如果您有非常长的序列,但只有几个分类群(例如,如果您从少量细菌基因组构建一棵树),那么 RAxML 和 PHYML 的性能会优于 FastTree。 一个由五个序列组成的树,长度为 400 万个碱基(在没有支持值的情况下计算)在 FastTree 中花费了大约 14 分钟,而在 RaxML 和 PHYML 中仅花费了大约 1 分钟。 Garli 不能很好地处理长序列,最适合用于较短的比对。 在完整的最大似然树构建器中,RAxML 似乎对来自 DNA 数据的大树最有效。 对于较小的数据集,PHYML 是一个不错的选择,因为根据 PHYML 手册,PhyML 的“舒适区”通常位于大约 100-200 个长度小于 2,000 个字符的序列。 PHYML 网站使用一系列数据集对 PHYML 和 RAxML 进行了一些广泛的比较。 PAUP* 是最大似然树构建器中最慢的,尤其是在使用默认选项运行时。PAUP* 默认使用树二分和重新连接 (TBR) 进行拓扑搜索,与 PHYML(NNI,最近邻交换)或 RAxML(快速爬山)中的默认拓扑搜索选项相比,它可以评估更多的树。 要将 PAUP* 配置为使用 NNI 而不是 PBR,请打开自定义命令块并将 SWAP=NNI 添加到 HSEARCH 行。 这将大大加快速度,但速度仍然无法接近 PHYML 或 RAxML 的速度。 我怎样才能让我的树跑得更快? 简短的回答是获得更快的计算机。 为树构建器提供更多 RAM 不一定会加快速度,但可能意味着您可以构建更大的树而不会耗尽内存。 速度主要由处理器的速度决定,目前这里提到的所有树构建器都只使用单个处理器,不能配置为跨多个内核运行。 那么,哪种树最好呢? 这个问题没有一个答案,因为它完全取决于数据集的性质,以及所选模型与数据的拟合程度。给定数据和您选择的模型,最大似然树构建器返回正确概率最高的树,但由于算法的差异,无法直接比较每个程序产生的似然值。使用一种以上的树构建方法来评估树拓扑的稳健性是一种很好的做法。
|
【本文地址】
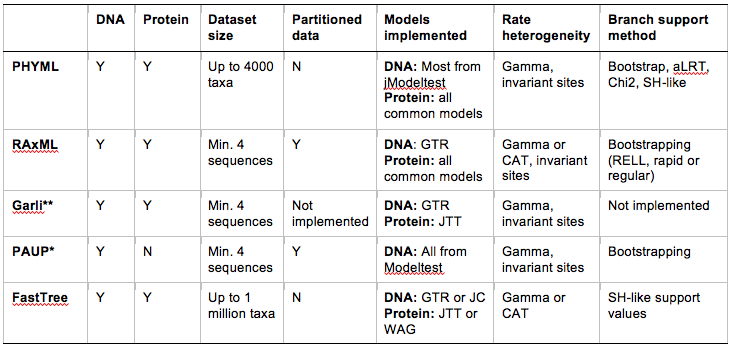 **由于Garli 的设置方式,目前Geneious 插件中仅实现了GTR+G+I 模型的默认选项且没有引导。但是,如果您想要不同的选项,例如引导或分区,请联系支持,或者您可以根据 Garli 文档自己编辑 Garli 配置文件(位于插件文件夹中)。
**由于Garli 的设置方式,目前Geneious 插件中仅实现了GTR+G+I 模型的默认选项且没有引导。但是,如果您想要不同的选项,例如引导或分区,请联系支持,或者您可以根据 Garli 文档自己编辑 Garli 配置文件(位于插件文件夹中)。