【耀文解读】mRNA篇 |
您所在的位置:网站首页 › page电泳结果分析方法 › 【耀文解读】mRNA篇 |
【耀文解读】mRNA篇
|
来源:雪球App,作者: 江苏耀海生物CDMO,(https://xueqiu.com/9950954021/228838634)
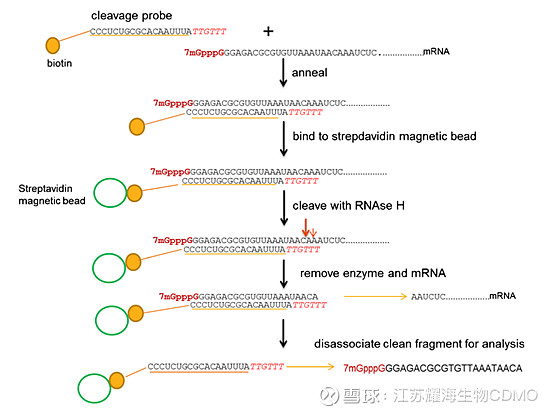
#RNA疗法# #CDMO# #CMO# 目前mRNA技术的应用仍处于科学前沿,在开发和生产过程中mRNA质量相关的监管法规和行业标准仍处于发展阶段。2020年NMPA发布的《新型冠状病毒预防用mRNA疫苗药学研究技术指导原则》中,建议对DNA模板、mRNA原液及成品LNP-mRNA进行质量控制,包括mRNA的特有表征:加帽率、poly A尾长、模板DNA残留、双链RNA(dsRNA)残留等。 耀海生物现推出mRNA质量分析系列文章,以总结当前mRNA的质量表征方法。今天和大家分享加帽率的检测方法。Part 1将和大家分享加帽率的检测方法,后续内容包括mRNA的完整性与纯度、序列准确性、poly A尾、dsRNA残留等分析策略。 mRNA的帽结构可维持体内稳定性,促进蛋白的翻译。带帽结构的mRNA占比可影响mRNA的免疫原性和翻译效率,因此加帽率是mRNA疫苗或药物的关键质量指标。 加帽与未加帽的mRNA仅相差一个核苷酸(m7G),相对于完整的mRNA太小,难以区分。当前加帽率的检测思路为:采用酶切方式获得5’端加帽或不加帽的寡核苷酸序列,随后通过尿素变性聚丙烯酰胺凝胶电泳(PAGE)、高效液相色谱(HPLC)或液相质谱联用(LC-MS)等方法分离不同大小的寡核苷酸片段,从而定量评估mRNA的加帽率。 2016年诺华公司Beverly M等发表在Anal Bioanal Chem杂志的一篇文章,《Label-free analysis of mRNA capping efficiency using RNase H probes and LC-MS》,研究基于磁珠及RNAse H酶切,得到酶解后、带或不带帽结构的5’端寡核苷酸片段,使用液相色谱质谱联用(LC-MS)检测,对带或不带帽结构的组分进行质谱峰面积定量,从而得到5’加帽比例。该方法是当前工艺中常用的加帽率检测方法。 2022年,BioNTech公司Vlatkovic I等在Pharmaceutics发表文章《Ribozyme Assays to Quantify the Capping Efficiency of In Vitro-Transcribed mRNA》。该研究提出,由于RNase H切割位点的专一性不稳定,可能产生其他切割产物,影响结果的可靠性。为改善这一不足,Vlatkovic I等设计了5个核酶(ribozyme),可识别5’UTR特异位点并切割,得到5’端酶切片段。通过硅胶柱去除长链RNA后,纯化得到核酶和5’端酶切产物。随后通过尿素变性聚丙烯酰胺凝胶电泳或LC-MS分离/分析带或不带帽结构的5’酶切产物,从而计算加帽率。 一、基于RNase H的mRNA加帽率分析 体外转录(IVT)制备两种不同长度的mRNA(2.2 kb与9 kb)进行测试,其5’末端的前50个核苷酸序列相同,可使用同一种切割探针。使用两种加帽方法:牛痘加帽酶、ARCA(Cap 0)帽类似物。将制备得到的mRNA进行琼脂糖凝胶电泳,确认mRNA的长度和完整性。 退火反应体系包括500 pmol RNase H探针(生物素标记)、10×RNase H缓冲液和100 pmol mRNA,使用5倍量的探针以保证全部mRNA与探针结合,500 pmol是与磁珠结合的的探针最大量。反应条件为:将探针与mRNA混合,在95℃退火5 min,随后以2 min的间隔降至65℃、55℃、40℃,最后降至22℃。 磁珠结合与酶切 ①结合:将退火的mRNA和探针加入预处理过的链霉亲和素磁珠中,室温孵育30 min。加入50 U RNase H,吹打混匀后在37℃下孵育3小时,然后置于磁力架,弃上清。 ②清洗:用含1M NaCl的清洗缓冲液(5mM Tris-HCl, 0.5mM EDTA, pH7.5)洗涤3次以去除酶、mRNA和反应缓冲液;然后用蒸馏水洗涤3次以除去过量的盐。 ③洗脱:加入75%甲醇(80℃预热),于80℃孵育1 min洗脱mRNA酶切片段。 ④换液:通过旋转蒸发干燥洗脱产物,重悬于含1%甲醇的0.1 mM EDTA溶液,用于LC-MS分析。
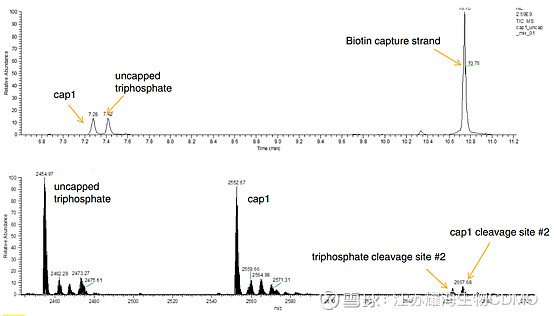
图1: 基于RNase H与磁珠,mRNA 5’端寡核苷酸的捕获 LC-MS分析 使用液相高分辨质谱联用仪,固定相为ACQUITY UPLC C18色谱柱,流动相A为200 mM六氟异丙醇+8.15 mM三乙胺(pH 7.9),流动相B为100%MeOH。洗脱条件为:5%B洗脱1min,1-12 min内B浓度由5%线性增加到25%,然后90%B冲洗1min后恢复5%B。紫外波长设置为260 nm。 LC-MS分离效果 与含有二磷酸、三磷酸或未甲基化G cap(GpppG)相比,含有甲基化M7GpppG帽结构的切割片段,其质量差异有助于加帽率的鉴定。如图2所示,借助反相色谱柱进行液相-质谱分析,可有效分离Cap 1与未加帽的三磷酸mRNA切割片段。除预期的寡核苷酸片段外,还存在RNase H第二个识别位点及生物素探针,生物素探针的出现可能是由于洗脱条件苛刻。
图2: 基于RNase H的加帽率分析. 上图为总离子色谱图; 下图为电喷雾质谱. 如上所述,文章介绍了一种定量分析mRNA加帽率的方法,且不需放射性同位素标记。该分析方法基于链霉亲和素磁珠特异性捕获生物素标记的探针,mRNA 5’端序列与探针结合,从而被RNase H识别并切割,经过清洗、洗脱得到mRNA 5’端单链寡核苷酸序列。然后通过LC-MS分离带或不带帽结构的寡核苷酸,从而得出mRNA的加帽率。 星耀小TIP: 2022年美国药典(USP)制定的《mRNA疫苗质量分析方法》草案中,基于RNase H切割作用,推荐RNase H与离子对反相高效液相色谱(IP-RP-HPLC),用于mRNA加帽率的测定:①使用RNase H特异性切割mRNA,以产生带或不带帽结构的5’端单链寡核苷酸序列;②通过IP-RP-HPLC分离寡核苷酸,使用C18柱、流动相A(0.1 mM冰醋酸三乙胺缓冲液pH 7.0),流动相B(A+25%乙腈);③根据带帽结构的寡核苷酸的峰面积占比计算加帽率。
二、基于核酶的mRNA加帽率分析 体外转录(IVT)制备不同长度的mRNA(100 nt至9.4 kb),使用两种加帽方法:牛痘加帽酶、ARCA(Cap 0)或CleanCap(Cap 1)帽类似物。通过琼脂糖凝胶电泳验证mRNA的质量。 靶向5’UTR的锤头型核酶 mRNA序列设计中常用的天然5’UTR序列为人α珠蛋白(hAg)和TEV病毒来源,基于这两种常见的5’UTR序列,研究设计了5条靶向性核酶。 注:核酶(ribozyme) 是一类具有酶特性的RNA分子,通过催化靶位点RNA链中磷酸二酯键的断裂,特异性地剪切底物RNA分子。文中基于常用的天然5’UTR序列设计了特异靶向性核酶,以扩大其适用范围。
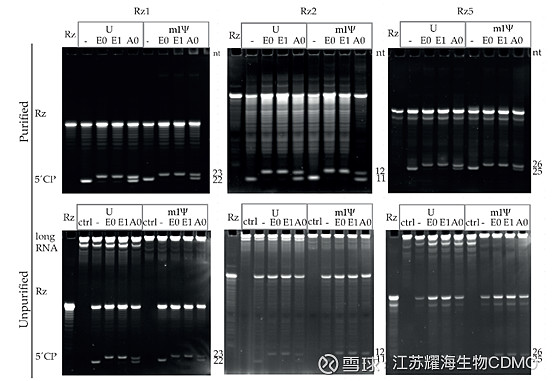
图3: 五种核酶的长度及切割位点 5’端短片段的切割与纯化 核酶与mRNA 5’ UTR特定区域退火结合,在镁离子存在下,发生核酶介导的酶切反应,产物包括10-30 nt的5’端短片段、3’端长片段、全长mRNA与核酶。其中5’端短片段带或不带帽结构,之间相差1个核苷酸。使用RNeasy Mini Kit RNA提取试剂盒,基于硅胶柱对核酶切割反应产物进行纯化,去除全长mRNA和3’端长片段,纯化得到的5’端短片段和核酶,用于后续的分析。 变性凝胶电泳分析加帽率 研究使用编码促红细胞生成素(EPO)的mRNA,使用尿素变性聚丙烯酰胺凝胶电泳(PAGE),通过带或不带帽结构的条带比例定量检测加帽率。结果显示,所有未加帽的mRNA的5’端短片段在变性胶底部22 nt处显示高亮条带,酶法加帽的mRNA 5’端短片段在23 nt处显示较亮条带。而ARCA共转录加帽的样品在变性胶底部存在两条带,即存在带和不带帽结构的5’端短片段。随后利用软件进行定量分析,以计算加帽率。这项研究中酶法加帽率为84-100%,而ARCA加帽率是34-77%,提示ARCA共转录加帽率低于酶法加帽。
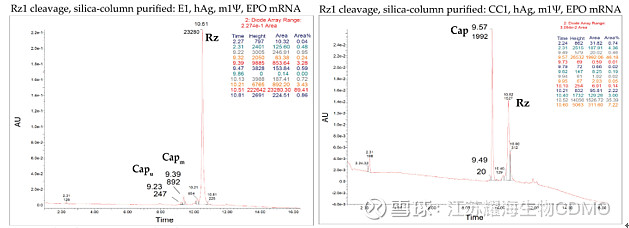
图4: 基于核酶的加帽率分析(尿素变性PAGE) 另外,研究发现,与核酶介导的酶切反应相比,RNase H酶切产物、包括纯化后的产物存在不同长度的长/短RNA条带,提示RNase H酶存在非特异性切割。 LC-MS分析加帽率。研究使用LC-MS对纯化后的5’端短片段进行定量分析,结果显示酶法加帽效率是89%,CleanCap共转录加帽的效率是99%,与尿素变性PAGE结果基本一致。
图5: 基于核酶的加帽率分析(LC-MS).左图为酶法加帽; 右图为共转录加帽. 三、结语 如文中所示,2016年诺华公司报道了一种无放射性标记的检测方法,使用生物素标记的RNase H切割探针,利用磁珠分离得到5’端寡核苷酸序列,随后通过LC-MS分析,有效分离加帽或不加帽的寡核苷酸序列,可定量分析mRNA样品的加帽率。 基于RNase H切割探针,结合HPLC或LC-MS分析,是当前工艺中常用的加帽率检测法,也是美国药典(USP)推荐的加帽率检测方法。但有报道显示,RNase H切割位点的专一性较差,可能影响结果的可靠性。为解决这一问题,BioNTech公司Vlatkovic I等开发出一种新的基于核酶的加帽率分析方法,筛选靶向5’UTR序列的切割核酶,经硅胶柱纯化后,进行变性PAGE或LC-MS,亦可有效分离带或不带帽结构的5’端寡核苷酸序列,实现mRNA加帽率的定量分析。且相比RNase H,核酶切割特异性较高,未产生非特异性条带,有望开发为研发或生产阶段mRNA的通用质控方法。 参考文献: [1] Beverly M, Dell A, Parmar P, et al. Label-free analysis of mRNA capping efficiency using RNase H probes and LC-MS. Anal Bioanal Chem. 2016 Jul;408(18):5021-30. doi: 10.1007/s00216-016-9605-x. [2] Vlatkovic I, Ludwig J, Boros G, et al. Ribozyme Assays to Quantify the Capping Efficiency of In Vitro-Transcribed mRNA. Pharmaceutics. 2022 Jan 29;14(2):328. doi: 10.3390/pharmaceutics14020328. [3] 药品审评中心. 新型冠状病毒预防用mRNA疫苗药学研究技术指导原则(试行). 2020. [4] USP. Analytical Procedures for mRNA Vaccines–Draft. 2022.
|
【本文地址】
今日新闻 |
推荐新闻 |