跟着iMeta学做图|分面箱线图展示alpha多样性并用标注差异分析结果 |
您所在的位置:网站首页 › origin怎么添加显著性差异 › 跟着iMeta学做图|分面箱线图展示alpha多样性并用标注差异分析结果 |
跟着iMeta学做图|分面箱线图展示alpha多样性并用标注差异分析结果
|
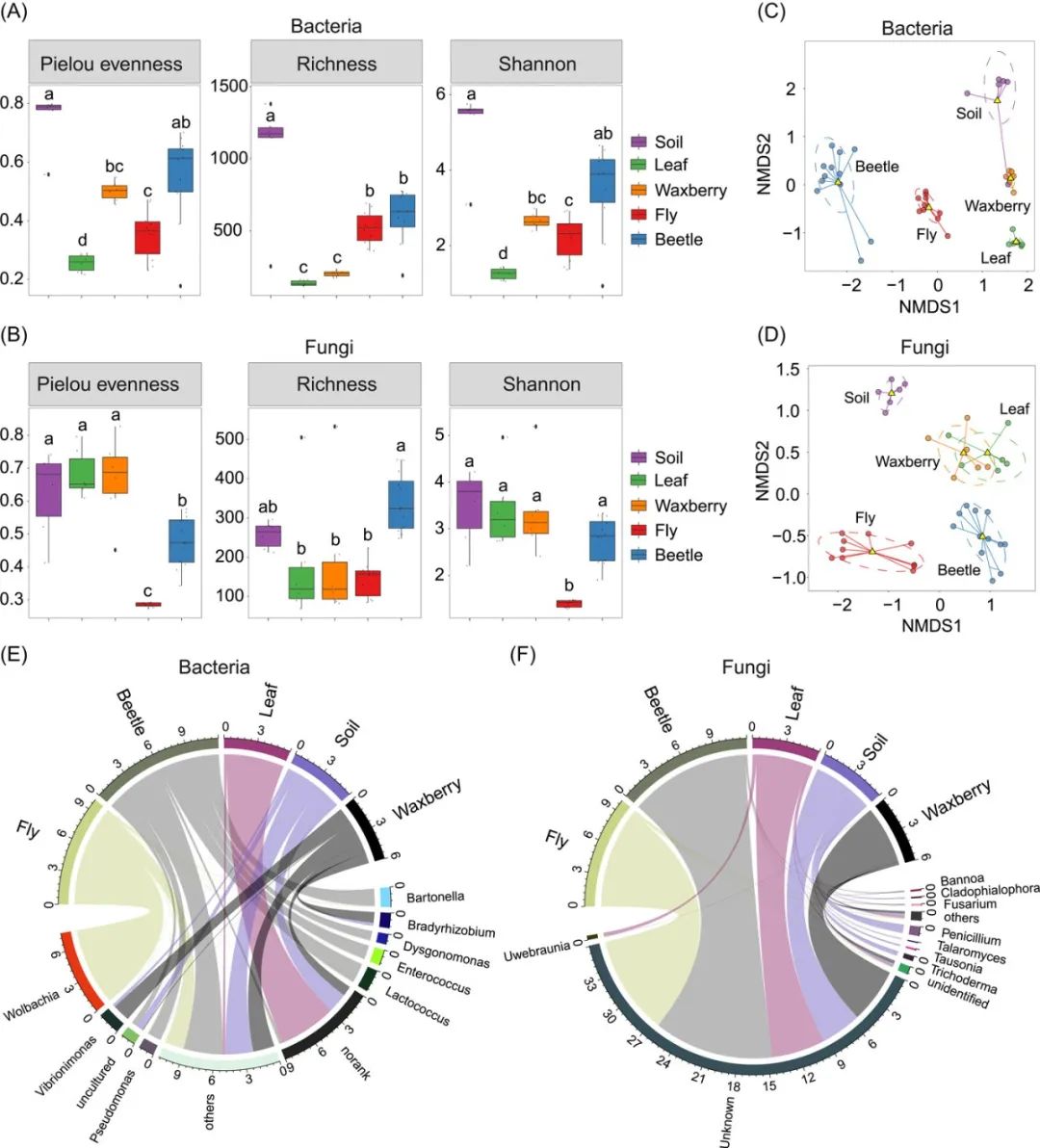
本教程相关代码已经上传至 https://github.com/iMetaScience/iMetaPlot/tree/main/230107FacetsBoxplot 如果你使用本代码,请引用:Yu-Xi Zhu. 2022. Gut microbiota composition in the sympatric and diet-sharing Drosophila simulans and Dicranocephalus wallichii bowringi shaped largely by community assembly processes rather than regional species pool. iMeta 1: e57. https://doi.org/10.1002/imt2.57 代码编写及注释:农心生信工作室 写在前面 箱线图 (boxplot) 是一种基于五位数摘要(“最小”,第一四分位数(Q1),中位数,第三四分位数(Q3)和“最大”)显示数据分布的标准化方法, 可以表示微生物群落的alpha多样性。本期我们挑选2022年10月13日刊登在iMeta上的Gut microbiota composition in the sympatric and diet-sharing Drosophila simulans and Dicranocephalus wallichii bowringi shaped largely by community assembly processes rather than regional species pool - iMeta | 扬州大学杜予州团队揭示同域内同食物的两种昆虫肠道微生物群落装配机制,选择文章的Figure 1A进行复现,基于vegan包和ggplot2包,讲解和探讨alpha多样性的计算、箱线图的可视化以及用ANOVA进行差异分析并用字母标注显著性,先上原图:
接下来,我们将通过详尽的代码逐步拆解原图,最终实现对原图的复现。 R包检测和安装 01 安装核心R包vegan、ggplot2以及一些功能辅助性R包,并载入所有R包。 # 下载包 if (!require("vegan")) install.packages('vegan') if (!require("ggplot2")) install.packages('ggplot2') if (!require("tidyverse")) install.packages('tidyverse') if (!require("agricolae")) install.packages('agricolae') # 加载包 library(vegan) library(ggplot2) library(tidyverse) library(agricolae)生成测试数据 02 由于缺少原始数据,因此本例使用vegan包自带的dune数据集进行测试。dune数据集的格式是otu表转置后的格式,包含了20个样品,每个样品有30个物种丰度,每一行是一个样品,每一列是一个物种。 # 载入dune数据集 data(dune) #载入dune包含分组信息等的元数据(即metadata),分组信息为Management列 data(dune.env)03 计算alpha多样性,包括均匀度、丰富度和香农指数。 #计算丰富度richness,即群落中丰度大于0的otu数量之和 richness 0) #计算香农指数Shannon diversity index,以e作为底数 shannon |
【本文地址】
今日新闻 |
推荐新闻 |