opencv实战 |
您所在的位置:网站首页 › opencv识别形状 › opencv实战 |
opencv实战
|
引言
在机器视觉中,有时需要对产品进行检测和计数。其难点无非是对于产品的图像分割。 由于之前网购的维生素片,有时候忘了今天有没有吃过,就想对瓶子里的药片计数...在学习opencv以后,希望实现对于维生素片分割计数算法。本次实战在基于形态学的基础上又衍生出基于距离变换的分水岭算法,使其实现的效果更具普遍性。 基于形态学的维生素片检测和计数 😀整体思路: 读取图片 形态学处理(在二值化前进行适度形态学处理,效果俱佳) 二值化 提取轮廓(进行药片分割) 获取轮廓索引,并筛选所需要的轮廓 画出轮廓,显示计数opencv实现: int main(int argc, char** argv) { Mat src, src_binary,dst,src_distance; src = imread("D:/opencv练习图片/维生素片机器视觉检测和计数.png"); imshow("原图片", src); Mat kernel = getStructuringElement(MORPH_RECT, Size(16, 16), Point(-1, -1)); morphologyEx(src, dst, MORPH_OPEN, kernel); imshow("形态学",dst); cvtColor(dst, dst, COLOR_RGB2GRAY); threshold(dst, src_binary, 100, 255, THRESH_OTSU); imshow("二值化", src_binary); vector contours; findContours(src_binary, contours, RETR_EXTERNAL, CHAIN_APPROX_NONE, Point(0, 0)); RNG rng(12345); double area; Point2i PL; for (size_t i = 0; i < contours.size(); i++) { area = contourArea(contours[i]); if (area < 500)continue; PL = contours[i].front(); Scalar color = Scalar(rng.uniform(0, 255), rng.uniform(0, 255), rng.uniform(0, 255)); drawContours(src, contours, i, color, 2, 8); putText(src, to_string(i), PL, FONT_HERSHEY_COMPLEX, 1, color, 2); } imshow("计数结果", src); waitKey(0); return 0; }效果展示:
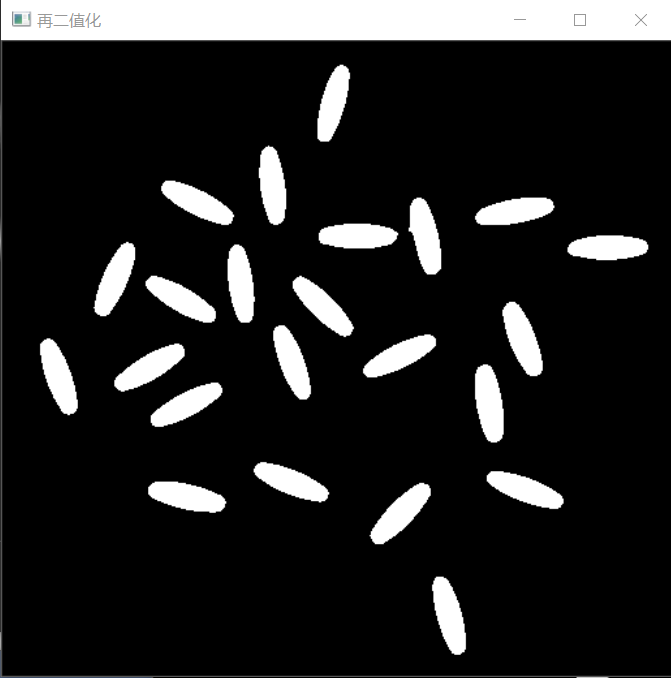
由上图可以看的,原图在经过形态学处理后,可以去除很多细节,简化后续的药片分割操作。 但是在计数结果图上发现,索引17号药片并没有完全分割(实际上修改形态学的结构元素尺寸(改为20*20)也可以完全分离这两个药片)。 🤨这不由得让我们思考,如果简单的形态学处理分割不了药片呢? 对于复杂的产品图片,我们可以使用基于距离变换的分水岭算法对其分割。 基于距离变换的分水岭算法检测和计数 OpenCV 采用了基于标记点的分水岭算法,在这种算法中我们要设置哪些山谷点会汇合,哪些不会。这是一种交互式的图像分割。我们要做的就是给我们已知的对象打上不同的标签(即添加注水点)。然后实施分水岭算法。每一次灌水,我们的标签就会被更新,当两个不同颜色的标签相遇时就构建堤坝,直到将所有山峰淹没,最后我们得到的边界对象(堤坝)的值为 -1。 🙁对于如何打上标签(即添加注水点)有两种办法: opencv中,对于一张二值化的图像,后续处理方式有两种。第一种方式就是利用findContours、drawContours等函数进行轮廓分析(opencv以对轮廓的处理为主)。第二种方式就是计算连通域进行区域分析。 第一种(基于轮廓):在二值化后,对图像寻找轮廓findContours,筛选出注水区域轮廓,然后通过drawContours对轮廓标记。 第二种(基于区域):在二值化后,先对寻找图像中的前景图(即注水点),再寻找到背景图(进行膨胀),最后找到未知区域(背景减去前景,得到边缘图),通过connectedComponents()获取标记点。 相关API: 分水岭函数watershed函数原型 void watershed( InputArray image, InputOutputArray markers );第一个输入参数 image,必须是CV_8UC3类型图像。 第二个输入/输出参数markers必须是32位单通道图像。和image尺寸一样。包含不同区域的轮廓,每个轮廓有一个自己唯一的编号。 😛在执行watershed函数后,算法会根据markers传入的轮廓作为种子,对图像上其他的像素点根据分水岭算法规则进行判断,并对每个像素点的区域归属进行划定,直到处理完图像上所有像素点。而区域与区域之间的分界处的值被置为“-1”,以做区分。 距离变换函数distanceTransform函数原型距离变换运算用于计算二值化图像中的每一个非零点距自己最近的零点的距离,距离变换图像上越亮的点,代表了这一点距离零点的距离越远。 距离变换通常用于求解图像的骨骼和查找物体的质心(即获取距离变换的极大值)和计算非零像素到最近零像素点的最短距离。 distanceTransform( InputArray src, OutputArray dst, int distanceType, int maskSize,int dstType = CV_32F);第一个输入参数src,必须是CV_8UC1类型的二值图像(只有0或1) 第二个输出参数dst,表示的是计算距离的输出图像,输出类型是CV_32F/CV_8U的单通道图像,大小与输入图片相同。 第三个参数distanceType,表示的是选取距离的类型,可以设置为DIST_L1,DIST_L2,DIST_C 第四个参数maskSize,表示的是距离变换的掩膜模板,可以设置为3,5(常用3) 第四个参数dstType,表示输出类型,可选择CV_32F/CV_8U 注:若输出类型为CV_32F,想要显示距离变换后的骨架图像,需要对其归一化。(normalize) 🧡💛💚💙💜🤎🖤 先来看看第一种标记mark(基于轮廓)的方法: (一)读入图像,形态学,二值化(消除噪声) Mat src, src_binary, dst, src_distance; src = imread("D:/opencv练习图片/维生素片机器视觉检测和计数.png"); imshow("原图片", src); Mat kernel = getStructuringElement(MORPH_RECT, Size(3, 3), Point(-1, -1)); morphologyEx(src, dst, MORPH_OPEN, kernel); imshow("形态学", dst); cvtColor(dst, dst, COLOR_RGB2GRAY); threshold(dst, src_binary, 100, 255, THRESH_OTSU); imshow("二值化", src_binary);  
(二)距离变换(归一化显示),再二值化 distanceTransform(src_binary, src_distance, DIST_L2, 3, 5); normalize(src_distance, src_distance, 0, 1, NORM_MINMAX); imshow("距离变换", src_distance); threshold(src_distance, src_distance, 0.4,1, THRESH_BINARY); imshow("再二值化", src_distance); 经过距离变换后的二值化,可以清晰看到,药片以及完全分割开来。 (三)打上标签(添加注水点),基于轮廓 //寻找标记点marsk的轮廓信息 也就是分水岭的水坝 src_distance.convertTo(src_distance, CV_8UC1); vector contours; findContours(src_distance, contours, RETR_TREE, CHAIN_APPROX_SIMPLE); //创建maker Mat markers = Mat::zeros(src.size(), CV_32S);// //因为分水岭后的边缘存储是-1,所以必须使用有符号的CV_32S for (size_t t = 0; t < contours.size(); t++) { drawContours(markers, contours, static_cast(t), Scalar(static_cast(t) + 1), -1);//轮廓数字编号 } circle(markers, Point(5, 5), 30, Scalar(255), -1);//关键代码(mark做一个小标记) int index1 = 0; //打印轮廓数据 有值的均为轮廓线 for (int row = 0; row < markers.rows; row++) for (int col = 0; col < markers.cols; col++) { index1 = markers.at(row, col); cout 0 && index |
【本文地址】
今日新闻 |
推荐新闻 |