Mac/Win's VScode+Linux服务器+docker/podman容器模式配置生物信息分析工作流 |
您所在的位置:网站首页 › mac与linux命令区别 › Mac/Win's VScode+Linux服务器+docker/podman容器模式配置生物信息分析工作流 |
Mac/Win's VScode+Linux服务器+docker/podman容器模式配置生物信息分析工作流
|
具体实现的内容可以直接跳到「具体实现 Mac --ssh--> container in local server」阅读,但前文还是很重要的,最好从头到尾阅读。为什么我们要用这样的容器模式建立这样的生信工作流 常见的生信工作模式:Mac/Win + conda environment in Linux 服务器(Centos or Ubuntu) 这样的工作模式有一个弊端,例如 16s 分析我们需要在服务器中用 conda/mamba env create -n qiime2 --file qiime2-version-pyversion-linux-conda.yml来创建一个 qiime2.yml 配置的集合的环境。「其中 mamba 是优化版的 conda。」这样做的问题在于如果需要一些定制开发,将会有其他的包被安装进来,有可能会由于工具的依赖冲突污染环境,工具版本冲突就是一个很好的例子,若工具之间耦合性很高,那么将会有很长时间在这里进行配置,也可以切换一种方案:再conda env create一个环境,在 pipeline 中切换 conda 环境。我们可以本地创建好一个 shell 流程或者 nextflow 流程,然后把流程代码 scp 进服务器中的工作目录跑上游分析流程。或者可以直接用远程 shell 工具做一个 ssh 连接来编辑服务器中的流程代码内容。 有一个更好的方案,就是把容器当作 pipeline 运行环境,而不需要使用 Linux 服务器下的虚拟环境。 容器工具除了我们熟知的 Docker,还有 RedHat 公司发行的 Podman。底层原理稍有区别,这个大家可以自己研究,粗略点的说可以把所有 docker 命令中首个单词 docker 直接用 podman 替换,命令几乎一摸一样。所以本文的容器就用 podman。docker 用户只需要换换单词就行了。 我们使用本地的 VScode 编辑器做代码编写。所以无论是 Mac 用户还是 Win 用户,其实都一样。由于本人是 Mac 用户所以我们就用 Mac 来介绍这样的工作模式的配置方法。 所以现在我们可以把这样的工作模式的配置简单的说成是 VScode+Linux服务器+podman容器。 由于本人刚辞职,还没有服务器来举例所以本文的实现方案是 VScode+本地Linux虚拟机+podman容器。这个和本文要介绍的「VScode+Linux服务器+podman容器」这种工作模式的区别就只有 IP 地址需要改动。会在后面指出。读者若没有服务器,也可以直接用本文来动手实现。若有服务器,在本文的后续内容将会指出需要改动的地方。 以上说的本地 Linux 虚拟机很多人认为是运行在 VMware for Windows 上的 Linux,又或者是 Parallel for MacOS 上的 Linux。——这样可以,但没必要。 因为 Podman in Linux 和 Podman in Mac/Win 是有区别的。这是由于 podman 的容器管理是基于 linux 运行环境的。而 mac/win 是没有这个 linux 环境的。 Linux 服务器中的 Podman: # podman in Ubuntu apt install -y podman # podman in Centos yum install -y podman当我们的 Linux 服务器中有了 podman 之后,我们现在来看一下我们构建一个镜像: podman build -t ubuntu_bioInfo: -f Dockerfile_ubuntu /path/to/files_that_need_to_add_in_image其中 Dockerfile_ubuntu 是你自己编写的 Dockerfile,用于生成你配置好的生信分析工具集的基础镜像。 这时候我们可以直接使用命令podman images来查看镜像列表。 Mac/Win 中的 Podman: # podman in Mac brew install podman # podman in Win scoop install podman当我们的 Mac/Win 中有了 podman 之后,我们并不能直接构建镜像,如果你要尝试运行这行命令: podman build -t ubuntu_bioInfo: -f Dockerfile_ubuntu /path/to/files_that_need_to_add_in_image或者podman pull [image_in_dockerhub]都会得到这样的报错: Error: failed to connect: dial tcp [::1]:53363: connect: connection refused原因是你需要运行 podman machine init来初始化你的 podman 运行环境,在这个初始化的过程中,其实 podman 构建了一个最简 linux 虚拟机,用于管理 podman 的镜像容器数据卷等。所以说 podman 的容器技术的底层实现都是需要运行在 linux 系统上的,所以需要 linux 作为底盘。而刚好 Mac/Win 无法作为底盘,所以需要这个初始化过程创建一个运行在本机上的 linux 虚拟机服务于容器管理技术,仅仅用作容器管理所以不需要很庞大完整的 Linux 系统,也就不需要用到 VMware for windows 或 Parallel for Mac 来创建 Linux 系统。 这时候我们我们还是不可以构建镜像,我们还需要 podman machine start来开启你的 podman 服务。相当于启动了这个用作 podman 镜像容器数据卷管理的 Linux 系统。这个系统可以使用命令podman machine ssh连进去。开启服务之后你就可以开始构建镜像了: podman build -t ubuntu_bioInfo: -f Dockerfile_ubuntu /path/to/files_that_need_to_add_in_image顺便一提,你可以podman machine stop来停止你的 podman 服务。这时候你已经可以执行podman build/create/run/start/stop/exec/commit/save/load/pull/push/ps/ls/top等命令了。 以上可以看到 Podman in Linux 和 Podman in Mac/Win 的区别。Mac/Win 中使用 podman 会多了podman machine init以及podman machine start的动作。 (值得注意的是podman machine init只需要运行一次,再次使用只需要 start 就行了,podman machine 可以在podman machine list中查看到。)所以我们可以用这样的逻辑看待问题: podman in Mac/Win:local --> podman machine --> containerpodman in Linux (Centos/Ubuntu):local --> containerLinux 服务器本身就是一个 podman machine从现在开始,将不会再写 Mac/Win 来描述用户本地操作系统,就写 Mac,也不会再写 Centos/Ubuntu 来描述 Linux 的所属系列,就写 Ubuntu。如果你是 Win 用户,也不用担心,一点问题没有。 在这里展示一下 Mac 中的 podman machine:  podman machine is a Fedora System podman machine is a Fedora System这是一个 Fedora 系统,是一个基于 Linux 的系统。 接下来将会介绍 「VScode for Mac」 + 「本地 Linux 虚拟机(podman machine) 」+ 「Ubuntu 容器」这样的工作流模式。 如果你想要的是标题所说的「VScode for Mac」 + 「远程 Linux 服务器(podman machine) 」+ 「Ubuntu 容器」,那仅仅是在路由设定中内网 IP 与公网 IP 的区别。本文改一个参数同样可以实现,后文中在相应的内容会标出。Mac's VScode+本地 Linux 虚拟机(Fedora)+Container(Ubuntu)生信分析工作,我们会有上下游分析,尤其是上游分析,这是肯定会在服务器上运行的。我们准备的 pipeline 代码都会存放在服务器上的容器里运行,这里给出的方案是 VScode 用 ssh 远程连接服务器。还有一个问题是,我们的生信数据(fastq/fasta 文件等等)都会存放在远程服务器上,通常会把存放生物测序的下机数据的硬盘挂载到服务器上的 /mnt/biodata/ 目录。我们需要做的就是再把远程服务器上的 /mnt/biodata/ 在挂载到容器中的 /mnt/biodata/ 中去。 现在我们来看两张图: Mac --ssh--> container in remote server  Mac ssh container in remote Linux server Mac ssh container in remote Linux serverMac --ssh--> container in local server  Mac ssh container in local Fedora server Mac ssh container in local Fedora server我们要实现的是第二张图的内容,其中第二张图 ssh 的 ip 地址其实是本地的内网 IP。这是为了形象化表示两者的理论区别而展示的两张图。实际上第二张图的实际样子是:(container in Fedora) in MacOS。(可以想象一下蓝色矩形在内部,绿色矩形在中部,红色矩形在外部的情形。) 以上两图分别是连接远程服务器模式和连接本地服务器模式: 一个是从远程服务器中的 /mnt/biodata/ 挂载到远程服务器中的 podman 容器中的 /mnt/biodata/ 目录。还有一个则是从本地 Mac 下的目录 ~/biodata/ 挂载到本地服务器中的 podman 容器中的 /mnt/biodata/ 目录。假设 Mac 中已经有了 podman 镜像,镜像名称为 ubuntu_biology:v1.0.0。在 Mac 中运行以下命令: podman run --name ubuntu_bioInfo -itd -v ~/biodata:/mnt/biodata -p 2222:22 ubuntu_biology:v1.0.0ubuntu_bioInfo —— 要被创建的容器的名称-v ~/biodata:/mnt/biodata —— 把本地的 ~/biodata/ 目录挂载到容器中的 /mnt/biodata/ 目录(~/bio/biodata 是 Mac 下的目录,不是 Fedore 下的)-p 2222:22 —— 把本地的 2222 端口映射到容器中的 22 端口再假设远程服务器 Linux server 中已经有了 podman 镜像,镜像名称为 ubuntu_biology:v1.0.0。在远程服务器 Linux 中运行以下命令: podman run --name ubuntu_bioInfo -itd -v /mnt/biodata:/mnt/biodata -p 2222:22 ubuntu_biology:v1.0.0ubuntu_bioInfo —— 要被创建的容器的名称-v /mnt/biodata:/mnt/biodata —— 把远程服务器下的 /mnt/biodata/ 目录挂载到容器中的 /mnt/biodata/ 目录(/mnt/biodata 是远程服务器下的目录)-p 2222:22 —— 把远程服务器自生的 2222 端口映射到容器中的 22 端口可以看出重点是 -v 参数的区别。-v 是目录挂载参数,-p 是端口映射参数。目录挂载的区别细节这里已经描述清楚,由于本人现在没有远程服务器,下文就实现 Mac --ssh--> container in local server。如果你想实现 Mac --ssh--> container in remote server。除了留意上文中说到的目录挂载区别,也请留意一下下文中的「网关 IP」。 具体实现 Mac --ssh--> container in local server# 安装 podman, Win 用户把 brew 换成 scoop 或 chocolate brew install podman # 创建 podman machine podman machine init # 启动 podman machine podman machine start # 创建镜像 podman load < /path/to/ubuntu_bioInfo.tar其中创建镜像有三种方式: load 已有的 tar 镜像包来提取镜像pull Dockerhub 上的基础包来获得镜像build 通过自己配置的 Dockerfile 镜像生成文件来构建镜像这里是由于有了自己制作的 tar 镜像包,所以直接用 load 构建了镜像,其中镜像名为 ubuntu_biology:v1.0.0,冒号右边是版本号。如果你没有自己的 tar 包也没有自己的 Dockerfile 镜像生成器文件,那你可以暂时直接用podman pull ubuntu:22.04来拉取一个官方的基础镜像包。之后有了镜像之后可以podman images查看自己的镜像列表。 现在我们已经有了镜像 ubuntu_biology:v1.0.0,我们接着来podman run创建并运行一个容器(其中podman run=podman create+podman start如果你本地没有 run 中指定的镜像,那么podman run=podman pull+podman create+podman start) podman run --name ubuntu_bioInfo -itd -v ~/biodata:/mnt/biodata -p 2222:22 ubuntu_biology:v1.0.0这个命令做了两个动作(创建并运行容器)可以用podman ps来查看容器列表。假如你podman stop ubuntu_bioInfo了,则可以用podman ps -a来查看到这个容器,也可以用podman start ubuntu_bioInfo来再次启动容器。 Fedora 的默认登陆用户是 core,容器的默认登陆用户是 root。 现在我们需要获取三样东西,分别是: Fedora 的子网掩码Fedora 的内网 IPv4 地址ubuntu_bioInfo 容器的子网 IPv4 地址# 从 Mac 中 ssh 进 Fedora podman machine ssh # 在 Fedora 中查看虚拟网卡配置信息 ifconfig # 查看完之后 exit 退出 Fedora 回到 Mac 中 exit ifconfig in Fedora# 从 Mac 中 exec 进 ubuntu_bioInfo 容器(我在镜像包中装了 zsh, 没有 zsh 的把最后一项改成 /bin/bash)
podman exec -it ubuntu_bioInfo /bin/zsh
# 在 ubuntu_bioInfo 中查看虚拟网卡配置信息
ifconfig
# 查看完之后 exit 退出 ubuntu_bioInfo 回到 Mac 中
exit ifconfig in Fedora# 从 Mac 中 exec 进 ubuntu_bioInfo 容器(我在镜像包中装了 zsh, 没有 zsh 的把最后一项改成 /bin/bash)
podman exec -it ubuntu_bioInfo /bin/zsh
# 在 ubuntu_bioInfo 中查看虚拟网卡配置信息
ifconfig
# 查看完之后 exit 退出 ubuntu_bioInfo 回到 Mac 中
exit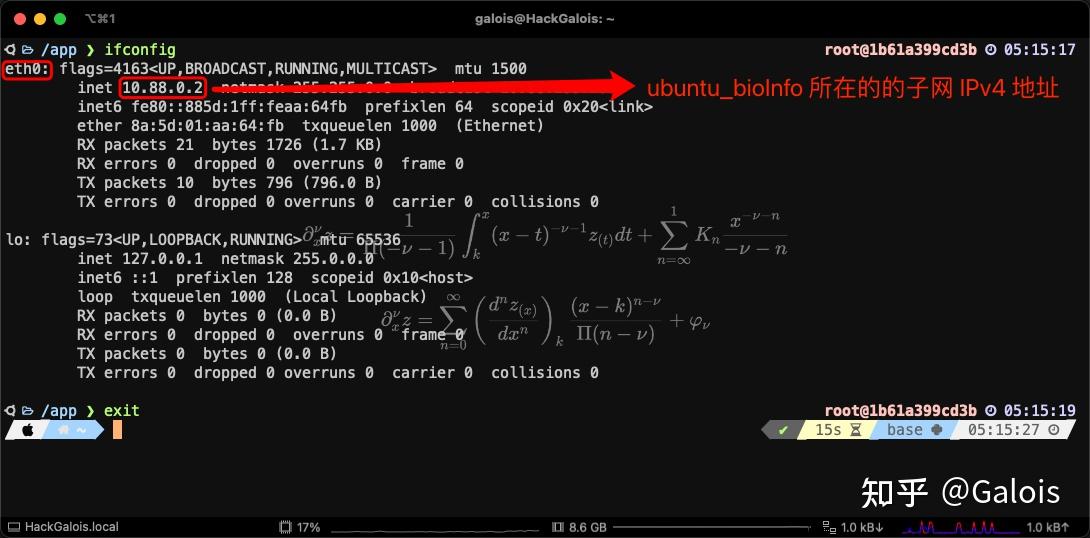 ifconfig in ubuntu_bioInfo container ifconfig in ubuntu_bioInfo container现在需要在容器中开启 ssh 服务: # 从 Mac 中 exec 进 ubuntu_bioInfo 容器 podman exec -it ubuntu_bioInfo /bin/zsh # 安装 ssh 服务工具包, Centos 容器的把 apt 改成 yum apt install -y openssh-server # 设置容器的 root 密码 passwd root # vim /etc/ssh/sshd_config 并把 PermitRootLogin 项的注释解开赋值 yes echo "PermitRootLogin yes" >> /etc/ssh/sshd_config # 开启 ssh 服务 service ssh start因为 Fedora 和 ubuntu_bioInfo 采用的是桥接模式,所以已经可以直接在 podman machine 中 ssh 访问进 ubuntu_bioInfo 容器了。 # 从 Mac 中 ssh 进 podman machine podman machine ssh # 从 podman machine 中 ssh 进容器 ssh [email protected] -p 2222 ssh [email protected] -p 22从 Mac 中 ssh 进 ubuntu_bioInfo 容器。 # 在 Mac 中设置路由 # route add -net [容器的网络地址] -netmask [Fedora 子网掩码] [Fedora IPv4 地址(用作网关地址)] sudo route add -net 10.88.0.0 -netmask 255.255.255.0 192.168.127.1 # 路由列表可以在 netstat -nr 中查看, 删除路由可以执行 route delete 10.88.* # 从 Mac 中 ssh 进 ubuntu_bioInfo 容器 ssh root@localhost -p 2222 ssh [email protected] -p 2222从 VScode 中 ssh 进容器,就是顺理成章的事情了,直接下载插件 remote-ssh 然后用ssh root@localhost -p 2222就能进入到容器里,之后选择目录打开就行。 具体实现 Mac --ssh--> container in remote server其实看了上面一节就已经可以知道本地 ssh 连接进服务器的实现方法了,我们在设置路由的时候把网关地址改成远程服务器的公网 IPv4 地址就行。 把上一节的podman machine ssh改成ssh yourusername@远程服务器公网IP -p 22 把上一节的 route add -net [容器的网络地址] -netmask [Fedora 子网掩码] [Fedora IPv4 地址(用作网关地址)]改成 route add -net [容器的网络地址] -netmask [远程服务器的子网掩码] [远程服务器的公网 IPv4 地址(用作网关地址)]最后从本地 ssh 到远程服务器中的容器中,使用以下命令: ssh root@远程服务器公网IP地址 -p 2222需要注意的是这里的 root 不是远程服务器的 root 用户,而是运行在远程服务器上的容器的 root 用户,后面的 2222 端口指的是服务器的 2222 端口,它会映射进容器的 22 端口。 如果你有 Rnaseq 这样的数据体量比较小的上游分析任务,就可以使用 VScode for Mac/Win --ssh--> container in local server 这样的工作模式。 我们的工作模式将会变成: 本地的 fastq 数据文件所在目录挂载到容器,实现容器内的 shell 读取 fq 数据使用 VScode 编辑器 ssh 连接到容器内部的项目目录实现隔空代码编写所有的 nextflow 脚本、shell 脚本、R 脚本、Python 脚本、conda/mamba 环境等都会在容器中方便管理可以在容器中用 git 工作流开发模式如果你在做一些数据体量大的分析任务,就可以使用 VScode for Mac/Win --ssh--> container in remote server 这样的工作模式。工作模式将会变成: 远程服务器的 fastq 数据文件所在目录挂载到容器,实现容器内的 shell 读取 fq 数据使用 VScode 编辑器 ssh 远程连接到容器内部的项目目录实现隔空代码编写所有的 nextflow 脚本、shell 脚本、R 脚本、Python 脚本、conda/mamba、jdk 环境等都会在容器中方便管理可以在容器中用 git 工作流开发模式Actually,不管你做生信分析还是 AI 算法模型都可以用到这个模式,只不过镜像内会预装 Pytorch、Tensorflow 等工具集。 顺带一提 VScode 中的 SSH-Container 插件很好用,码字不易,转载请注明出处。 |
【本文地址】
今日新闻 |
推荐新闻 |