如何分析m6A环状RNA?这里有提炼好的策略! |
您所在的位置:网站首页 › m6a甲基化修饰 › 如何分析m6A环状RNA?这里有提炼好的策略! |
如何分析m6A环状RNA?这里有提炼好的策略!
|
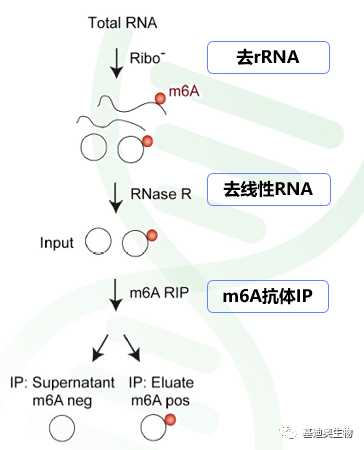
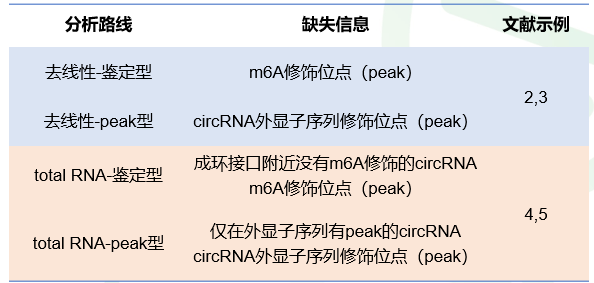
图1 去线性路线的m6A circRNA实验策略 1.2 total RNA路线 这种路线在实验步骤上,和去rRNA方式(相对的是polyA方式)的m6A-seq测序基本相似。主要是在分析层面加入了m6A circRNA。既然实验简单,当然在分析上会受限制,通常作为total RNA路线分析的补充拓展,或者是m6A circRNA方向的初步试探。 具体分析的时候,文献中有两类策略: 一种是基于IP数据直接鉴定circRNA,因为circRNA的鉴定,是基于成环接口处的reads,若circRNA成环接口附近有m6A 修饰,则会被IP获取,从而鉴定出来。 一种是circRNA call peak后,去除和外显子序列交集的peak,剩余的peak则作为m6A circRNA的peak。即避开peak来源模糊的部分,不予分析。 可见,该路线的两种方法都有未知程度的m6A circRNA损失,也有部分文章使用。 我们总结如下,方便大家取舍参考。 表1 m6A circRNA研究思路对比
Part2 m6A circRNA数据挖掘方案 明白了m6A circRNA鉴定的思路,接下来就是数据挖掘啦。虽然有不同路线,但最终在数据分析时,都可以从有无m6A circRNA、m6A circRNA甲基化富集倍数两份基础数据开展分析。 另: 差异对比会有两个方向,①有无m6A对比;②分组间对比。 2.1 有无m6A对比 总的来说,如图2,鉴定出了两类circRNA后,可以从circRNA数量、circRNA类型、外显子数量、长度分布、表达量分布等角度,进行所有样本或分组间比较。可以初步了解m6A修饰的circRNA潜在的各类型特征。
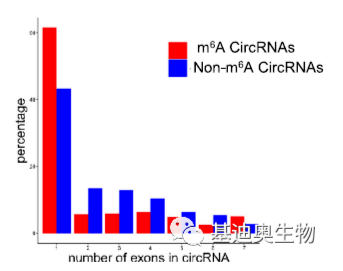
图2 有无m6A circRNA对比 以图3为例,是对外显子数量的统计,横轴表示circRNA含外显子的数量,纵轴表示对应circRNA占比;颜色表示有无m6A。由图可知,对于含一个外显子的circRNA,存在m6A修饰的比例更高。
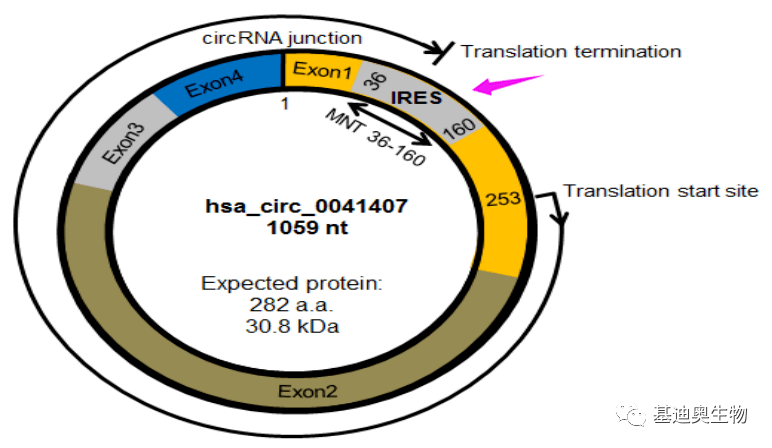
图3 有无m6A circRNA外显子数量占比 比较特别的,还可以从IRES角度对比。 真核细胞中,RNA的翻译起始可以分为两大类: 1)5’帽子依赖型翻译起始(5’ cap dependent),即通过mRNA 5’端的帽子结构,招募核糖体,起始翻译; 2)5’帽子非依赖型翻译起始(5’ cap independent),比如通过m6A、IRES(internal ribosome entry site,内部核糖体进入位点)等招募核糖体,启动翻译。其中,IRES是一段核酸序列,它的存在能够使RNA不依赖5’帽子而直接与核糖体结合,起始RNA的翻译。 circRNA没有mRNA的5’帽子结构,但可能通过IRES或m6A发挥翻译功能(图4[6]),进一步开展IRES分析,有利于评估预测circRNA的潜在翻译价值。
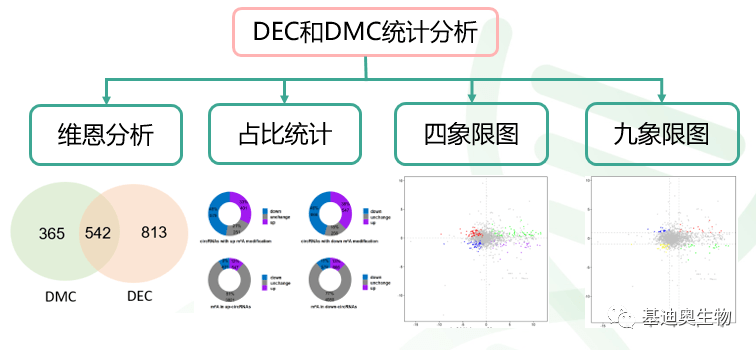
图4 circRNA IRES序列示例 2.2 甲基化与表达量关联分析 基于circRNA的m6A甲基化富集倍数和表达量,我们可以分别鉴定出差异表达的circRNA(DEC,Differential Expression circRNA)和差异甲基化的circRNA(DMC,Differential Methylation circRNAs)。获得DEC、DMC信息后,即可开展维恩、占比、四九象限图的多角度分析(图5),详细讨论circRNA甲基化差异与表达量差异的趋势变化,潜在关联。
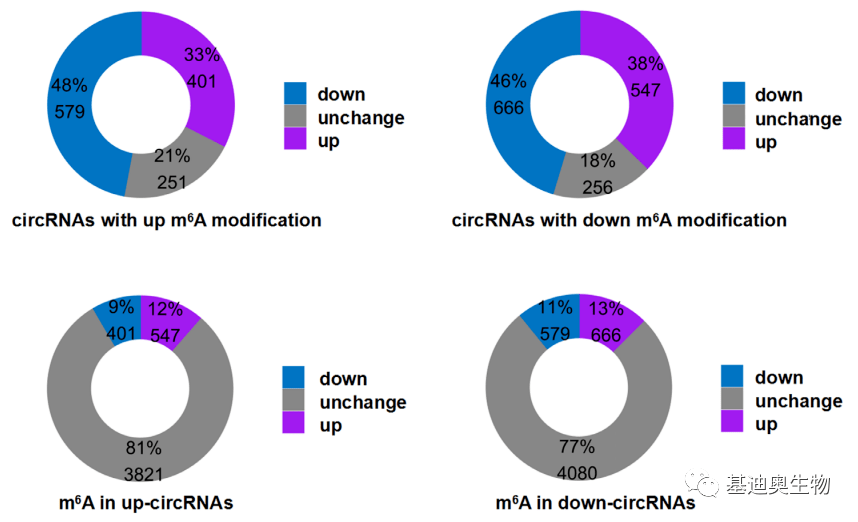
图5 DEC和DMC关联分析 如图6,第一行为DMC中,表达量上调、下调、不变的circRNA比例;第二行为DEC中,甲基化上调、下调、不变的circRNA占比。左边为对应的上调统计,右边为对应的下调统计。由图第一行可知,发生m6A修饰的circRNA与表达量有较强的关联,占DMC的80%左右;剩余有20%左右circRNA表达量没有显著差异,暗示m6A有可能参与了其他过程的影响,比如circRNA的翻译。由图第二行,DEC中,仅有20%左右的circRNA有甲基化差异,暗示众多差异表达的circRNA中,还有许多m6A以外的调控机制。
图6 DEC和DMC占比统计 除了以上两类分析,基迪奥还可以开展甲基化富集倍数与表达量的相关性分析、DMC参与的CeRNA网络等。欢迎有m6A circRNA兴趣的小伙伴咨询当地销售。 ▼ 参考文献 ▼ [1] Multifaceted regulation of translation by the epitranomic modification N6-methyladenosine. Critical Reviews in Biochemistry and Molecular Biology. 2021. [2] Genome-wide maps of m6A circRNAs identify widespread and cell-type-specific methylation patterns that are distinct from mRNAs. Cell Rep. 2017. [3] m6A-dependent biogenesis of circular RNAs in male germ cells. Cell Research. 2020. [4] Landscape of N6-Methyladenosine Modification Patterns in Human Ameloblastoma. Front. Oncol. 2020. [5] Identification and Characterization of N6-Methyladenosine CircRNAs and Methyltransferases in the Lens Epithelium Cells From Age-Related Cataract. Invest Ophthalmol Vis Sci. 2020. [6] CircInteractome: A web tool for exploring circular RNAs and their interacting proteins and microRNAs. RNA Biology. 2020. 实用工具操作教程分享 前沿组学文章实时解读 实验技术分析方法探讨 独家生信视频教程推荐 基迪奥 | 专业定制测序服务返回搜狐,查看更多 |
【本文地址】
今日新闻 |
推荐新闻 |