GEO数据挖掘 |
您所在的位置:网站首页 › dna数据怎么对比分析 › GEO数据挖掘 |
GEO数据挖掘
|
From生物技能树 GEO数据挖掘第一节 (pipeline) 文章目录 1.图表分析2.GEO背景介绍及分析思路3.代码分析流程4.复杂数据分析理论知识1.数据从哪里来2.有什么类型的数据可挖掘3.如何筛选基因(分析方法)在这里插入图片描述 图表介绍1.热图2.散点图和箱线图3.火山图4.主成分分析图 如何查找数据,GEO数据库1.表达数据实验设计2.数据库介绍 分析思路需要整理的信息芯片差异分析所需的输入数据 代码分析流程(在pipeline文件夹)安装 R 包下载数据,提取表达矩阵和临床信息提取表达矩阵exp检查表达矩阵是否异常提取临床信息提取芯片平台编号,后面要根据它来找探针注释 Group(实验分组)和ids(探针注释)实验分组的设置 需要把Group转换成因子,因子非常适合组织有重复值的向量,并设置参考水平,指定levels,对照组在前,处理组在后探针注释的获取运行 find_anno(gpl_number) 代码会出现的报错:解决办法:查看ids,要求ids只能有两列,第一列探针id,第二列symbol,并且列名都要是小写的列名 1.图表分析 2.GEO背景介绍及分析思路 3.代码分析流程 4.复杂数据分析提示:这是GEO数据挖掘的大概内容: 理论知识广义的基因有 6w+个,如何缩小范围到课题相关? 1.数据从哪里来
基因表达芯片 转录组 单细胞 突变(外显子)、甲基化、拷贝数差异。。。 3.如何筛选基因(分析方法)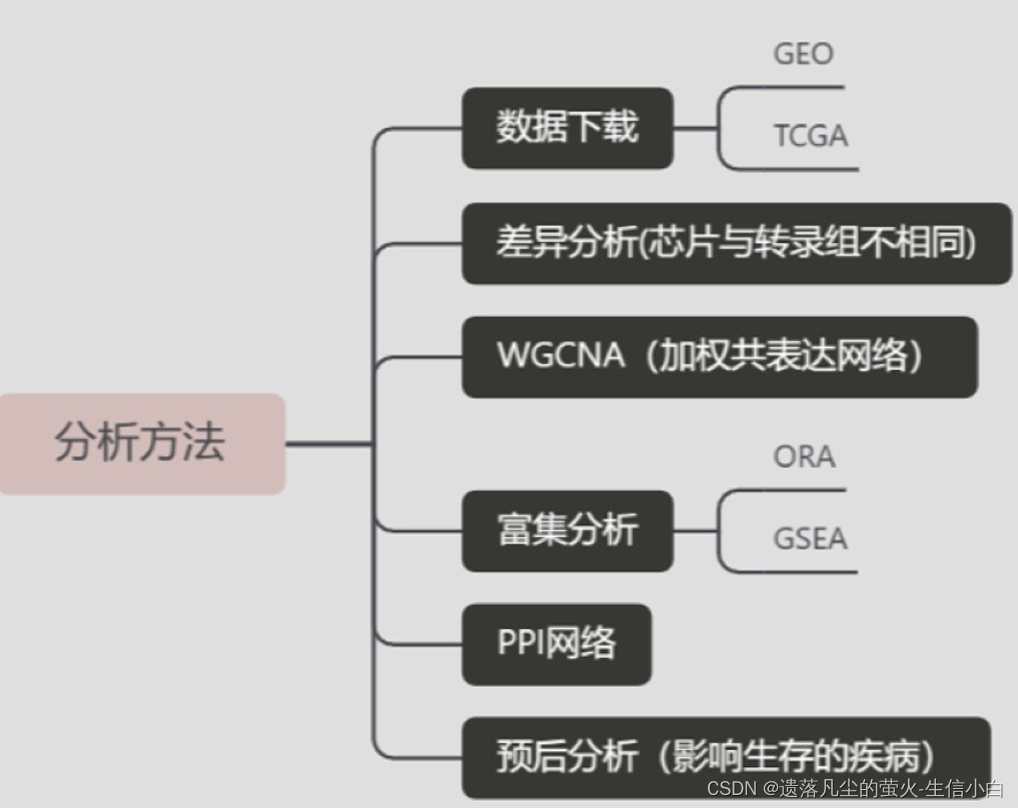 图表介绍
1.热图
输入数据是数值型矩阵/数据框颜色的变化表示数值的大小
2.散点图和箱线图
横坐标是下标/组纵坐标是表达值箱线图的五条线:max、75%、median、25%、min箱线图:用于单个基因在两组之间的表达差异 输入数据是一个连续型向量和一个有重复值的离散型向量
图表介绍
1.热图
输入数据是数值型矩阵/数据框颜色的变化表示数值的大小
2.散点图和箱线图
横坐标是下标/组纵坐标是表达值箱线图的五条线:max、75%、median、25%、min箱线图:用于单个基因在两组之间的表达差异 输入数据是一个连续型向量和一个有重复值的离散型向量 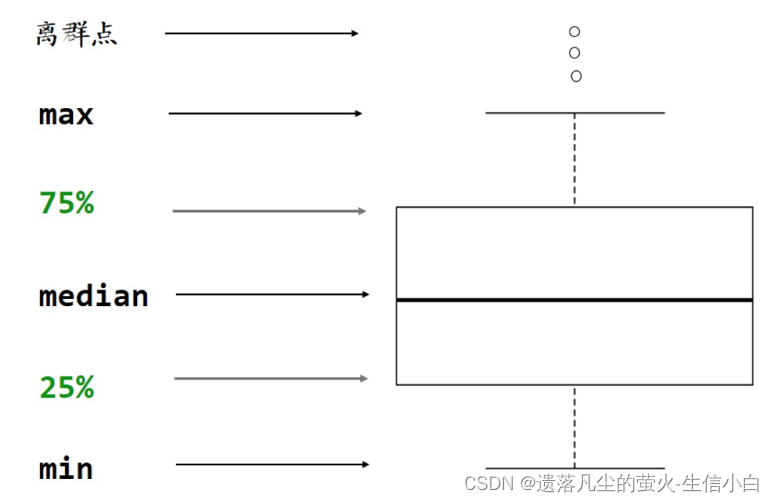 3.火山图
3.火山图
Foldchange(FC):处理组平均值/对照组平均值 log2Foldchange(logFc):Foldchange取log2 List item火山图:差异分析,用于多个基因在两组间的表达差异横坐标是 logFC,纵坐标是-log10(Pvalue)logFC: Foldchange取log2,Foldchange为处理组表达量平均值/对照组表达量平均值,实际计算时,为先取log2,然后log2(处理组)-log2(对照组),既没有单位也没有意义,只是一个相对值的意义永远都是处理组-对照组,而不是大的-小的logFC 如果太大:是不是没取 log?顶多在 ±12 之间logFC > 0,treat > control,基因表达量上升logFC < 0,treat < control,基因表达量下降通常所说的上调、下调基因是指表达量显著上升/下降的基因(需要结合显著性p值)logFC 的阈值设置是自行规定的,p 值阈值一般为 0.01,常见的 logFC 阈值有:1、2、1.2、1.5、2.2、(0.585=log2(1.5):即上升1.5倍),阈值可调。为什么 y 轴要取 lg?把 p 值拉到同一个数量级上,比较好画图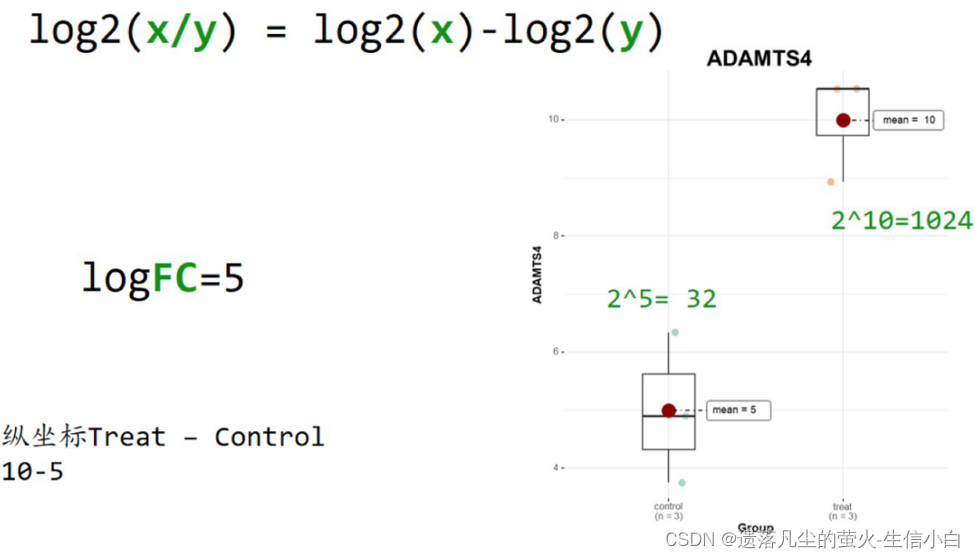 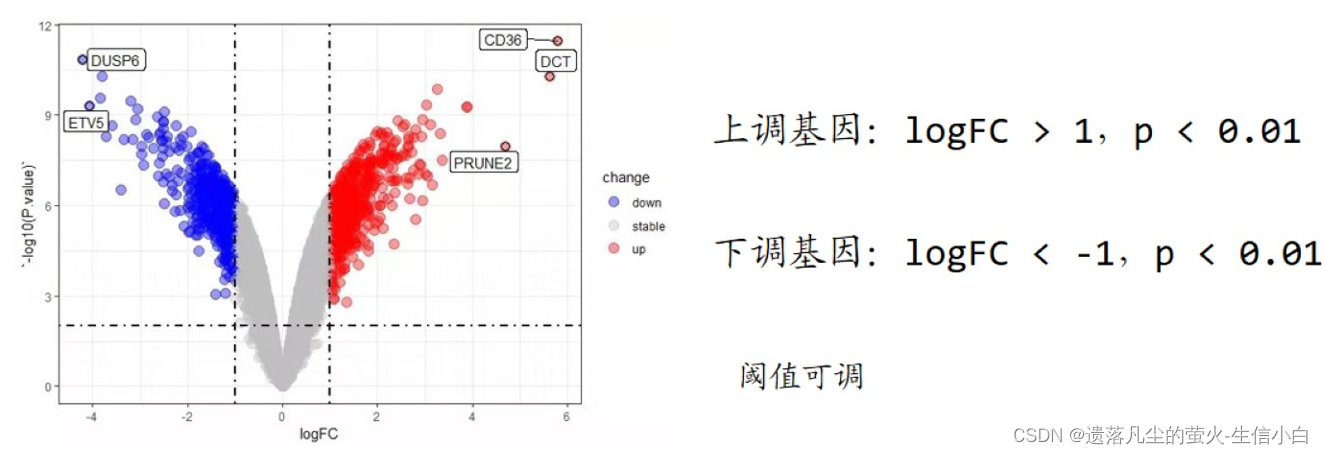 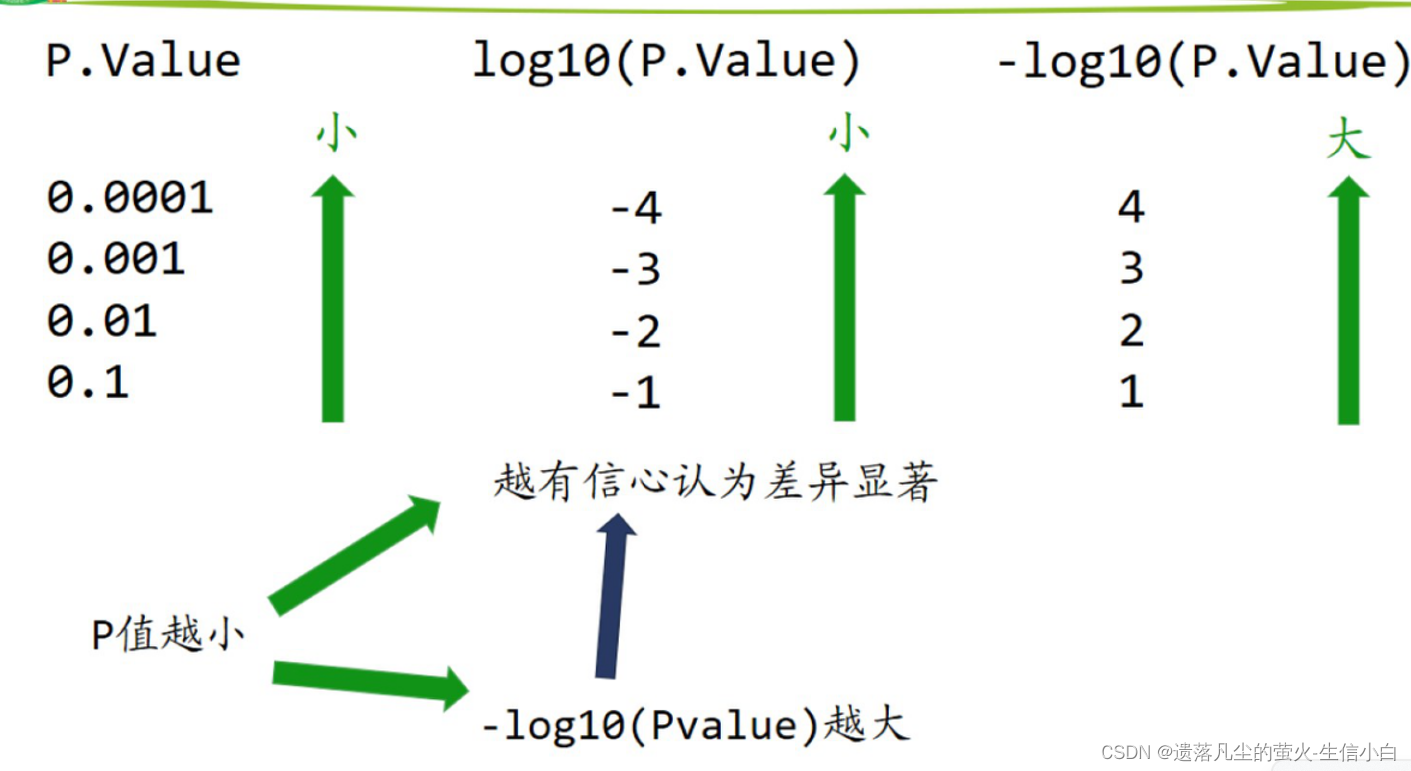
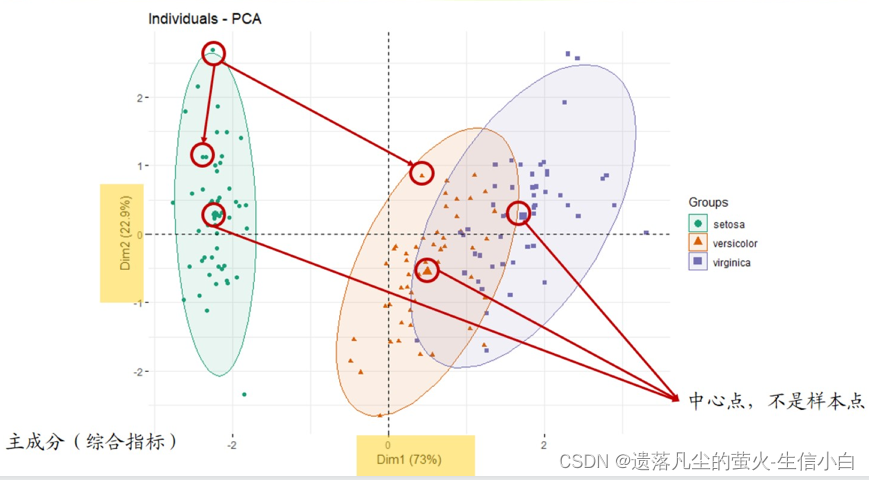
芯片差异分析的起点是一个取过log的表达矩阵(0-20), 如果拿到的是未log的矩阵(0-很大),需要自行log 4.主成分分析图1.主成分分析,旨在利用降维的思想,把多指标转化为少数几个综合指标(即主成分) 根据这些主成分对样本进行聚类,代表样本的点在坐标轴上距离越远,说明样本差异越大, 2. 图上的点代表样本(除中心点外),点与点之间的距离表示样本与样本的区别 3. 主成分的值没有意义,是很多个变量降维得到的一个值,dm1和dm2是维度,dm1+dm2>90%, 4. 用处:用于“预实验”简单查看组间是否有差别。同一分组是否聚成一簇(组内 重复好),中心点之间是否有距离(组间 差别大)。
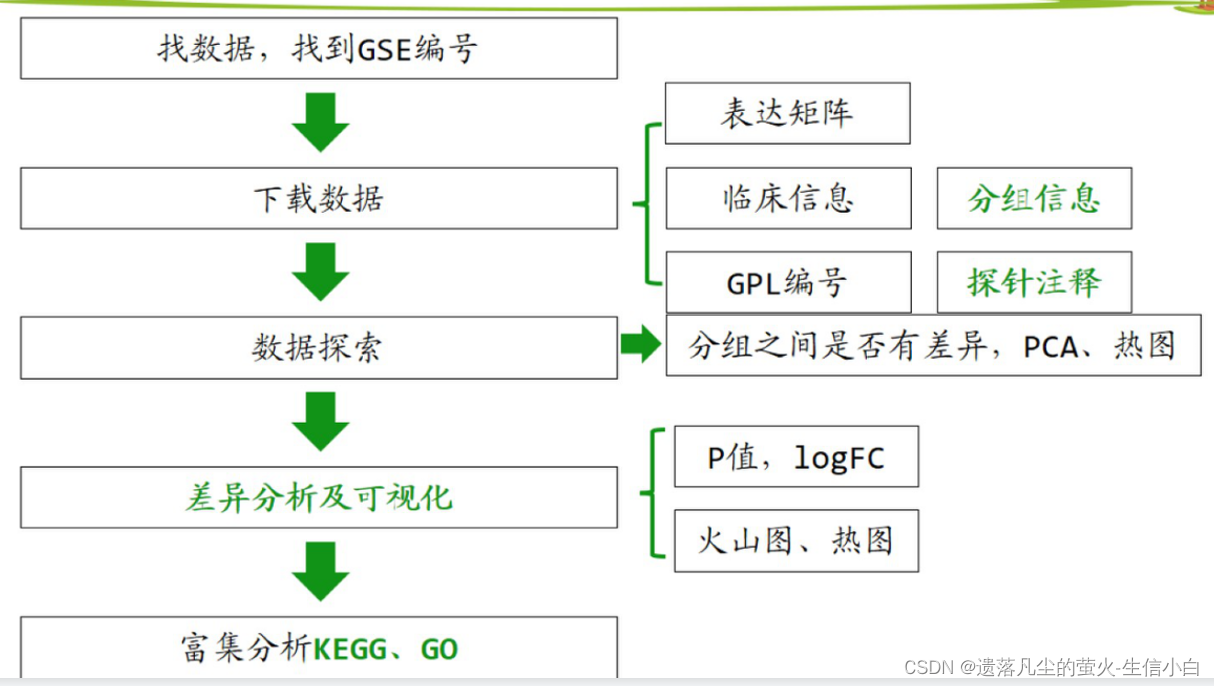
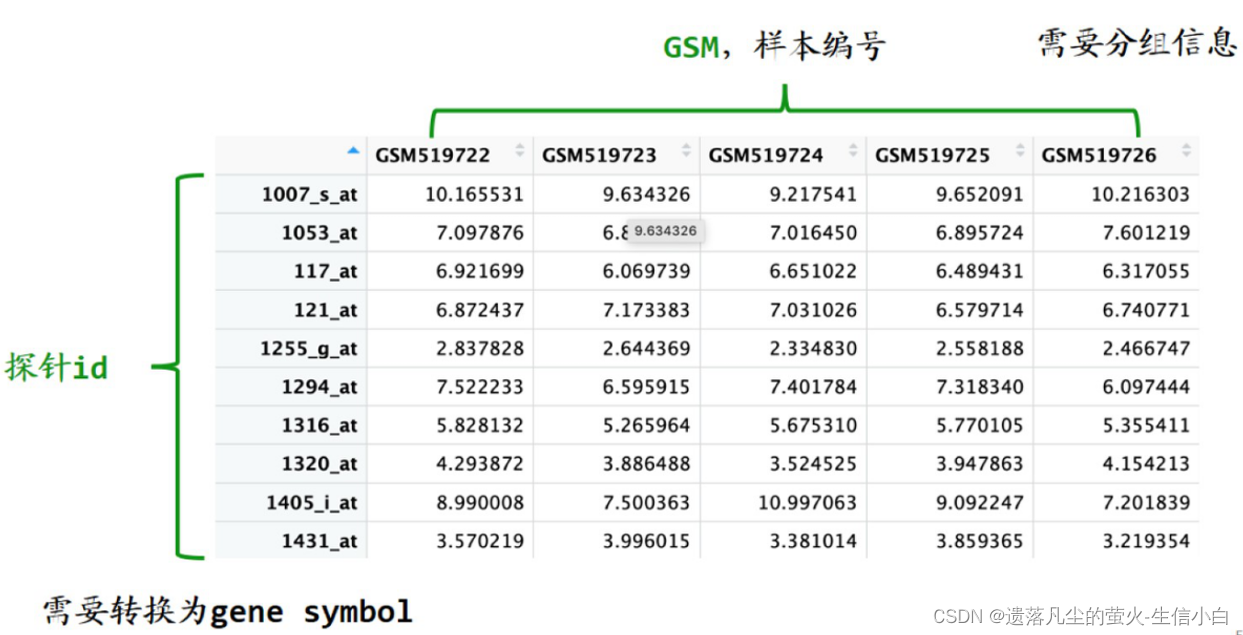
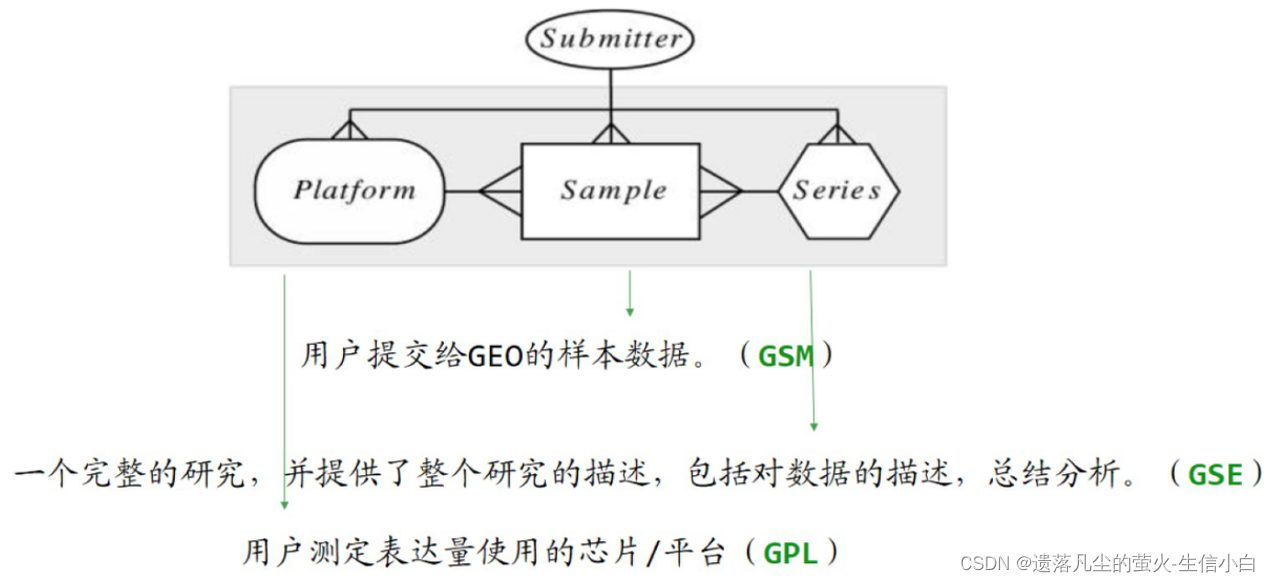
添加链接描述 点进去series查找自己感兴趣的数据(主要有两种数据类型:基因表达芯片和转录组测序),点开样本查看数据分布范围,有没有全部都在0附近,如果有大量负值,数据不正常。然后查看分组信息。 1.实验目的:通过基因表达量数据的差异分析和富集分析来解释生物学现象 病变组织vs健康组织 药物处理 vs 对照组(水/DMSO) 开花前vs开花后 ·动物/昆虫不同发育期 ·有色/无色果皮 高产/低产品种 有差异的材料→差异基因→找功能/找关联→解释差异,缩小基因范围 2.数据库介绍1.GEO首页有一个GEO2R网页工具,它可以给相应的代码,但是没有判断数据是否正常的机制,使用默认的参数,无法得到全部的默认的基因,代码仅供参考。 1.找数据,找到 GSE 编号 2.下载并读取数据 3.表达矩阵 4.临床信息 → 分组信息:需要自己整理 5.GPL 编号 → 探针注释(每个探针属于哪个基因,探针与基因的对应关系) 6.数据探索:分组之间是否有差异、PCA、热图(离散基因热图) 7.差异分析及可视化:limma包差异分析(P值、logFC)、画图(火山图、差异基因热图) 8.富集分析:GO、KEGG 1.探针 id→ 基因名称 2.GSM 样本 ID→ 分组信息
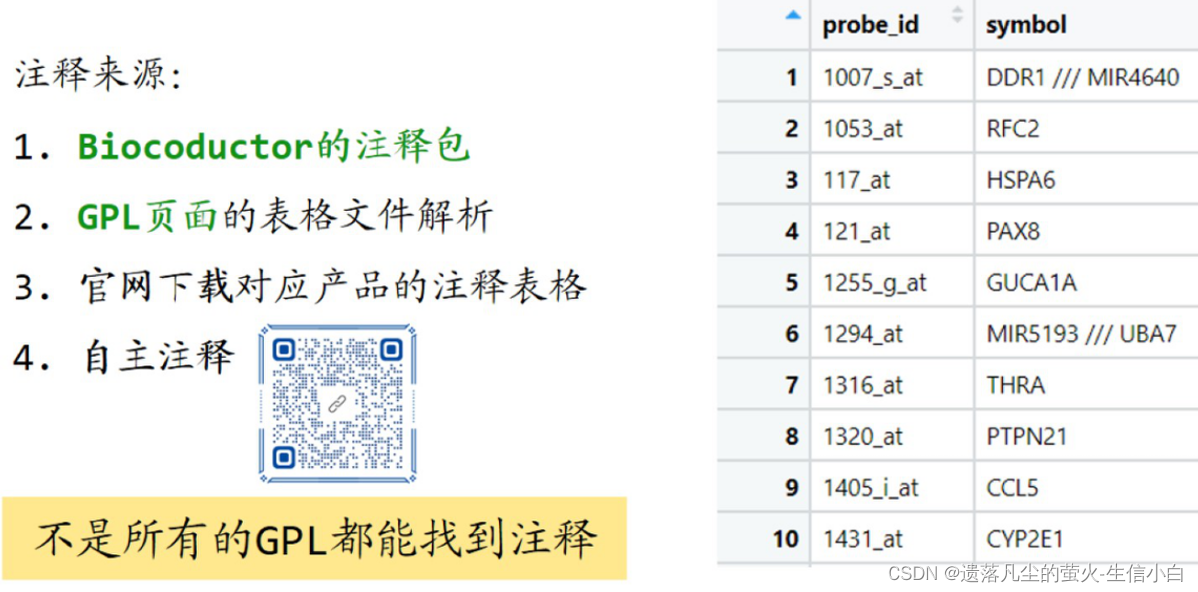
00_pre_install 全选运行安装里面的 R 包 require(pkg,character.only=T), 这个参数告诉 require() 函数,pkg 是一个字符向量。 下载数据,提取表达矩阵和临床信息 下载数据,大多数情况下只要在 R 中用代码下载即可 rm(list = ls()) #打破下载时间的限制,改前60秒,改后10w秒 options(timeout = 100000) options(scipen = 20)#不要以科学计数法表示 #传统下载方式 #getGEO() 函数是 GEOquery 包提供的一个工具,用于从GEO数据库下载和导入数据集。getGEO()有则直接读取,没有的话就帮忙下载并读取,在工作目录下 library(GEOquery) eSet = getGEO("GSE7305", destdir = '.', getGPL = F) #destdir = '.'是指下载后放在工作目录,getGPL是指不下载GPL文件,下载的话会变得很慢,不需要它网速太慢,下不下来怎么办? 1.从网页上下载/发链接让别人帮忙下,放在工作目录里 2.试试geoChina,只能下载2019年前的表达芯片数据 library(AnnoProbe) eSet = geoChina("GSE7305") #选择性代替eSet = getGEO("GSE7305", destdir = '.', getGPL = F)手动下载的方法: 1.GEO 首页-Series GSE:一个完整的研究,包括实验设计描述、数据描述、总结分析等GSM:用户提交给 GEO 的样本数据GPL:指用户用于测定表达量的芯片平台 2.搜索需要的疾病 3.按 array 筛选检测方法 4.点进一个 GSE 5.Series Matrix File 6.matrix.txt.gz 是表达矩阵的压缩包(至少应该是六七百 k) #下载下来后,研究一下这个eSet class(eSet) ## [1] "list" #返回列表里有几个元素 length(eSet) ## [1] 1 #应该是1,如果有2说明是一个SuperSeries里面有两批测序结果,如果不需要合并的话最好是分开提取,分开分析 eSet = eSet[[1]] #去掉列表数据,留下里边数据 class(eSet) ## [1] "ExpressionSet" ## attr(,"package") ## [1] "Biobase" #ExpressionSet :数据的对象的类型 提取表达矩阵exp exp #rep用法: # rep函数的其他用法?相间、两组的数量不同? > rep(c("Disease","Normal"),each = 10) [1] "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" [9] "Disease" "Disease" "Normal" "Normal" "Normal" "Normal" "Normal" "Normal" [17] "Normal" "Normal" "Normal" "Normal" > rep(c("Disease","Normal"),times = c(8,9)) [1] "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" [9] "Normal" "Normal" "Normal" "Normal" "Normal" "Normal" "Normal" "Normal" [17] "Normal" > rep(c("Disease","Normal"),times = 10) [1] "Disease" "Normal" "Disease" "Normal" "Disease" "Normal" "Disease" "Normal" [9] "Disease" "Normal" "Disease" "Normal" "Disease" "Normal" "Disease" "Normal" [17] "Disease" "Normal" "Disease" "Normal" # **第三种方法**,匹配关键词,使用字符串处理的函数获取分组,比较万能 k = str_detect(pd$title,"Normal");table(k) #按Normal这个关键词对pd$title中进行检测,有的话就是T,没有就是F Group = ifelse(k,"Normal","Disease") #根据T还是F赋值group #替换成功 > ifelse(k,"Normal","Disease") [1] "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" "Disease" [9] "Disease" "Disease" "Normal" "Normal" "Normal" "Normal" "Normal" "Normal" [17] "Normal" "Normal" "Normal" "Normal" #检查,#用pd的title列和group生成一个数据框放在一起,检查一下分组对不对 > data.frame(pd$title,Group) pd.title Group 1 Endometrium/Ovary-Disease 1 Disease 2 Endometrium/Ovary-Disease 2 Disease 3 Endometrium/Ovary-Disease 3 Disease 4 Endometrium/Ovary-Disease 4 Disease 5 Endometrium/Ovary-Disease 5 Disease 6 Endometrium/Ovary-Disease 6 Disease 7 Endometrium/Ovary-Disease 7 Disease 8 Endometrium/Ovary-Disease 8 Disease 9 Endometrium/Ovary-Disease 9 Disease 10 Endometrium/Ovary-Disease 10 Disease 11 Endometrium-Normal 1 Normal 12 Endometrium-Normal 2 Normal 13 Endometrium-Normal 3 Normal 14 Endometrium-Normal 4 Normal 15 Endometrium-Normal 5 Normal 16 Endometrium-Normal 6 Normal 17 Endometrium-Normal 7 Normal 18 Endometrium-Normal 8 Normal 19 Endometrium-Normal 9 Normal 20 Endometrium-Normal 10 Normal 需要把Group转换成因子,因子非常适合组织有重复值的向量,并设置参考水平,指定levels,对照组在前,处理组在后 Group = factor(Group,levels = c("Normal","Disease")) Group ##[1] Disease Disease Disease Disease Disease Disease Disease Disease Disease Disease ##[11] Normal Normal Normal Normal Normal Normal Normal Normal Normal Normal ##Levels: Normal Disease #levels(Group) ##[1] "Normal" "Disease" ##单独提取levels,查看Group向量里都有哪些取值,这就叫因子的水平,顺序对照组在前,处理组在后 探针注释的获取原理:探针与基因的对应关系表格,一个数据框 需要把probe_id和基因symbol对应起来:基因组注释 注释来源: 1.Biocoductor的注释包:添加链接描述 2.GPL页面的表格文件解析:找探针和symbol这两列,但不是所有文件都可以通过GPL页面找到这两列 |
【本文地址】
今日新闻 |
推荐新闻 |

 2.
2. 3.数据库数据
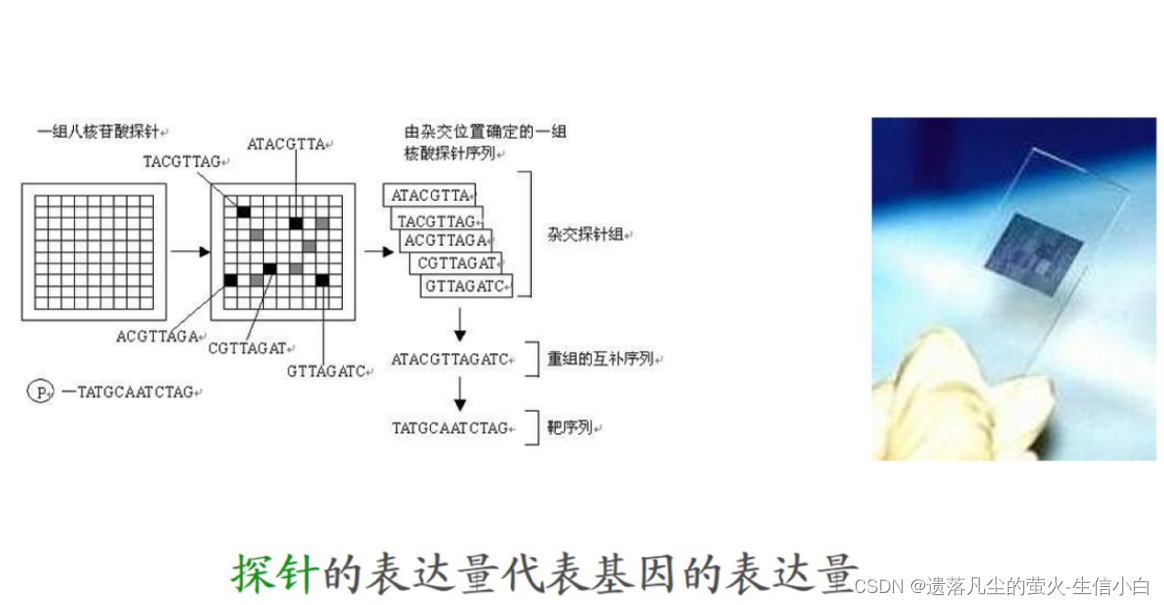
3.数据库数据 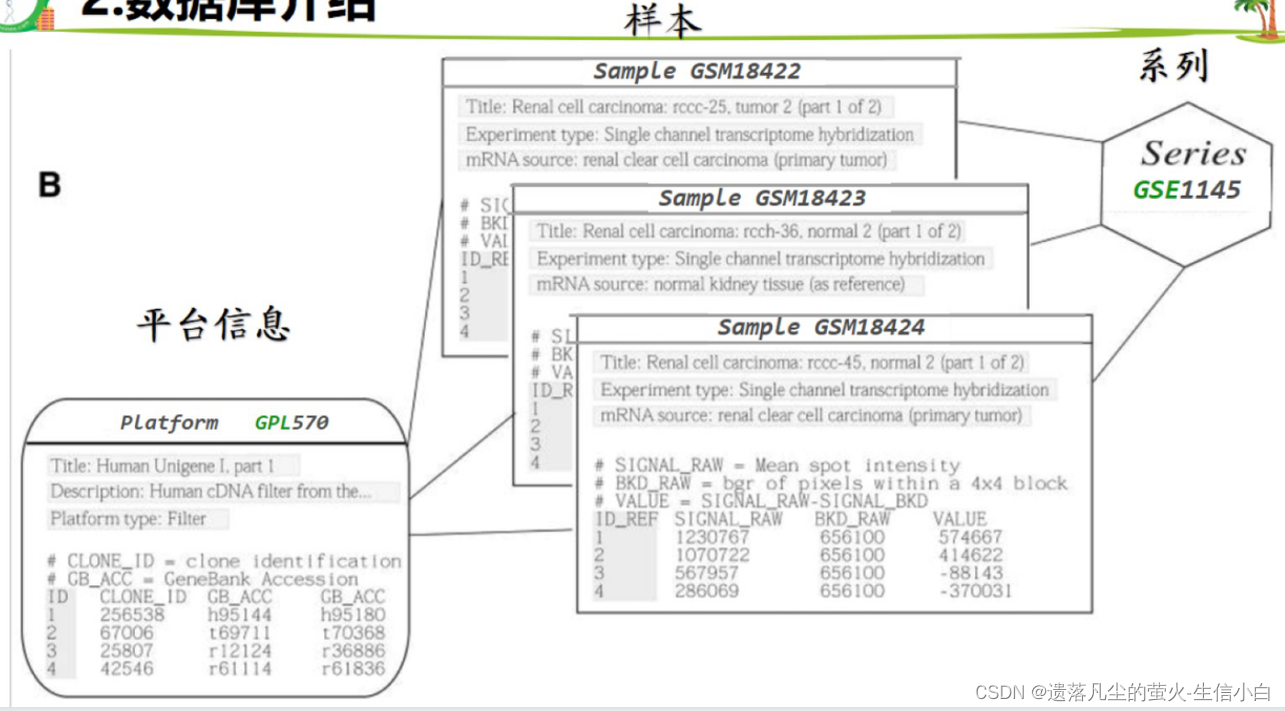 3.基因表达芯片的原理
3.基因表达芯片的原理