GO KEGG相关分析 |
您所在的位置:网站首页 › deseq2全称 › GO KEGG相关分析 |
GO KEGG相关分析
|
案例:胆道闭锁小鼠模型中肝外胆管和胆囊基因 研究目的 胆道闭锁(BiliaryAtresia,BA)是一种肝内外胆管出现阻塞并可导致淤胆性肝硬化天性胆道闭锁而最终发生肝功能衰竭的疾患。轮状病毒可导致小鼠患BA 疾病。因此我们利用轮状病毒感染新生小鼠构建的胆道闭锁的时间表达谱,分析疾病样本异常表达基因及其功能作用等,来阐述胆道闭锁模型的分子机制。 方法结果 一、实验设计 实验样本设计如下:盐水处理组织的对照样本,处理时间点为3天(Day 3 after normal saline injection,简称Day3_NS)、7天(简称Day7_NS)、**天(简称Day**_NS),每个时间点3个样本;轮状病毒处理组织的实验样本,处理时间点为3天(简称Day3_RRV)、7天(简称Day7_RRV)、**天(简称Day**_RRV),每个时间点3个样本。制作芯片。 二、表达数据预处理及差异表达分析 芯片数据首先使用R软件包RMA法预处理。随后将探针转换为对应的gene symbol。对于多个探针匹配同一个gene symbol,取这些探针均值作为基因最终表达值。 计算分析Day3_RRV vs. Day3_NS、Day7_RRV vs. Day7_NS、Day**_RRV vs. Day**_NS,三个对比组的差异表达基因。使用R包提供的T检验的方法计算差异表达基因的显著性P值。并使用BH方法矫正P值(即FDR)对于每个显著差异表达基因,要求其差异表达的FDR小于0.051, 51, 51); font-family: 宋体;">5,同时要求log2FC的绝对值不小于1, 51, 51); font-family: 宋体;">1。 利用基因表达值可对多个数据集进行文氏图分析,快速筛选出重要基因。我们输入3个时间点差异表达基因和基因的log2FC值,观察3个时间点差异表达基因的异同。文氏图分析结果见图1。有115个上调基因同时出现在3个时间点中,但是下调基因只有1个。后续进一步分析这116个基因。  图1 VennPlex文氏图分析结果。0表示contra-regulated,指表达方向相反的基因,如day3和day7有1个表达方向相反的基因 三、差异表达基因功能富集分析 使用富集分析工具分别分析每个时间点中上调和下调基因分别参与GO功能和KEGG通路。参数富集基因个数count>=2,超几何检验显著性阈值Pvalue 图2 转录因子和靶基因调控网路,红色节点属于交集基因,三角形节点表示转录因子
参考:基因(蛋白)功能注释分析和功能富集分析(GO、KEGG) 下图展示了,GO KEGG相关分析的流程,输入差异基因集和背景基因集、对应的GO注释和KEGG注释,得到GO和KEGG富集结果,并绘富集结果制散点图和柱状图。包含,GO富集结果、KEGG富集结果、GO富集散点图、GO富集柱状图、GO有向无环图、KEGG富集散点图。  一、基因的差异表达分析 一、基因的差异表达分析参考:差异基因筛选方法 对于基因的差异表达分析,能够发现一组在正常样本和患病样本中表达不同的基因,这为生物工作者进行实验验证提供了较好的候选基因。 通常的检测是对两种不同实验条件下的差异基因表达的问题进行模式化,一种检验对应一种基因,如果基因的表达值是零假设,那么它是无差异的。差异基因的筛选方法有很多,最简单的是阈值法,用倍数分析基因表达水平差异,即计算基因在两个条件下表达水平的比值,确定比值的阈值,将绝对值大于此阈值的基因判断为差异基因。另外还有些方法包括统计学的 T 检验法和 SAM 等方法。 倍数变化法 倍数变化法 (Fold change),计算患病组和正常组的表达值的差异倍数,是用于检测差异表达基因的最基本的方法,由于其简单,易理解和不错的实验结果,使得其成为差异表达直观分析的首要选择。 整体而言,Fold Change 方法在探测差异表达基因时,能够直接的得到差异变化值,因此在与差异表达绝对值相关的研究时具有优势。但是其较难选定其所需的阈值,在缺少假阳性的控制的情况下,其检测的基因假阳性结果比率相对较高。 T 检验法 T-test 检验是差异基因表达检测中常用的统计方法,通过合并样本间可变的数据,来评价差异表达,用于判断某一基因在两个样本中是否有差异表达。 由于芯片实验成本较高,样本量较少,从而对总体方差的估计不很准确,T 检验的检验效能降低。 SAM 算法 SAM 算法就是通过控制 FDR 值纠正多重假设检验中的假阳性率。SAM 方法检验差异表达,通过对分母增加一个常量 T 检验过程减小了假阳性发生的概率。根据文献记载,相比较其他算法,SAM 算法更为稳定,筛选出的结果也更为准确。 SAM 方法以 q-value< 0.05 作为筛选差异表达基因的标准,从公式上来看,p-value 和 q-value 较为相似,而差异筛选是一个典型的多重假设检验过程。对于多重假设检验,单次检验中差异显著基因的假阳性率 (p-value 较小) 可能会较大,而 q-value 和 FDR 值较常见的 BH 校正方法得到的 FDR 值而言,改进了其对假阳性估计的保守性。  差异基因分析 参考:小L生信学习日记-6丨你最关心的差异基因是怎么挑出来 RNA-seq数据分析 09:DESeq2差异表达分析 做基因表达分析时必然要做差异基因分析,做差异基因分析最常用的软件就是DESeq2,使用DESeq2对两组进行比较,得到显著性P-value,P.adjust值和基因间的差异倍数(Fold Change)。根据这三个统计学参数来筛选差异基因,一般设置的筛选条件为:p-value 上图列头及其含义说明: Gene_id:基因在Eesembl数据库的编号Gene_Symbol:基因名称,与NCBI GenBank的基因标识一致Chr:该基因所在染色体名称Start:该基因在染色体上的起始位点End:该基因在染色体上的终止位点Strand:该基因所在染色体所属的链的属性。'+'表示正链,'-'表示负链Length:该基因的长度log2FoldChange:log2的差异倍数。本次取值大于1.5倍或者小于0.666667倍。p-value:p值,用来表示差异基因的显著性水平,本次取p值小于0.05。padj:p 值校正值 FDR,即对相同条件下的试验的差异显著性进行多重检验校正。前面几个列头代表的意思很容易理解,我们最关注的是log2FoldChange、P值和Padjust值 P-value:p值,是统计学检验变量,代表差异显著性,一般认为P < 0.05 为显著, P ▲FC的计算方式,有兴趣可以详细看。 用上面的方法筛选到差异基因之后, 得到了非常多的数据。直接查看数据不方便观察整体情况,我们一般会将数据做成火山图来展示组间差异的整体情况。 火山图 火山图从差异倍数(Fold Change)和差异显著性水平(p-value)两个方向对组间差异进行评估,如图:  横轴:log2FoldChange,对fold change值取log2。横坐标的绝对值越大,说明表达量在两样本间的表达量倍数差异越大。 纵轴:-log10P-value,对P-vallue取-log10。纵坐标值越大,说明差异基因表达越显著,筛选得到的差异表达基因越可靠。 图中差异倍数大于1.5且p值小于0.05的为上调基因, 用红色点展示;差异倍数小于0.666667并且p值小于0.05的为下调基因, 用绿色点展示;其中非显著差异的基因用灰色点展示。 上面,我们提到了上调基因、下调基因。那么,什么是上调基因,下调基因?简单解释一下: 上调基因:up-regulated gene,相对于对照组,在实验组中该基因转录成mRNA时受到正向调控,表达量增加。下调基因:down-regulatedgene,相对于对照组,在实验组中该基因转录成mRNA时受到抑制,表达量减少。差异基因聚类分析 基因的作用不是孤立的,因此对差异基因进行层次聚类分析,从聚类分析结果中发现样本组内和组间的相关性情况。 (顾名思义,聚类分析可以基于分析对象的相似性将其各自分成不同的组,使组内样本相似,组间样本有差异。) 根据每个基因在样本中的信号值来分别对基因和样本进行聚类分析,聚为一类的基因群在样本中具有相似的表达情况,这说明它们可能具有相似的功能。 聚类结果用heatmap(热图)进行展示:  横坐标:样本名称 纵坐标:差异基因 从图上可以看出,样本分为“xxxC”和“xxx43”两组,红色代表该差异基因在分组样本中表达值高,绿色代表差异基因在分组样本中表达值低。不同列代表不同的样本 二、基因(蛋白)功能注释分析和功能富集分析(GO、KEGG)基因(蛋白)常见的功能分析方法有:代谢信号通路(pathway)和GO(Gene ontology,基因本体论)分析。另外还有COG(Clusters of Orthologous Groups of proteins)、蛋白功能域(protein Domain)等分析。 GO和pathway都是研究基因功能的,那么它们的区别是什么呢? GO主要是研究基因功能的,而pathway是研究基因和蛋白功能的。 GO功能主要分成三大类:BP(生物学过程)、MF(分子功能)和CC(细胞组成);其中最常用的就是GO BP分析。常见的pathway数据由KEGG、Reactome、Biocarta等。  功能分析主要分成两类:功能注释分析和功能富集分析。 功能注释分析是指对基因进行GO、pathway的注释(Annotation),例如DDR1基因参与GO:0001558 regulation of cell growth、GO:0007155 cell adhesion、GO:0031100 organ regeneration等20个生物学过程(GO BP)。 功能富集分析是指对一个基因集(gene sets)进行富集分析,使用超几何分布算法获得该基因集中的基因显著富集的功能。一般会有一个显著性的阈值,例如p 图例: A代表通路图;B代表差异上下调基因的GO富集分析;C代表KEGG pathway富集分析; D代表GO和pathway富集分析 KEGG数据库(一)KEGG数据库介绍 数据挖掘中最后一步往往是KEGG和GO分析,那么KEGG究竟是什么呢? KEGG,全称Kyoto Encyclopedia of Genes and Genomes,是一个从分子水平信息,特别是基因组测序和其他高通量实验技术产生的大规模分子数据库,以了解细胞、有机体和生态系统等生物系统的高级功能和效用的数据库资源。  以获得诺奖的氧感知研究相关的HIF为例,我们来看一下这个数据库。  搜索框中键入HIF,找到该因子相关的通路。  进入该通路,可以看到关于HIF-1信号通路的介绍: 首先是总体的描述: 缺氧诱导因子1 (HIF-1)是一种转录因子,是调节氧稳态的主要因子。它由两个亚基组成:诱导表达的HIF -1α亚基和组成表达的HIF -1β亚基。在常氧条件下,HIF-1在特定的前体残基上发生羟基化,从而导致亚基的立即泛素化和随后的蛋白酶体降解。相反,在缺氧条件下,HIF-1亚基变得稳定,并与p300/CBP等共激活因子相互作用,调节其转录活性。最终,HIF-1在缺氧条件下成为众多缺氧诱导基因的主调控因子。HIF-1的靶基因编码增加氧气O2传递的蛋白,介导对O2剥夺的适应性反应。尽管它的名字是HIF-1,但HIF-1的产生不仅是由于氧可获得性降低,而且还包括由其他刺激物,如一氧化氮或各种生长因子引起的。  之后是通路图,我们点开它,可以看到整个HIF-1信号通路  同样的,方框标记的因子可以进一步点开介绍:  可以看到缺氧诱导因子羟化酶(PHD)的介绍   在另一种KO表现形式中,方框中的因子背景被标记成了紫色。  同样,我们也可以对物种进行选择。  回来看,通路之后是有关疾病的介绍,可以看到HIF-1与糖尿病视网膜病变、恶性副神经节瘤等疾病的发生发展有关  之后是大量的引文,我们在作者中发现了诺奖获得者之一彼得·拉特克利夫(Sir Peter J. Ratcliffe)  我们再看一下肝癌的有关通路:   我们可以看到,正常肝脏经历肝炎、肝硬化、增生结节、早期肝硬化和转移的过程中的信号通路。患者在酗酒、吸烟等高危因素下,感染乙肝、丙肝病毒,成为肝炎携带者。之后TGF和IGF-Ⅱ等因子过表达,分别作用于细胞表面的EGFR和IGFR,经过钙离子、PI3K-Akt等信号通路后,引起一系列病理生理反应,造成不典型增生、DNA损伤、细胞坏死等,影响预后。 (二)KEGG使用教程 参考:数据库|最全的KEGG使用教程在这里! (yunbios.net)1. KEGG数据库 KEGG是一个综合数据库,它们大致分为系统信息、基因组信息、化学信息和健康信息四大类。进一步可细分为15个主要的数据库。  KEGG PATHWAY是最核心的数据库之一,该数据库是一个手工画的代谢通路的集合,包含以下几方面的分子间相互作用和反应网络。  代谢(Metabolism),遗传信息处理(Genetic Information Processing),环境信息处理(Environmental Information Processing),细胞过程(Cellular Processes),有机系统(Organismal Systems),人类疾病(Human Diseases),药物开发(Drug Development),该表只列出了部分代谢通路,详细的代谢通路信息见:https://www.kegg.jp/kegg/pathway.html。 2. KEGG通路中ko和K等编号说明  例如:ko04010 -> KEGG Ontology hsa00010 -> 人特有代谢通路 M00072 ->MODULE K10258 -> KEGG orthologs rn00110 -> pathway Reaction R00259 -> KEGG Reaction C00083 -> compound EC:1.3.1.93 -> enzyme 3. 如何理解KEGG通路-ko查询KEGG的代谢通路的具体信息,以ko04010为例。  查询结果如下: 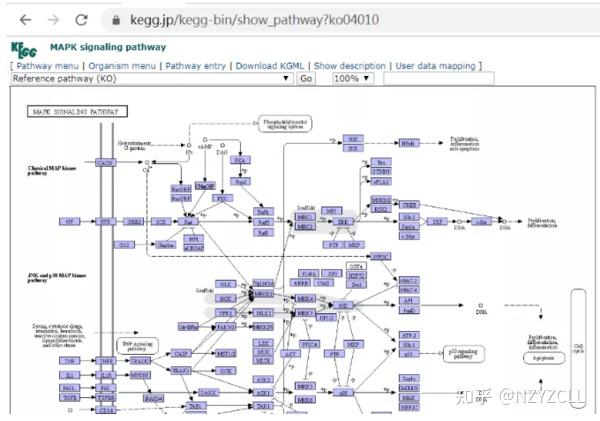 其中长方形节点表示基因产物(如酶或一些RNA调节子),所有蓝色背景的基因产物都属于KEGG ORTHOLOGY(KO)分类体系(序列高度相似,并在同一条通路上有相似功能的蛋白质被归为一组KO),而白色背景的基因产物则不在KO分类体系之列。 通路中每个元素类型说明:   数据来源:https://www.genome.jp/kegg/document/help_pathway.html 4. 如何理解KEGG的蛋白和酶促反应编号查询KEGG的蛋白质或者酶分类编号的具体功能描述和参与代谢通路方法如下,以K10258为例。  查询结果如下:  查询KEGG的酶促反应编号的具体信息如下,以R00259为例。  查询结果如下:  5. 蛋白序列在线KEGG注释 5. 蛋白序列在线KEGG注释在基因组研究中,特别是当组装了一个新物种的基因组,我们首先要做的就是注释,从而大致推断这些基因的功能。KEGG提供了在线注释功能,进入KEGG中自动注释工具界面,KAAS(http://www.genome.jp/tools/kaas/),如下图,以KAAS job request(BBH method)为例。  点击KAAS job request(BBH method)进入注释界面,提交注释序列:  信息填写完成之后,点击右下角“Compute”按钮,提交成功。注释结果会以邮件的形式发送。 GO数据库介绍参考: 三、功能富集分析在线绘图网站这里重点推荐一些在线绘图网站,能简化不少麻烦: 生信在线生物信息学数据可视化 网址:http://www.bioinformatics.com.cn/ 1.微生信可以绘制多种图形,如聚类热图、GO富集弦图(GO chord plot)、火山图(Volcano plot,ps可能不如Origin软件绘制的火山图美观)、韦恩图(Venn diagram)等等,界面左侧显示能作的图形种类,右侧是具体图形 微生信能够作的生信图形种类实在太多,等待大家自己探索与使用。  2. 联川生物在线分析与绘图网址:http://www.lc-bio.cn/overview包括多种微生物分析、富集分析以及各种统计图的绘制     3. 多组venn图绘制的在线神器推荐 Venny 2.1网址:https://bioinfogp.cnb.csic.es/tools/venny/index.html 2. Draw venn diagram 网址:http://bioinformatics.psb.ugent.be/webtools/Venn/  4. BioRender这个之前已经有介绍过,功能强大,不过需要付费,有些小伙伴表示没钱,无能为力,感兴趣可以看看原回答 5. http://Draw.io在线绘图免费、适用于多平台,无需安装!适用于流程图、思维导图、图表等的制作。网址:https://www.draw.io/  模式图的绘制可谓是八仙过海各显神通,有人使用PS,有人使用AI,还有人使用PPT。选择自己顺手的就好。 参考:生信绘图只能用R语言吗? |
【本文地址】
今日新闻 |
推荐新闻 |