小众或非模式生物的自建库GO/KEGG富集分析 |
您所在的位置:网站首页 › david数据库go富集分析 › 小众或非模式生物的自建库GO/KEGG富集分析 |
小众或非模式生物的自建库GO/KEGG富集分析
|
本人已委托维权骑士(http://www.rightknights.com)进行原创维权。 思想高屋建瓴,知识原创共享。 转载请引用链接。 在之前的富集分析系列文章(糖糖家的老张:最全的GO, KEGG, GSEA分析教程(R),你要的高端可视化都在这啦![包含富集圈图])中,向大家介绍了GO、KEGG富集分析中各种天花乱坠的可视化来让你的文章或汇报更高端,但无论是GO(基因本体)数据库还是KEGG(京都基因和基因组百科全书)数据库,都只能在官方支持的物种(此处暂称为模式生物,实际是充分但不必要的关系)中进行分析与可视化。 这就带来了一个问题:如果科学家研究的物种比较小众,或者是一类变异的亚群,如何针对这种小众或非模式生物进行GO与KEGG富集分析呢?我咨询了两家小有名气的生物科技公司如何解决这个问题,公司表示无法提供官方数据库以外的个性化服务。 事实上,我们可以通过自己建立该物种的GO或KEGG的数据库来解决。 首先我们先来看看GO和KEGG数据库官方支持的物种有哪些? GO数据库支持19个物种的分析,实在是少得可怜,研究微生物的课题组那么多,提供生信服务的公司也不少,我问了一圈没想到大家的回答基本都是GO分析不支持我们就不做GO了。 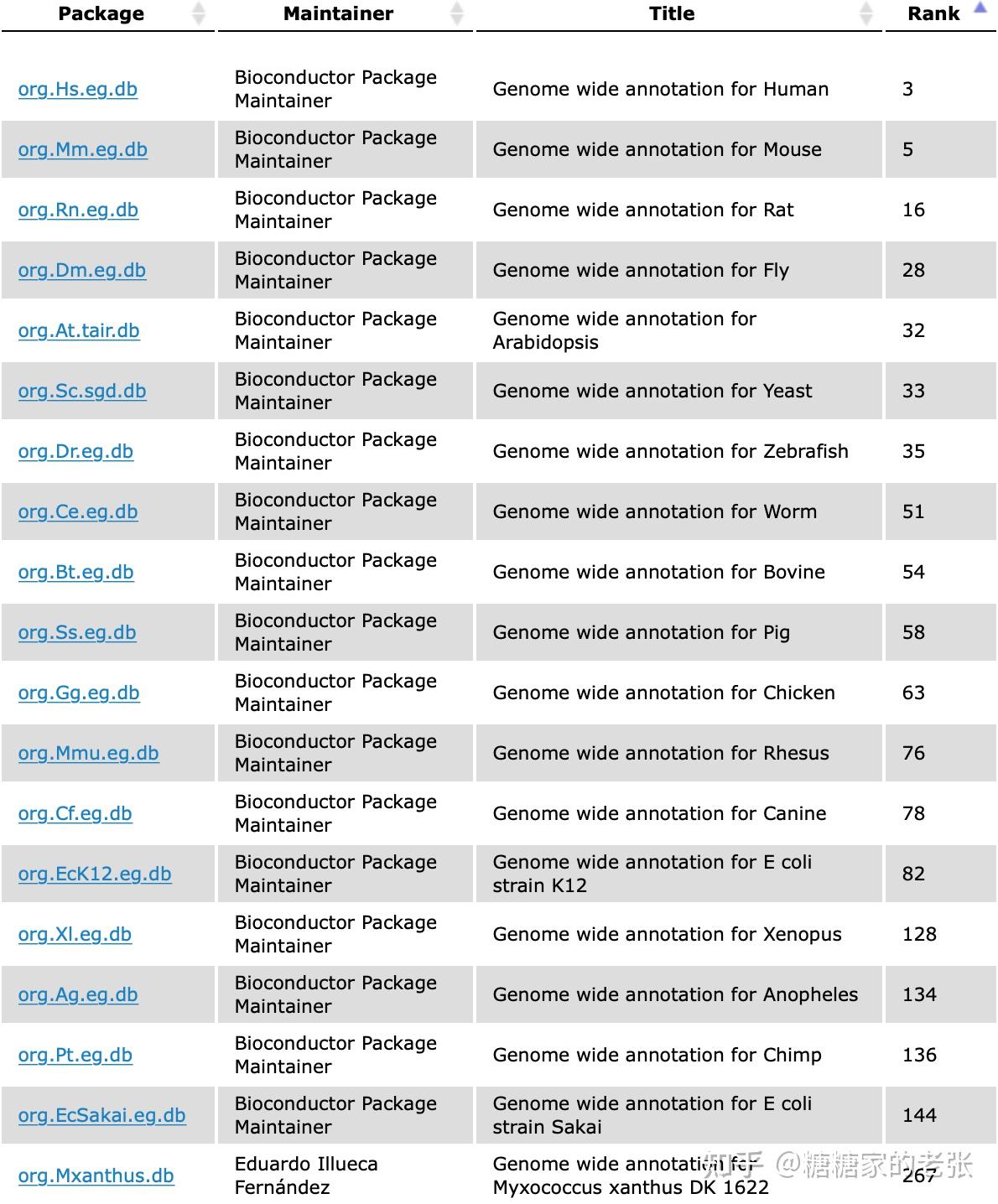 KEGG数据库支持的物种倒是很丰富,但是也难以做到适用任意种群的生物。 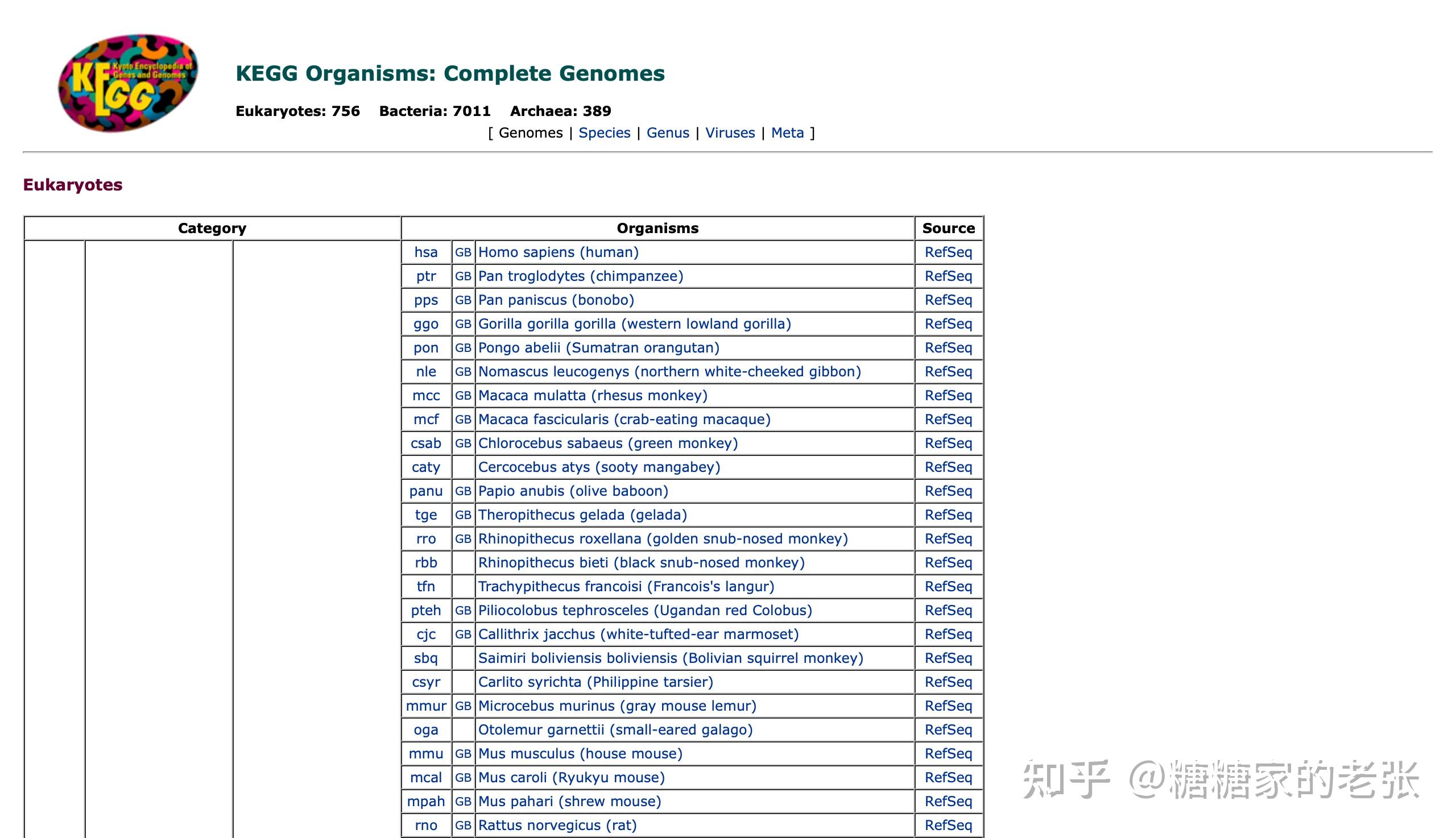 综上所述,GO和KEGG富集分析的使用之广而官方支持的物种之少都表明针对小众或非模式生物的自建库分析是研究和市场都亟待解决和普及的重要内容。接下来把思路和代码公开出来供大家参考。 一.准备工作1.蛋白质组序列从 https://www.ncbi.nlm.nih.gov/genome/ 可以检索和下载到所需物种的基因组及蛋白质组序列,当然,也可以通过其他方式自行准备。 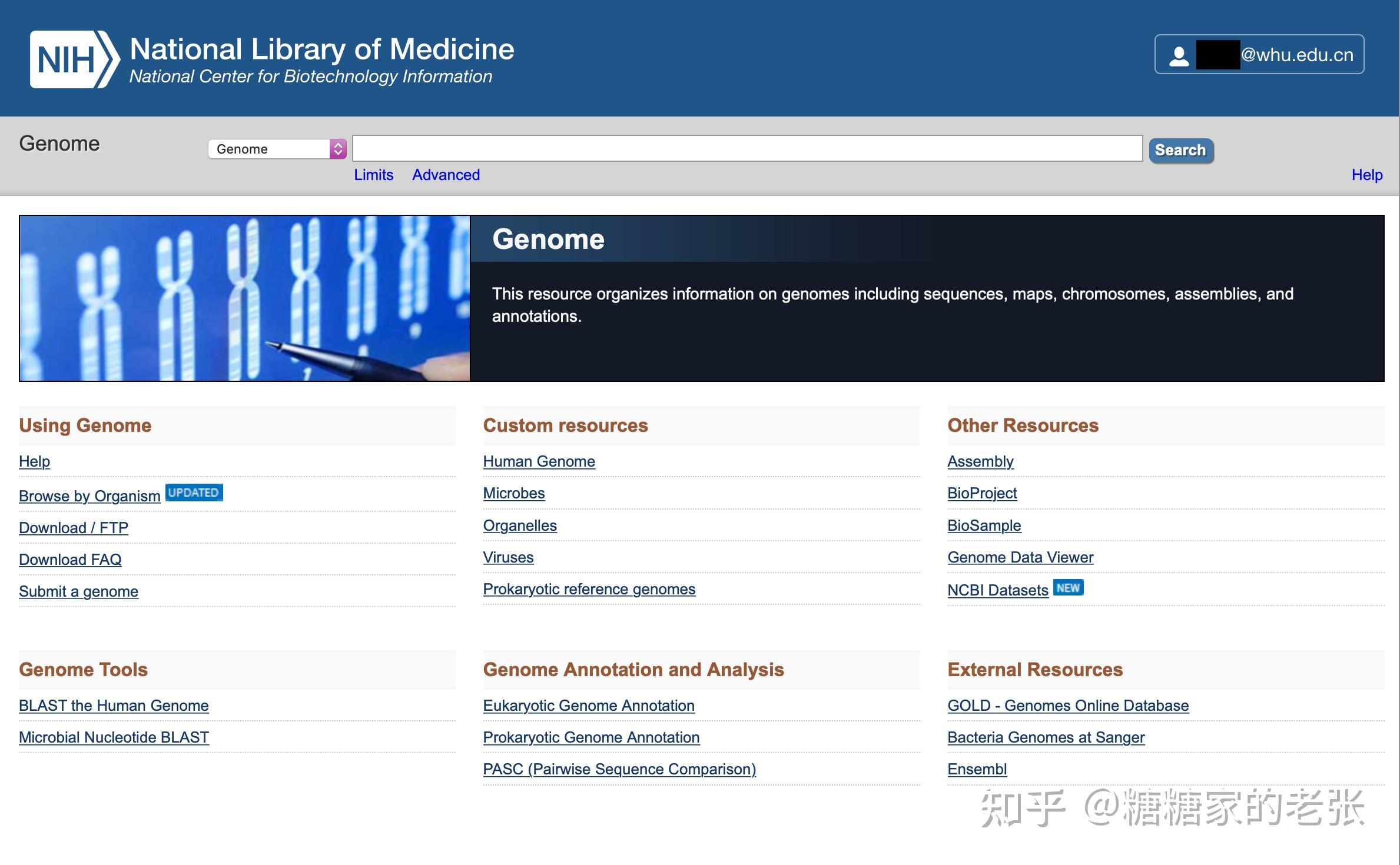 2.KEGG数据库官方的基因注释信息文件 2.KEGG数据库官方的基因注释信息文件从 可以下载到KEGG数据库对所有基因的注释信息j |
【本文地址】
今日新闻 |
推荐新闻 |