清华大学刘俊杰团队开发新型Casπ基因编辑系统,并揭示其DNA识别切割机制 |
您所在的位置:网站首页 › crispr-cas系统原理 › 清华大学刘俊杰团队开发新型Casπ基因编辑系统,并揭示其DNA识别切割机制 |
清华大学刘俊杰团队开发新型Casπ基因编辑系统,并揭示其DNA识别切割机制
 CRISPR-Cas系统作为细菌、古菌以及巨噬菌体抵抗外源核酸入侵的一种适应性免疫系统,已被广泛应用于基础生物学研究、农业育种、人类疾病治疗以及体外核酸检测等方面。 近年来的研究根据Cas核酸酶的种类将CRISPR-Cas系统具体分为2个大类,6个亚型,其中V型Cas12系统的种类和功能最为丰富多样且存在多种较小分子尺寸的核酸酶系统,如CasX(Cas12e)、Cas14(Cas12f),CasΦ(Cas12j)以及Cas12i等。尽管研究者通过理性设计和定向进化等方法提升了这些系统的基因编辑效率,但是目前已经鉴定的Cas12蛋白只能识别富含T的PAM(T-rich PAM),这严重阻碍了Cas12家族的体内基因编辑以及体外临床检测等的应用。 近日,清华大学生命学院、北京生物结构前沿研究中心刘俊杰课题组在 Cell Research 期刊发表了题为:The compact Casπ (Cas12l) ‘bracelet’ provides a unique structural platform for DNA manipulation 的研究论文。 该研究报道了一种自主鉴定的、名为Casπ(Cas12l)的新型CRISPR-Cas12家族核酸酶, Casπ核酸酶通过识别CCN PAM来切割底物DNA,能在哺乳动物细胞中实现有效的基因编辑。此外,该研究还解析了Casπ蛋白结合sgRNA与底物DNA的复合体结构,揭示该系统底物识别机制,为RNA引导的核酸酶系统的进化和改造提供了新的视角。  刘俊杰(Jun-Jie Gogo Liu)课题组长期关注DNA和RNA核酸酶研究及相关核酸操纵工具的开发和应用。近年来,刘俊杰及其合作者发现并鉴定了一类可用于基因编辑操作、识别T-rich PAM的小型CasX核酸酶(Nature,2019),并通过蛋白质和sgRNA的结构改造进一步提升了该系统的基因编辑效率(Molecular Cell,2022)。 为了寻找区别于已知家族的全新CRISPR-Cas系统,研究团队首先构建了一套优化的生物信息挖掘流程,对Cas蛋白的RuvC结构域序列构建隐马尔可夫模型(HMM),利用多轮迭代,在维持Cas核心特点的同时,增加差异度。利用该迭代模型结合多种CRISPR-Cas预测方法,在北京和天津的城市污水宏基因组数据中,发现一类新型非致病菌来源的小型CRISPR-Casπ(Cas12l)系统,Casπ蛋白仅由850-867个氨基酸组成,属于Class 2 Type V CRISPR-Cas系统(图1)。 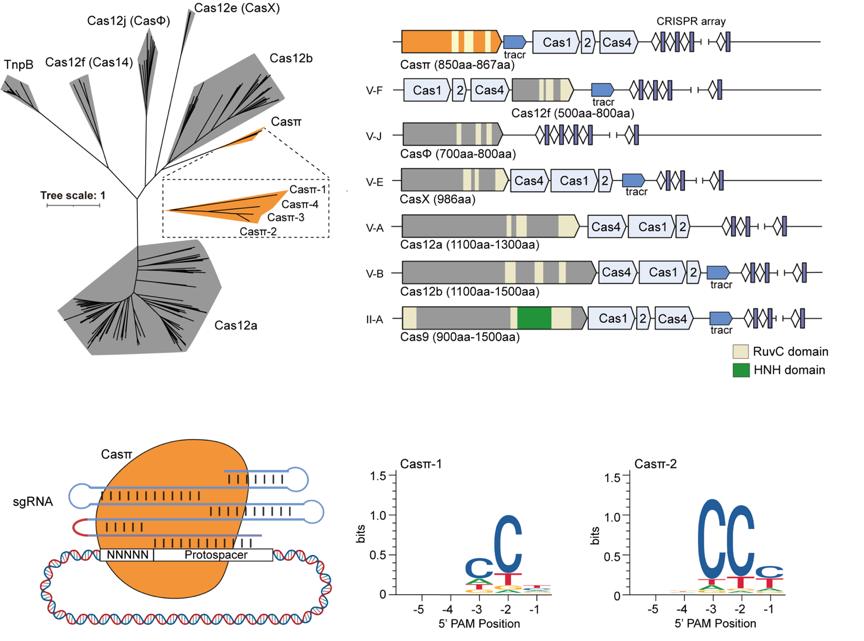 图1:CRISPR-Casπ系统的挖掘、鉴定和PAM筛选分析 通过PAM筛选实验证明,Casπ核酸酶识别CCN PAM,这一性质与以往报道的Cas12家族蛋白均识别T-rich PAM不同,拓展了CRISPR基因编辑工具的靶标广度(图1)。Casπ的顺式切割和反式切割活性与广泛使用的LbCas12a(1228氨基酸)活性相当。系列的生化评估表明,区别于一般小型CRISPR-Cas蛋白脆弱的耐受性,Casπ核酸酶对高低温度、不同盐浓度和超高蛋白浓度均具有良好的耐受度,并能被多种二价离子激活,生化鲁棒性强,为Casπ的核酸检测应用奠定了基础。 为了进一步探索Casπ系统的基因编辑潜力。研究团队首先尝试了该系统在原核生物中的DNA切割能力,结果表明,靶向毒蛋白基因ccdB的Casπ系统,可有效解除ccdB毒性对大肠杆菌的生长抑制。进一步,研究团队利用Casπ系统对HEK293A细胞系基因组中插入的移码突变GFP报道系统位点(out-of-frame EGFP cell line)和内源基因位点进行编辑操作,并利用深度测序,分析和评估了编辑结果。结果表明,还未经改造优化的Casπ可对真核生物基因组产生有效编辑,与LbCas12a效率相当,最高可达到SpyCas9编辑效率的一半(图2)。有意思的是,在片段删除的编辑类型中,Casπ主要产生 |
【本文地址】
今日新闻 |
推荐新闻 |