R语言中缺失值NA的处理 |
您所在的位置:网站首页 › cox回归列线图R语言调用内插至少需要俩个非NA值的数据 › R语言中缺失值NA的处理 |
R语言中缺失值NA的处理
|
一般在项目中,数据可能会因为设备故障、未作答问题或误编码数据的原因不完整。在R中NA(not available,不可用)表示缺失值。
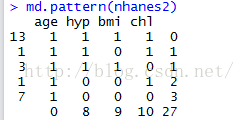
函数is.na()允许你检测缺失值是否存在。该函数作用于检测对象之后将返回一个相同大小的对象,其中缺失值的位置被改写为true,其他不是缺失值的位置则为false。 > which(is.na(nhanes2)) #返回缺失值的位置 > sum(is.na(nhanes2)) #计算数据集nhanes2中的缺失值总数 > sum(complete.cases(nhanes2)) #统计数据集中完整样本的个数 可以通过md.pattern()获取缺失值的分布情况(mice包),其中1表示没有缺失数据,0表示存在缺失数据:
第一行第一列的13表示有13个样本数据是完整的,倒数第二行7表示有7个样本少了hyp、bmi、chl三个变量,最后一行表示各个变量缺失的样本数合计。 1.剔除缺失部分确定缺失值以后要在分析数据前删除这些缺失值。因为含有缺失值的算术表达式和函数的计算结果也是缺失值。 一些函数计算时拥有na.rm=TRUE,可以在计算以前移除缺失值并使用剩余值进行计算;可以通过函数complete.cases()检查变量中至少含有一个缺失数据的观测值的个数。函数compete.cases()产生一个布尔值向量,该向量的元素个数与数据框中的行数相同,如果数据框的响应行中不含NA值,函数返回值就是TRUE。 函数na.omit()移除所有含有缺失值的观测,na.omit()可以删除所有含有缺失数据的行。但踢出所有包含缺失值记录的方法是很极端的,这样处理缺失值太多的样本则样本数据几乎没有意义。 manyNAs(algae,0.2)可以找出数据集algae中缺失值个数大于列数20%的行,第二个参数中可以设置一个精确的列数作为界限。 2.用最高频率来填补缺失值填补含有缺失值记录的另一个方法是尝试找到这些缺失值最可能的值。对于变量分布近似正态分布时可以选用平均值;偏态分布一般采用中位数代表数据中心趋势的指标。 样本algae第48行变量mxPH有缺失值,该变量分布近似正态分布,采用平均值填补缺失值: > algae[48,"mxPH"] algae[is.na(algae$Chla),"Chla"] algae cor(algae[,4:18],use = "complete.obs")参数use = "complete.obs"可以忽略含有NA的记录,另外函数symnum()可以输出用符号表示相关值的形式。测试数据集中,相关值大于0.9的两个变量可以通过相关性填补这两个变量的缺失值。以P04和oP04为例,需要先找到这两个变量之间的线性相关关系: > lm(PO4~oPO4,data = algae) 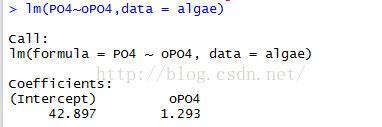 函数lm()可以用来获取线性模型。得到PO4=42.897+1.293*oPO4
函数lm()可以用来获取线性模型。得到PO4=42.897+1.293*oPO4
可以使用上述线性关系计算变量的缺失值,填补样本28在变量PO4上的缺失值: > algae[28,"PO4"] algae donate accept sa sa |
【本文地址】
今日新闻 |
推荐新闻 |