pymol命令行 |
您所在的位置:网站首页 › cmdcolor命令 › pymol命令行 |
pymol命令行
|
pymol中,改变颜色用到的是 color函数,想知道color怎么用的可以在命令行输入:help color 如下图,希望将A链和C链染成橙色,而B和D则染为红色。(首先show_as cartoon只显示蛋白质的卡通结构) show_as cartoon color orange, chain A+C color red, chain B+D 由于之前我进行了show_as cartoon操作,只展示了蛋白质大分子,而配体小分子被隐去了,可以用下面的代码显示出小分子的棍棒结构: show sticks, not polymer # 除了蛋白质大分子,其他都显示棍棒结构然后我想将小分子中的N原子、O原子等染成其他颜色,而碳原子保持不变,就可以: color atomic, not elem C # atomic就是根据原子类型染色,然后再排除掉C元素因为这个蛋白(PDBid:1a00)的小分子配体是HEM,resn(残基名称)就是HEM  RCSB PDB 官网查到的配体叫HEM RCSB PDB 官网查到的配体叫HEM我想以A链的HEM小分子为中心视角,可以这么做: orient resn HEM and chain A # 这里的and就是python的逻辑与,表示同时满足两个条件的才行 保存指令,直接在pymol命令行一次性粘贴 保存指令,直接在pymol命令行一次性粘贴如果要进行多次这样的操作,可以将所有的命令存在一个文本文档中,下次打开pymol的时候直接一次性粘贴输入所有的命令即可! fetch 1a00 show as cartoon color orange, chain A+C color marine, chain B+D show sticks, not polymer orient resn HEM and chain A color atomic, not elem C在终端直接运行.pml文件或者将一行一行的命令直接保存为pml格式的脚本: 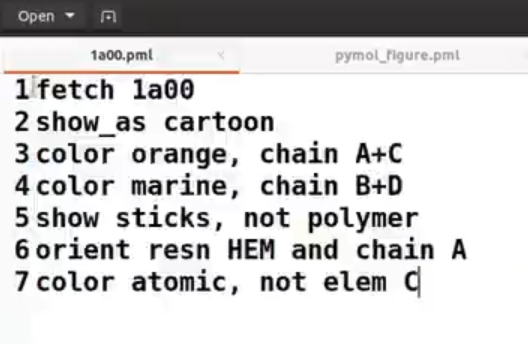 让我们来看下面这个pml文件脚本:  在终端执行:  会蹦出pymol的窗口,然后就可以看到已经执行完毕:  显示相互作用使用Python来实现 显示相互作用使用Python来实现也可使用python的 pymol API(cmd),在python文件中执行代码: 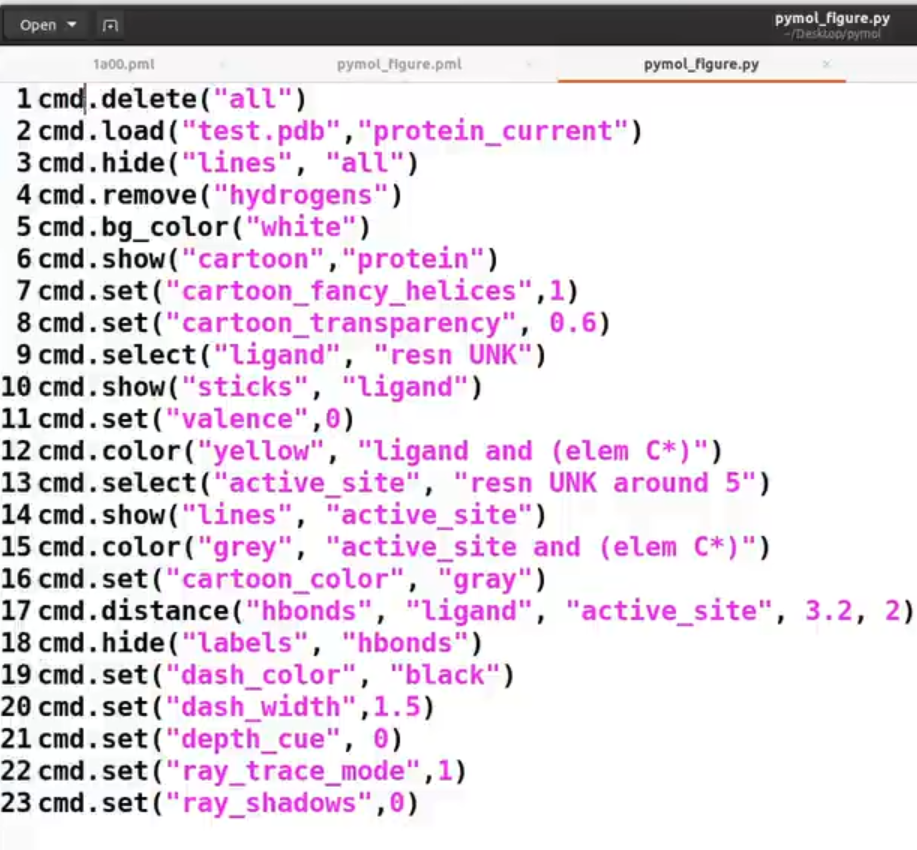 案例展示 案例展示 和.pml脚本的效果是一样的! 所有的pymol指令都可以在python中使用API“cmd”来实现!官网有介绍:PyMOL Command Reference 而甚至有些操作只有用python API 可以代码实现,而在pymol程序的命令行中不能实现,如下: 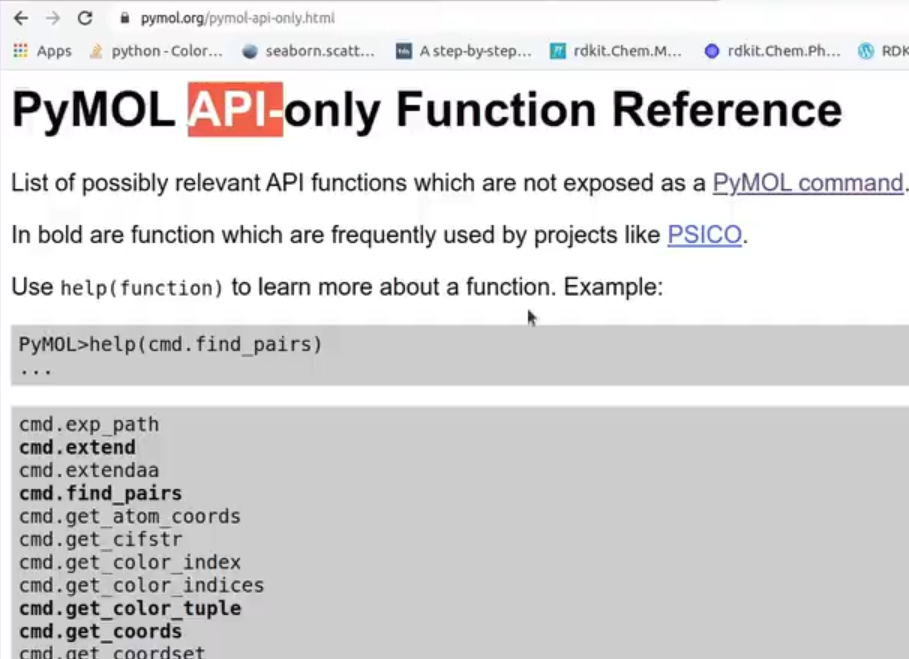 为什么要使用python的API?因为有的大项目中会使用到pymol,例如获取原子坐标。此时直接在python的脚本里使用cmd执行pymol会更方便。 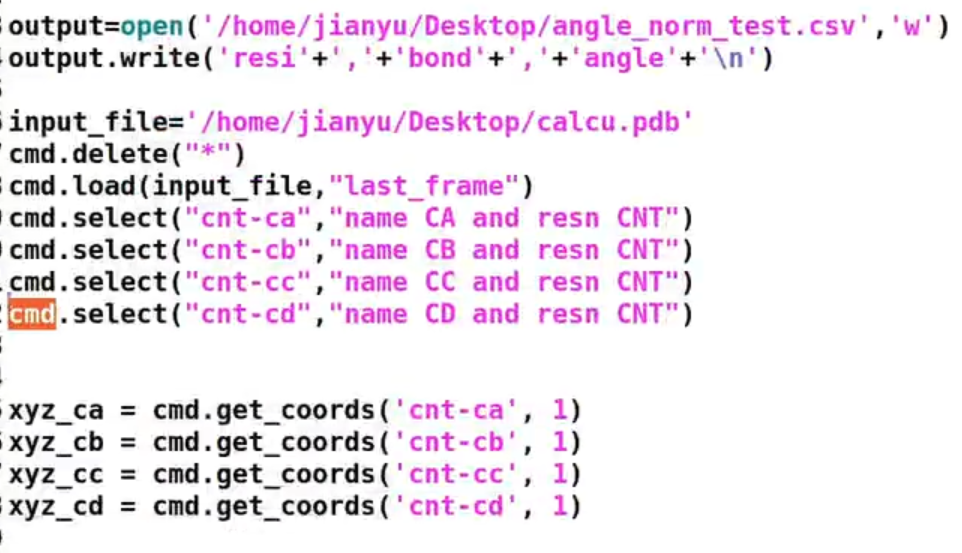 后续会使用numpy等其他的python库函数对原子坐标进行处理,计算二面角之类的 后续会使用numpy等其他的python库函数对原子坐标进行处理,计算二面角之类的
|
【本文地址】
今日新闻 |
推荐新闻 |